Mi SciELO
Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO -
 Accesos
Accesos
Links relacionados
-
 Citado por Google
Citado por Google -
 Similares en
SciELO
Similares en
SciELO -
 Similares en Google
Similares en Google
Compartir
Archivos de Zootecnia
versión On-line ISSN 1885-4494versión impresa ISSN 0004-0592
Arch. zootec. vol.59 no.225 Córdoba mar. 2010
Diversidad genética de Piaractus mesopotamicus utilizado en programas de repoblación
Genetic diversity of Piaractus mesopotamicus stocks used in stocking programs
Lopera Barrero, N.M.1*, Ribeiro, R.P.1, Vargas, L.1, Povh, J.A.2, Lopes, T.S.3, Oliveira, S.N.1 y Gomes, P.C.1
1Universidade Estadual de Maringá. Centro de Ciências Agrárias. Núcleo de Pesquisa PeixeGen. Av. Colombo, 5790. CEP 87020-900. Maringá, PR. Brasil. *nelson.peixegen@gmail.com
2Universidade Federal de Mato Grosso. Instituto de Ciências Agrárias e Tecnológicas. Rodovia Rondonópolis-Guiratinga, km 06. CEP 78735-910. Rondonópolis, MT. Brasil.
3Centro de Aquicultura da UNESP. Via de acesso Prof. Paulo Donato Castellane. CEP 14884-900. Jaboticabal, SP. Brasil.
RESUMEN
Actualmente se ha verificado la disminución y desaparición de muchas especies de peces en varios ambientes acuáticos en el Brasil. Como forma de minimizar ese impacto, programas de repoblación vienen siendo utilizados con más intensidad, entretanto, se le ha dado poca importancia a la variabilidad genética, parámetro fundamental para cualquier práctica de conservación. El objetivo de este estudio fue analizar la diversidad genética de lotes de Piaractus mesopotamicus usados en programas de repoblación, mediante el marcador molecular RAPD. Se analizaron 60 reproductores de dos estaciones de piscicultura ubicadas en las ciudades de Sapopema (S) y Cambará (C) en el Estado del Paraná (Brasil) y 30 larvas de la progenie del lote de Sapopema (PS). Los 10 oligonucleótidos seleccionados produjeron 69 fragmentos de los cuales 62 (89,85%) fueron polimórficos. Fueron observadas diferencias (p<0,05) en la frecuencia de 42 fragmentos, con cuatro exclusivos para el lote de Sapopema. Los resultados de variabilidad genética estimados por el porcentaje de fragmentos polimórficos (S: 70,15%; PS: 73,13% y C: 77,81%) y por el índice de diversidad genética de Shannon (S: 0,365; PS: 0,379 y C: 0,468) mostraron que la variabilidad genética de S fue mantenida en la progenie (PS), debido posiblemente al buen manejo reproductivo.
La variabilidad genética encontrada entre los dos lotes de reproductores indicó una diferenciación genética entre ellos, posiblemente debido al efecto fundador. De acuerdo con la AMOVA, la mayor parte de la variación está dentro de cada lote (62,43%). Este resultado se corroboró con los valores de FST (0,375) y del número efectivo de emigrantes (2,20), que mostraron una alta diferenciación genética y un bajo flujo génico. También se constató que S y SP fueron los más semejantes genéticamente (identidad genética, IG= 0,851) y que S y C presentaron menos genes en común (IG= 0,786).
Palabras clave: Genética de la conservación. Pacu. Pez. Progenie.
SUMMARY
Nowadays it has been verified the decrease and the disappearance of many fish species in several aquatic environments in the Brazil. As form of minimizing that impact, stocking programs come being used with more intensity, meantime, little importance has been given to the genetic variability, fundamental parameter for any conservation practices. The objective of this study was to analyze the genetic diversity of Piaractus mesopotamicus stocks used in stocking programs, through the RAPD molecular marker. Sixty broodstocks of two fish farms stations located in the Sapopema (S) and Cambará (C) cities in the Paraná State (Brazil) and 30 larvae of the Sapopema offspring (PS) were analyzed. The 10 selected primers yielded 69 fragments of which 62 (89.85%) were polymorphic. Differences (p<0.05) in the frequency of 42 fragments were observed, with four exclusive for the Sapopema stock. The genetic variability results estimated by the percentage of polymorphic fragments (S: 70.15%; PS: 73.13% and C: 77.81%) and for the genetic diversity of Shannon index (S: 0.365; PS: 0.379 and C: 0.468) showed that the genetic variability was maintained in the offspring, due possibly to the good reproductive management. On the other hand, the opposing genetic variability among the two broodstocks indicated a genetic differentiation among them, possibly due to the founder effect. According with the AMOVA, most of the variation is within each stock (62.43%). This result was corroborated with the FST (0.375) values and the effective number of migrants (2.20) that showed a high genetic differentiation and a low gene flow. It was also verified that S and SP were those but similar genetically (genetic identity, IG= 0.851) and that S and C presented less genes in common (IG= 0.786).
Key words: Conservation genetics. Pacu. Fish. Offspring.
Introducción
Factores como la contaminación de los ríos (Monteiro et al., 2006), la construcción de empresas hidroeléctricas (Aho et al., 2006), la sobrepesca (Povh, 2007) y el ecoturismo mal planeado (Metzger y Casatti, 2006) asociados a los contundentes cambios climáticos, han contribuido con la desaparición de diversas especies de peces.
Entre esas especies, el pacu, Piaractus mesopotamicus (orden Characiformes, familia Characidae, subfamilia Myleinae) conocido también como caranha o pacu caranha (Nakatani et al., 2001), es una especie nativa migratoria de las cuencas de los ríos Paraná, Paraguay y Uruguay (Urbinati y Gonçalves, 2005) que en los últimos años ha presentado una reducción progresiva de sus poblaciones naturales (figura 1).
Debido a ser una especie de elevado valor comercial y cultural se están llevando a cabo tareas de conservación, y entre las más utilizadas en el Brasil estarían los programas de repoblación (Hilsdorf et al., 2006). Los programas de repoblación destacan por ser estrategias de conservación viables para la mayoría de los piscicultores (Sirol y Britto, 2006), sin embargo, es necesario un apoyo científico que permita la correcta orientación y determinación de los sistemas reproductivos, técnicas y estrategias a ser usadas.
Los análisis genéticos de lotes de pisciculturas permiten obtener información de gran importancia para conseguir resultados significativos para la producción y conservación de peces (Lopera Barrero, 2005), ya que la pérdida de variabilidad genética en lotes de piscifactorías debido al inadecuado manejo reproductivo (Frost et al., 2006) o por deficiencias en el número efectivo de reproductores (Aho et al., 2006) puede producir problemas de endogamia, adaptabilidad y supervivencia de progenies usadas en los programas de repoblación (Povh, 2007). Esos problemas pueden consecuentemente afectar las poblaciones naturales de peces (Sønstebø et al., 2007) y el ecosistema en general, pudiendo conducir la especie a la extinción (Agostinho et al., 2005).
Para analizar la diversidad genética de lotes de piscifactorías, el marcador molecular RAPD (Random Amplified Polymorphic DNA) ha sido utilizado con éxito en investigaciones de varias especies de peces (Ramella et al., 2006; Lopera Barrero et al., 2006; Basavaraju et al., 2007; Gomes, 2007) y de animales terrestres (Egito et al., 2005a) como herramienta en la búsqueda de la conservación genética, del éxito de programas de mejora genética y de la creación de bancos de ADN (Egito et al., 2005b).
Por tanto, el objetivo de este estudio fue analizar la diversidad genética de lotes de P. mesopotamicus usados en programas de repoblación, mediante el marcador molecular RAPD. Estos resultados permitirán orientar objetivamente estos programas realizados en ríos brasileños y así reducir la pérdida de diversidad genética en los lotes de reproductores y en las poblaciones naturales.
Material y métodos
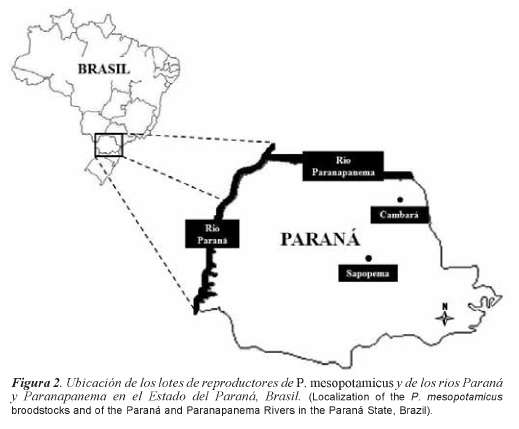
Las muestras de aleta fueron recolectadas de dos lotes de reproductores de P. mesopotamicus (30 individuos de cada lote) en piscifactorías ubicadas en las ciudades de Sapopema (23o54'39''S y 50o34'48''W) y Cambará (20o02'00''S y 50o06'00''W), en el Estado del Paraná (Brasil), los cuales son utilizados en programas de repoblación de los ríos Paraná y Paranapanema (figura 2). Después de la inducción hormonal (extracto de hipófisis de carpa) de los reproductores de Sapopema, utilizando el sistema reproductivo por extrusión descrito por Zaniboni-Filho y Nuñer (2004), fueron recolectadas 30 larvas de tres días de vida.
Para extraer el ADN se utilizó la metodología descrita por Bardakci y Skibinski (1994), modificada por Povh et al. (2005). Fragmentos de aleta caudal de aproximadamente 0,5 cm2 y larvas enteras se colocaron en micro-tubos con 550 μl de tampón de lisis (50 mM Tris-HCl pH 8,0, 50 mM EDTA, 100 mM NaCl, 1 % SDS) y 200 μg/ml de proteinasa K, y se incubaron en baño maría a 50oC durante 12 horas. El ADN se lavó con dos extracciones con fenol y tres de cloroformo, se precipitó con dos veces y media de volumen de alcohol etílico absoluto helado y un décimo de volumen de acetato de sodio en relación al volumen recuperado y se incubó durante dos horas a -20oC. El ADN fue centrifugado, lavado con 2 ml de alcohol etílico 70%, suspendido en 60 μl de tampón TE (10 mM Tris pH 8,0 y 1 mM EDTA) y tratado con 30 μg/ml de RNAasa. Las muestras se incubaron durante 40 minutos en baño maría a 37oC y se conservaron a -20oC.
El ADN se cuantificó en espectrofo-tómetro Shimadzu (UV 1601, EE.UU.), en una amplitud de onda de 260 nm y se diluyó en tampón TE a una concentración de 10 - 5 ng/μl (aletas y larvas, respectivamente). La integridad del ADN se verificó en electro-foresis horizontal usando un gel de agarosa al 1%, a 70 voltios durante 60 minutos, en tampón TBE 1X (500 mM Tris-HC1, 60 mM ácido bórico, 83 mM EDTA). El gel se tiñó con bromuro de etidio (0,5 μg/ml) durante 30 minutos y la imagen se capturó mediante el sistema fotográfico EDAS (Kodak 1D Image Analysis 3.5, EE.UU.).
Las condiciones de amplificación utilizadas fueron las descritas por Williams et al. (1990), con algunas modificaciones. El ADN se amplificó en un volumen de reacción de 25 μl, en el cual se utilizó tampón Tris-KCl 1X (Tris-HCl 20 mM pH 8,4 y KCl 50 mM), 2 mM MgCl2, 100 ng de oligonu-cleótido, 0,2 mM de cada dNTP, una unidad de Taq ADN Polimerasa y 20 ng de ADN molde. Las reacciones de RAPD se amplificaron en un termociclador de PCR Eppendorf Mastercycler® Gradient (EE.UU.), programado para 40 ciclos, con un paso inicial de desnaturalización a 94oC durante cuatro minutos y un paso final de extensión a 72oC durante cinco minutos. Cada ciclo consistió de un minuto a 94oC, 90 segundos a 40oC y dos minutos a 72oC.
Fueron evaluados 60 oligonucleótidos del Kit Operon (Operon Technologies Inc. Alameda, Ca, EEUU). Para evaluar los lotes y la progenie se seleccionaron aquellos oligonucleótidos que resultaron con buenas características reproducibles y de nitidez. Los productos de amplificación se separaron en gel de agarosa al 1,5%. Se utilizaron 20 μl del producto amplificado y 2 μl de tampón de muestra (40% de sacarosa y 0,25% de azul de bromofenol) en electrofo-resis horizontal. La electroforesis se hizo con 70 voltios por cuatro horas usando tampón TBE 1X. Para verificar la existencia de contaminación, se utilizó un control negativo para cada reacción, donde su amplificación se hizo adicionando todos los componentes citados, excepto el ADN. El gel se reveló utilizando un baño en solución de 0,5 μg/ml de bromuro de etidio por 30 minutos. Cada gel se fotografió usando el sistema fotográfico EDAS (Kodak 1D Image Analysis 3.5, EEUU).
El tamaño de los fragmentos se estimó por comparación con el patrón ADN Ladder de 100 pb (15 bandas con tamaño entre 100 y 2072 pb - Invitrogen® EE.UU.). La presencia o ausencia de fragmentos de tamaños moleculares idénticos se usó para la construcción de una matriz de similitud con base en el cálculo del coeficiente de de similitud de Jaccard, codificando 1 como presencia de fragmento y 0 como su ausencia.
El índice de diversidad genética de Shannon (Lewontin, 1972) se estimó mediante la utilización del programa PopGene 1.31 (Yeh et al., 1999). El programa TFPGA 1.3 (Miller, 1997) se utilizó para determinar el porcentaje de fragmentos polimórficos (critério de 95%), distancia e identidade genética (Nei, 1978) entre los lotes y la frecuencia de los fragmentos por el test exacto (Raymond y Rousset, 1995).
El programa Arlequin 3.0 (Excoffier et al., 2005) se utilizó para determinar la diferenciación genética entre los lotes por médio de las estimaciones de FST (Weir y Cockerham, 1984) y para el análisis de variancia molecular (AMOVA) (Excoffier et al., 1992). El nivel de significación de estas estimaciones fue verificado mediante el método de permutaciones aleatorias con 1000 y 10 000 permutaciones. La magnitud de diferenciación genética entre los lotes fue determinada según la definición de Wright (1978), donde valores de FST entre 0,00 a 0,05; 0,05 a 0,15; 0,15 a 0,25 y > 0,25 indican respectivamente pequeña, moderada, alta y elevada diferenciación genética. Este programa también se utilizó para determinar el número efectivo de emigrantes (Nm).
La matriz del coeficiente de similitud de Jaccard y el método de agrupamiento UPGMA se usaron para elaborar un dendrograma, mediante el programa estadístico NTSYS 1.7 (Rohlf, 1989).
Resultados y discusión
La amplificación de las muestras de P. mesopotamicus con los 10 oligonucleótidos seleccionados (OPA01, OPA02, OPA03, OPA06, OPA07, OPW01, OPW02, OPW03, OPW19 y OPX01), utilizando el marcador molecular RAPD, produjo un total de 69 fragmentos (62 polimórficos= 89,85%) con tamaño entre 200 pares de bases (pb) (obtenido con la amplificación del oligonucleótido OPA03) y 1500 pb (obtenido con la amplificación del oligonucleótido OPW19). El número de fragmentos varió entre 4 (OPA02 y OPA07) y 15 (OPW19). Se observaron diferencias (p<0,05) en las frecuencias de 42 de los 69 fragmentos entre los lotes de reproductores y la progenie. Se observaron cuatro fragmentos exclusivos en el lote de Sapopema (OPW01 - 1300 pb, OPW02 - 1400 pb y OPW03 - 600 y 1000 pb) lo que representa una de las características de las diferencias genéticas entre los dos lotes de reproductores (tabla I).
Los valores del porcentaje de fragmentos polimórficos, índice de diversidad genética de Shannon, FST y del número efectivo de emigrantes son presentados en la tabla II. El porcentaje de fragmentos polimórficos (%FP) y el índice de diversidad genética de Shannon (IS) para el lote de reproductores de Sapopema y su progenie mostró que la variabilidad genética fue preservada en la primera generación, debido posiblemente al adecuado manejo reproductivo del plantel. Según Moreira et al. (2007), una pérdida de variabilidad genética en la piscicultura siempre es esperada cuando existe un mal manejo reproductivo debido al cruzamiento de individuos emparentados, lo que consecuentemente aumenta el coeficiente de en-dogamia (Kang et al., 2006) y reducirá el número efectivo de reproductores (Frost et al., 2006). Esta situación es bastante común en piscifactorías, ya que el método más ampliamente usado en la formación de nuevos lotes es la selección de individuos con características favorables, lo que puede dar a lugar a un cuello de botella (bottleneck effect) si la selección implica la elección de un número reducido de reproductores estrechamente emparentados, ocasionando pérdida de variabilidad genética en sus progenies (Aho et al., 2006).
Los resultados del presente estudio demostraron que este efecto no afectó al lote de Sapopema, en el que se encontraron altos valores de variabilidad genética entre sus reproductores (%FP= 70,15%; IS= 0,365), demostrando un buen manejo reproductivo. Este resultado es evidenciado al preservarse la variabilidad genética en su primera generación (%FP= 73,13%; IS= 0,379;) como consecuencia de la disminución de la correlación negativa entre la similitud genética y el número de generaciones (Freitas y Galetti Jr, 2005).
Algunos autores estiman que la disminución de la variabilidad genética en piscifactorías es normalmente irreversible (Wasko et al., 2004; Sekino et al., 2004). Sin embargo, con un adecuado manejo reproductivo que implique la formación de lotes con suficiente variabilidad genética (efecto fundador), la introducción de reproductores genéticamente divergentes y de diferentes orígenes (Freitas y Galetti Jr, 2005) y la utilización de sistemas reproductivos y de cruzamientos eficientes (Povh, 2007) es posible mantener el pool genético de poblaciones mantenidas en ambientes controlados y utilizadas con fines de repoblación, como es el caso de los lotes de P. mesopota-micus analizados en este estudio.
Al analizar la variabilidad genética de los lotes de reproductores (Sapopema y Cambará) se encontró una diferenciación genética entre ellos posiblemente debida al efecto fundador y al aislamiento genético de los reproductores (tabla II). De acuerdo con los resultados del AMOVA, la mayor parte de esa variación está dentro de cada lote (62,43%) y no entre los lotes (37,57%) de P. mesopotamicus (tabla III). Estos resultados fueron confirmados con los valores de FST, que mostraron una alta diferenciación genética (según definición de Wright, 1978), haciendo presumir que en el momento de la formación de los lotes estos tenían un origen diferente (pertenecían a poblaciones naturales diferentes), a pesar de ser ambos procedentes del río Paraná. El número efectivo de emigrantes (Nm) mostró igualmente un bajo flujo génico. Esta situación es más clara en el dendrograma de similitud genética, donde no hubo agrupamiento de los individuos de los dos lotes y por el contrario fue observada la formación de dos grupos diferentes (figura 3).
Los valores estimados de identidad genética (IG) y de distancia genética (DG) son mostrados en la tabla IV. Esos valores indicaron que el lote de Sapopema y su progenie fueron los más semejantes genéticamente (IG= 0,851) y que los lotes de reproductores fueron los más divergentes (IG= 0,786).
Estos resultados son de gran importancia, ya que a partir de ellos, el manejo reproductivo de esos lotes utilizados en programas de repoblación pueden ser orientados correctamente y con mayor objetividad, en el sentido de evitar el cruzamiento de individuos emparentados que pueda ocasionar una reducción de la variabilidad genética. Las especies de importancia comercial y especialmente aquellas amenazadas de extinción como es el caso del P. mesopota-micus, requieren un constante análisis genético de sus lotes mantenidos en las piscifactorías y de sus poblaciones naturales. El análisis genético de lotes de piscifactorías proporciona información de gran importancia para conseguir ganancias en la producción y en la conservación de peces (Povh, 2007).
A partir de estas evidencias y de los resultados de variabilidad, distancia e identidad genética, podemos sugerir que el manejo reproductivo, genético y de mejora de los lotes de P. mesopotamicus de la región de Sapopema y Cambará deben ser realizados de forma individual, como lotes genéticamente diferentes, especialmente cuando el objetivo sean programas de conservación de poblaciones naturales de esta especie mediante la repoblación. Así mismo, para mantener la variabilidad genética de esos lotes, es necesaria la introducción de nuevo material genético (reproductores), los cuales pueden ser colectados a partir de poblaciones naturales genéticamente diferentes de los ríos Paraná o Paranapanema o también con el intercambio de reproductores entre estas piscifactorías, ya que se mostraron divergentes, pudiendo contribuir con un pool genético que permita preservar la variabilidad genética en ambos lotes. Esta introducción e intercambio de individuos debe ser realizada con mucha cautela y basada primordialmente en análisis genéticos, ya que cuando dos poblaciones están aisladas por mucho tiempo sus diferencias genéticas pueden ser significativas, a punto de la formación de híbridos entre sus individuos llegar a ser genéticamente perjudicial (depresión exogámica).
De esta forma, la correcta elección de los reproductores que serán usados en la formación del lote de una piscifactoría y el análisis y monitoreo genético de estos pueden ofrecer bases importantes para formular estrategias de manejo reproductivo (Sønstebø et al., 2007). Estas estrategias permitirán un intercambio e introducción de reproductores y de esa forma fragmentar ciclos de endogamia que son comunes en ambientes controlados (Moreira et al., 2007) y que en ciertos casos, pueden definir la conservación de una especie y su futuro potencial biológico (Melo et al., 2006).
Otros autores han llevado a cabo estudios con esta especie destinados a programas de repoblación. Lopera Barrero et al. (2007) analizando un lote de reproductores y su progenie encontraron que hubo una gran reducción de la variabilidad genética en la progenie (IS= 0,439 y 0,224, respectivamente), debido posiblemente a la razón sexual utilizada durante el cruzamiento. Povh (2007) comparando un lote de reproductores y una población natural de Piaractus mesopotamicus, encontró una mayor variabilidad genética en la población (IS= 0,289 y 0,345, respectivamente), debido posiblemente a un deficiente manejo reproductivo en la piscifactoría. En este estudio, la variabilidad genética del lote de Sapopema fue preservada en la progenie, demostrando cómo con la utilización de una correcta orientación genética es posible mantener un manejo eficiente de los reproductores, evitando la disminución de la variabilidad genética.
Otra característica que debemos abordar es el efecto que puede ocasionar la liberación de progenies en los ríos sobre las poblaciones naturales de peces. La repoblación de los ríos es una estrategia de conservación de la biodiversidad acuática que consiste en el restablecimiento de poblaciones naturales a partir de la liberación de alevines obtenidos con el auxilio de estaciones de piscicultura. Su finalidad es la protección, la preservación del flujo génico y la perpetuación y establecimiento reproductivo de las poblaciones naturales de peces. A pesar del objetivo claro y de las buenas intenciones de este tipo de programa, puede representar riesgos genéticos y reproductivos a las poblaciones naturales.
Según Sønstebø et al. (2007), el cruzamiento de individuos genéticamente distintos a aquellos encontrados en una población natural puede promover la pérdida de adaptabilidad al ambiente, que puede influir sobre la supervivencia de progenies en el ambiente natural (Melo et al., 2006). Por esta razón, la primera actuación a realizar en la implantación de programas de repoblación, como es el caso para los lotes de P. mesopotamicus analizados en este estudio, es verificar la diversidad genética de los lotes, de las progenies y de las poblaciones naturales en todos los periodos del año, evitando la pérdida de variabilidad genética y de adaptabilidad en las poblaciones naturales de peces y al mismo tiempo verificando la eficacia de este tipo de programa de conservación.
Los resultados obtenidos en el presente estudio demostraron que el marcador molecular RAPD puede ser usado con éxito para estimar el nivel de variabilidad genética de lotes de reproductores y progenies, informaciones importantes para la correcta y objetiva orientación y monitoreo de programas de piscicultura y de repoblación de peces. Se debe también considerar la necesidad de mayores investigaciones genéticas y más asistencia a los productores, para orientarlos correctamente en los aspectos relacionados al manejo genético y reproductivo, la forma de obtener un mayor retorno económico y una conservación del ecosistema.
Conclusión
La variabilidad genética en la progenie del lote de Sapopema fue preservada debido al adecuado manejo reproductivo. Se encontró diferenciación genética entre los lotes de reproductores de Sapopema y Cambará. Estos resultados pueden contribuir a un manejo más adecuado de los lotes de P. mesopotamicus de cada piscifactoría y la orientación objetiva de programas de repoblación utilizados en los ríos Paraná y Paranapanema.
Bibliografía
Agostinho, A.A., Thomaz, S.M. and Gomes, L.C. 2005. Conservation of the biodiversity of Brazil's inland waters. Conserv. Biol., 19: 646-652. [ Links ]
Aho, T., Rönn, J., Piironen, J. and Björklund, M. 2006. Impacts of effective population size on genetic diversity in hatchery reared Brown trout (Salmo trutta L.) populations. Aquaculture, 253: 244-248. [ Links ]
Bardakci, F. and Skibinski, D.O.F. 1994. Application of the RAPD technique in tilapia fish: species and subspecies identification. Heredity, 73: 117-123. [ Links ]
Basavaraju, Y., Prasad, D.T., Rani, K., Kumar, S.P., Naika, U.D., Jahageerdar, S., Srivastava, P.P., Penman, D.J. and Mair, G.C. 2007. Genetic diversity in common carp stocks assayed by random-amplified polymorphic DNA markers. Aquac. Res., 38: 147-155. [ Links ]
Egito, A.A., Fuck, B., Spritze, A.L., Oliveira, R.R., McManus, C., Mariante, A.S., Ribeiro, M.N., Albuquerque, M.S.M., Paiva, S.R., Castro, S.T.R. and Santos, S.A. 2005a. RAPD markers utilization on the formation or maintenance of conservation nuclei of livestock species. Arch. Zootec., 54: 277-281. [ Links ]
Egito, A.A., Albuquerque, M.S.M., Castro, S.T.R., Paiva, S.R., Marques, J.R.F., McManus, C., Mariante, A.S., Abreu, U.P.G., Santos, S.A., Sereno, J.R., Fioravanti, M.C.S., Vaz, C.M., Nobre, F.V., Oliveira, J.V., Carvalho, J.H. de, Costa, M.R., Ribeiro, M.N. e Lara, M.A. 2005b. Situação atual do banco de DNA de recursos genéticos animais no brasil. Arch. Zootec., 54: 283-288. [ Links ]
Excoffier, L., Laval, G. and Schneider, S. 2005. Arlequin Ver. 3.0: an integrated software package for population genetics data analysis. Evol. Bioinf. Online, 1: 47-50. [ Links ]
Excoffier, L., Smouse, P.E. and Quattro, J.M. 1992. Analysis of molecular variance inferred from metric distances among DNA haplotypes: application to human mitochondrial DNA restriction data. Genetics, 131: 479-491. [ Links ]
Freitas, P.D. and Galetti Jr., P.M. 2005. Assessment of the genetic diversity in five generations of commercial broodstock line of Litopenaeus vannamei shrimp. Afr. J. Biotechnol., 4: 1362-1367. [ Links ]
Frost, L.A., Evans, B.S. and Jerry, D.R. 2006. Loss of genetic diversity due to hatchery culture practices in barramundi (Lates calcarifer). Aquaculture, 261: 1056-1064. [ Links ]
Gomes, P.C. 2007. Diversidade genética de três populações de piapara (Leporinus elongatus), utilizando marcadores moleculares. Dissertação (Mestrado em Zootecnia). Programa de Pós-Graduação em Zootecnia. Universidade Estadual de Maringá. Maringá. 75 pp. [ Links ]
Hilsdorf, A.W.S., Resende, E.K. e Marques, D.K.S. 2006. Genética e conservação de estoques pesqueiros de águas continentais no Brasil: Situação atual e perspectivas. Embrapa Pantanal. Corumbá. 44 pp. [ Links ]
Kang, J.H., Noh, J.K., Kim, J.H., Lee, J.H., Kim, H.C., Kim, K.K., Kim, B.S. and Lee, W.J. 2006. Genetic relationship between broodstocks of olive flounder, Paralichthys olivaceus (Temminck and Schlegel) using microsatellite markers. Aquac. Res., 37: 701-707. [ Links ]
Lewontin, R.C. 1972. The apportionment of human diversity. Evol. Biol., 6: 381-398. [ Links ]
Lopera Barrero, N.M. 2005. Diversidade genética de populações de piracanjuba (Brycon orbignyanus) com a técnica de RAPD. Dissertação (Mestrado em Zootecnia). Programa de Pós-Graduação em Zootecnia. Univer-sidade Estadual de Maringá. Maringá. 45 pp. [ Links ]
Lopera Barrero, N.M., Streit Jr., D.P., Sirol, R.N., Ribeiro, R.P., Povh, J.A., Vargas, L., Gomes, P.C., Lopes, T. da S., Jacometo, C.B. e Blanck, D.V. 2007. Monitoramento da diversidade genética dos reproductores e da progênie de Piaractus mesopotamicus obtida pelo sistema seminatural. Em: Congresso Brasileiro de produção de peixes nativos de água doce, 1. Dourados. Anais. Embrapa Agropecuária Oeste. Dourados. Documentos 87. [ Links ]
Lopera Barrero, N.M., Ribeiro, R.P., Sirol, R.N., Povh, J.A., Gomes, P.C., Vargas, L. and Streit Jr., D.P. 2006. Genetic diversity in piracanjuba populations (Brycon orbignyanus) with the RAPD (Random Amplified Polimorphic DNA) markers. J. Anim. Sci., 84: 170-170. [ Links ]
Melo, D.C., Oliveira, D.A.A., Ribeiro, L.P., Teixeira, C.S., Souza, A.B., Coelho, E.G.A., Crepaldi, D.V. e Teixeira, E.A. 2006. Caracterização genética de seis plantéis comerciais de tilápia (Oreochromis) utilizando marcadores microssatélites. Arq. Bras. Med. Vet. Zootec., 58: 87-93. [ Links ]
Metzger, J.P. e Casatti, L. 2006. Do diagnóstico à conservação da biodiversidade: o estado da arte do programa BIOTA/FAPESP. Biota Neotrop., 6: 1-23. [ Links ]
Miller, M.P. 1997. Tools of population genetic analysis (TFPGA) 1.3: a Windows program for the analysis of allozyme and molecular population genetic data. Utah State University, 1997. Disponible en: <http://www.marksgeneticsoftware.net/tfpga.htm>. Acceso en abr. 2008. [ Links ]
Monteiro, D.A., Almeida, J.A., Rantin, F.T. and Kalinin, A.L. 2006. Oxidative stress biomarkers in the freshwater characid fish, Brycon cephalus, exposed to organophosphorus insecticide Folisuper 600 (methyl parathion). Comp. Biochem. Physiol., 143: 141-149. [ Links ]
Moreira, A.A., Hilsdorf, A.W.S., Silva, J.V. e Souza, V.R. 2007. Variabilidade genética de duas variedades de tilápia nilótica por meio de marcadores microssatélites. Pesqui. Agropecu. Bras., 42: 521-526. [ Links ]
Nakatani, K., Agostinho, A.A., Baumgartner, G., Bialetzki, A., Sanches, P.V., Makrakis, M.C. e Pavanelli, C.S. 2001. Ovos e larvas de peixes de água doce. EDUEM, Maringá. 378 pp. [ Links ]
Nei, M. 1978. Estimation of average heterozygosity and genetic distance from a small number of individual. Genetics, 89: 583-590. [ Links ]
Povh, J.A. 2007. Avaliação da diversidade genética e do manejo reprodutivo do pacu, Piaractus mesopotamicus. Tese (Doutorado em Zootecnia). Programa de Pós-Graduação em Zootecnia. Universidade Estadual de Maringá. Maringá. Brasil. 75 pp. [ Links ]
Povh, J.A., Moreira, H.L.M., Ribeiro, R.P., Prioli, A.P., Vargas, L., Blanck, D.V., Gasparino, E. e Streit Jr., D.P. 2005. Estimativa da variabilidade genética em tilápia do Nilo (Oreochromis niloticus) com a técnica de RAPD. Acta Sci. An. Sci., 27: 1-10. [ Links ]
Ramella, M.S., Kroth, M.A., Meurer, S., Nuñer, A.P. de O., Zaniboni-Filho, E. and Arisi, A.C.M. 2006. Genetic variability in four fish species (Pimelodus maculatus, Prochilodus lineatus, Salminus brasiliensis and Steindachneridion scripta) from Uruguay River Basin. Braz. Arch. Biol. Techn., 49: 589-598. [ Links ]
Raymond, M. and Rousset, F. 1995. An exact test for population differentiation. Evolution, 49: 1280-1283. [ Links ]
Rohlf, F.J. 1989. NTSYS-Pc: Numerical taxonomy and multivariate analysis system. Exeter Publishers. New York. 191 pp. [ Links ]
Sekino, M., Sugaya, T., Hara, M. and Taniguchi, N. 2004. Relatedness inferred from microsatellite genotypes as a tool for broodstock management of Japanese flounder Paralichthys olivaceus. Aquaculture, 233: 163-172. [ Links ]
Sirol, R.N. e Britto, S.G. 2006. Conservação e manejo da ictiofauna: repovoamento. Em: M.G. Nogueira, R. Henry e A. Jorcin. (Eds.). Ecologia de reservatórios: impactos potenciais, ações de manejo e sistemas em cascatas. RiMA. São Carlos. pp. 275-284. [ Links ]
Sønstebø, J.H., Borgstrøm, R. and Heun, M. 2007. Genetic structure of brown trout (Salmo trutta L.) from the Hardangervidda mountain plateau (Norway) analyzed by microsatellite DNA: a basis for conservation guidelines. Conserv. Genet., 8: 33-44. [ Links ]
Urbinati, E.C. e Gonçalves, F.D. 2005. Pacu (Piaractus mesopotamicus). Em: B. Baldisse-rotto e L.C. Gomes. (Eds.). Espécies nativas para piscicultura no Brasil. UFMS. Santa Maria. pp. 225-246. [ Links ]
Wasko, A.P., Martins, C., Oliveira, C., Senhorini, J.A. and Foresti, F. 2004. Genetic monitoring of the Amazonian fish matrinchã (Brycon cephalus) using RAPD markers: insights into supportive breeding and conservation programmers. J. Appl. Ichthyol., 20: 48-52. [ Links ]
Weir, B.S. and Cockerham, C.C. 1984. Estimating F statistics for the analysis of population structure. Evolution, 38: 1358-1370. [ Links ]
Williams, J.G.K., Rafalski, J.A., Kubelik, A.R., Livak, K.J. and Tingey, S.V. 1990. DNA polymorphism amplified by arbitrary primers are useful as genetic markers. Nucleic Acids Res., 18: 6531-6535. [ Links ]
Wright, S. 1978. Evolution and genetics of population. University of Chicago Press. Chicago. 580 pp. [ Links ]
Yeh, F.C., Boyle, T.Y.Z. and Xiyan, J.M. 1999. PopGene Version 131: Microsoft Window-based freeware for population genetic analysis. University of Alberta and Center for International Forestry Research. Alberta. 29 pp. [ Links ]
Zaniboni-Filho, E. e Nuñer, A.P.O. 2004. Fisiologia da reprodução e propagação artificial dos peixes. Em: J.E.P. Cyrino, E.C. Urbinati, D.M. Fracalossi e N. Castagnolli. (Eds.). Tópicos especiais em piscicultura de água doce tropical intensiva. TecArt. São Paulo. pp. 45-73. [ Links ]
Recibido: 29-9-07.
Aceptado: 14-5-08.