Mi SciELO
Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO -
 Accesos
Accesos
Links relacionados
-
 Citado por Google
Citado por Google -
 Similares en
SciELO
Similares en
SciELO -
 Similares en Google
Similares en Google
Compartir
Archivos de Zootecnia
versión On-line ISSN 1885-4494versión impresa ISSN 0004-0592
Arch. zootec. vol.62 no.240 Córdoba dic. 2013
https://dx.doi.org/10.4321/S0004-05922013000400008
Avaliação genética de características morfométricas em tilápias do Nilo cultivadas
Genetic evaluation for body traits in farmed Nile tilapias
Kunita, N.M.1; Oliveira, C.A.L.1*; Oliveira, S.N.1; Yoshida, G.M.1; Rizzato, G.S.1; Resende, E.K.2 e Ribeiro, R.P.1
1Universidade Estadual de Maringá. Centro de Ciências Agrárias. Departamento de Zootecnia. Maringá-PR. Brasil.*caloliveira@uem.br
2Empresa Brasileira de Pesquisa Agropecuária-EMBRAPA. Centro de Pesquisa Agropecuária do Pantanal. Mato Grosso do Sul. Brasil
RESUMO
No presente estudo estimou-se valores genéticos, as diferenças existentes nas respostas à seleção, os componentes de (co)variância e parâmetros genéticas de características morfométricas de tilápias do Nilo (O. niloticus), cultivados em sistema de tanques rede. Foram analisadas características morfométricas e de desempenho de 1824 animais. As estimativas de herdabilidades oscilaram entre 0,08541 a 0,194 e a participação relativa dos ambientes comuns de larvicultura e alevinagem variaram entre 0,07402 a 0,1586 e 0,00087 a 0,0795 respectivamente. Os valores de correlações genéticas e de ranking apontaram existência de forte associação genética entre as características avaliadas. Os maiores ganhos genéticos foram para peso e ganho em peso diário, com a melhor resposta a seleção indireta foi verificada para peso. A seleção para velocidade de crescimento promoveram ganho genético em características morfométricas.
Palavras chave: Herdabilidade. Linhagem GIFT. Melhoramento genético de peixes. Oreochromis niloticus.
SUMMARY
Breeding values, the differences in responses to selection, the (co)variance components and genetic parameters of the morphometrics traits of Nile tilapia (O. niloticus) reared in cages were estimated. Morphometric and performance traits of 1824 animals were analyzed. The estimated heritability ranged from 0,08541 to 0,194 and the proportion of the variation due common environmental/hatchery and nursery effects ranged from 0,07402 to 0,1586 and 0,00087 to 0,079, respectively. The values of genetic correlations and ranking indicated a strong association between genetic traits. The largest genetic gains were for weight and daily weight gain, and the best response to indirect selection was checked for weight. Selection for growth rate led to genetic gain in morphometric traits.
Keywords: Heritability. Genetic improvement of fishes. GIFT strain. Oreochromis niloticus.
Introdução
Segundo dados da FAO (2012), a aquicultura mundial está em franca expansão, a produção mundial de pescados foi cerca de 148 milhões de toneladas em 2010. A produção brasileira de pescados no mesmo período foi superior a 1,2 milhões de toneladas, equivalendo menos de 1 % da produção de pescados. Estes percentuais são mantidos quando se contrasta os resultados da aquicultura. Em se tratando da piscicultura de água doce no Brasil, a produção equivale a 1,2 % da produção mundial. Na piscicultura de água doce do Brasil, a tilápia do Nilo é a espécie de maior importância, com produção superior a 155 mil toneladas no ano de 2010, o que representa 40 % da produção brasileira. Este percentual é cerca de cinco vezes maior que a participação da tilapicultura na produção mundial. A produção de tilápias no Brasil representa 6 % da produção mundial (FAO, 2012).
Em função disso, esforços têm sido realizados para aumentar qualidade genética das tilápias produzidas no Brasil. Até muito recentemente, não havia no Brasil nenhum programa de melhoramento genético de peixes, que utilizasse métodos quantitativos consolidados, com controle individual de pedigree (Santos, 2009; Resende et al., 2010; Oliveira et al., 2012). A inexistência deste tipo de ação caracteriza um sistema de produção de peixes em que os animais cultivados apresentam potencial produtivo menor ou igual aos animais disponíveis no ambiente natural (Ponzoni, 2006).
Com a importação de animais da linhagem GIFT em 2005, iniciou-se um programa de melhoramento genético de tilápias, cujo objetivo é realizar a avaliação e seleção desta variedade em condições brasileiras de cultivo. Este grupo genético foi produzido por meio de um programa de melhoramento genético conduzido a partir de 1988 na Malásia, sendo distribuída para vários locais de cultivo no mundo (Ribeiro e Legat, 2008).
Considerando o estágio inicial do programa de melhoramento genético de tilápias no Brasil e o número reduzido de informações a respeito de parâmetros genéticos de peixes tropicais em condições de cultivo do Brasil e América Latina, com a realização deste trabalho objetivou-se estimar componentes de (co)variância e parâmetros genéticos, bem como, estimar as diferenças nas respostas à seleção ao utilizar como critério de seleção o ganho em peso diário, ou características morfométricas como comprimento total, padrão e de cabeça, altura e peso vivo de tilápias do Nilo (Oreochromis niloticus) da variedade GIFT cultivadas em sistema de tanque rede.
Material e métodos
O conjunto de dados utilizado nas análises foi composto por informações de 58 grupos de irmãos completos e meio irmãos, da terceira geração do programa de melhoramento genético da tilápia GIFT no Brasil. A população base que deu origem a esses animais era composta por aproximadamente 20 animais de 30 grupos de irmãos completos de tilápia nilótica da linhagem GIFT (Genetic Improvement of Farmed Tilapias) do WorldFish Center (Malásia), importada para o Brasil no ano de 2005 conforme descrito por Ribeiro e Legat (2008) e Santos (2009).
Os animais avaliados foram produzidos na estação de reprodução de novembro de 2008 e fevereiro de 2009, a partir do acasalamento hierárquico de 120 fêmeas e 60 machos. Os reprodutores foram alojados em hapas individuais de um metro cúbico, na Estação de Piscicultura CODAPAR-UEM, localizada no distrito de Floriano no município de Maringá no estado do Paraná (23o 31'S e 52o 2' W). Todos os hapas eram vistoriados em dias alternados, em busca de fêmeas com características reprodutivas aparentes (aptas à desova); após esta verificação os machos eram colocados nos hapas das fêmeas, e quando verificada a desova os machos eram recolocadas em seus hapas.
A estação de reprodução foi realizada de novembro de 2008 a fevereiro de 2009, a primeira desova foi observada no dia 28 de novembro de 2008, enquanto no dia 14 de fevereiro de 2009 foram observadas quatro desovas. Durante este período foram produzidas as 58 famílias utilizadas neste trabalho, o número de filhos avaliados por família variou de 8 a 71 indivíduos, com média de 30,2 animais. As diferenças nas datas de eclosão causaram variação da idade e, consequentemente, nas médidas das características utilizadas (tabela I).
Após a eclosão, as larvas foram mantidas com as mães, nas unidades de reprodução, até o final da estação reprodutiva, o que gerou uma fonte de variação comum, denominada de ambiente comum de larvicultura, que continha os efeitos do número de indivíduos na família, o tempo de permanência com a mãe e cuidado parental.
Ao término da estação reprodutiva (14 de fevereiro de 2009) foram transferidos 50 representantes de cada família para hapas de alevinagem, distribuídas em um viveiro de terra. A alocação dos alevinos nos hapas considerou a existência de efeitos da posição no viveiro sobre a qualidade de água e disponibilidade de oxigênio. Os alevinos da mesma família foram divididos em dois grupos e distribuídos ao acaso em diferentes locais no viveiro. Esta ação tem objetivo de diminuir o efeito de posição no tanque sobre o desempenho das famílias, porém a manutenção de alevinos da mesma família no mesmo hapa gerou um efeito comum denominado de efeito comum de alevinagem.
Ao atingirem peso superior a 12 g os animais foram identificados por meio de microchips PITT (Passive Integrated Transponder Tags), implantados na cavidade visceral, nesta data (19 de abril de 2009) foi realizada a primeira biometria. Após a identificação, os animais foram transferidos para sistema de cultivo em tanques rede, localizados no Rio do Corvo, município de Diamante do Norte -PR ( 22o36' S e 52o50' W), foram utilizados dois tanquesrede com 6m3, com densidade de estocagem de 100 peixes/m3. Em cada tanque foram distribuídos aleatoriamente representantes de cada família. O período de cultivo foi de 170 dias, compreendido entre os meses de abril a setembro de 2009. Os animais receberam ração com 32 % de proteína bruta, distribuídas em três vezes por dia, as oito, treze e dezessete horas. O ajuste do fornecimento de ração foi de acordo com biometrias realizadas no decorrer do cultivo.
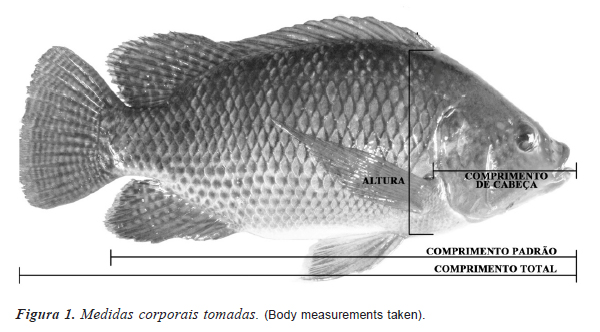
As características morfométricas mensuradas foram comprimento total, padrão e de cabeça; altura e peso vivo do peixe (peso) (figura 1). Além destas, foi calculado o ganho em peso diário (GPD) dado pela razão entre peso vivo e idade do peixe ao final do cultivo. Para as medições de peso foi utilizada balança digital com precisão de 0,1 g. Os comprimentos total e padrão foram medidos utilizando um ictiômetro graduado em centímetros e as demais medidas foram realizadas com auxílio de paquímetro graduado em milímetros (mm). As medidas foram tomadas ao final de aproximadamente 170 dias de cultivo.
No momento das medições foram anotadas as informações de sexo, idade à pesagem, além das informações de identificação individual.
Para estimação dos componentes de variância e parâmetros genéticos foram utilizadas modelos animal unicaracter e bicaracter, conforme descrito abaixo:

Admitindo-se que a, m, c e e possuem distribuição conjunta normal multivariada, tem-se:

Para as análises bicaracter tem-se:

Para as análises bicaracter, se combinou o ganho em peso diário com as demais características e estas características entre si, permitindo estimar a estrutura de associação genética entre as características.
Os componentes de (co)variância, parâmetros genéticos foram estimados utilizando-se métodos bayesianos implementados no sistema computacional MTGSAM - Multiple Trait Gibbs Sampler for Animal Models (Van Tassel e Van Vleck, 1995). Consideraram-se os efeitos genéticos aditivos, de ambientes comuns de larvicultura, de alevinagem e residual, como tendo distribuição a priori normal para as análises unicaracter e bicaracter. Para os componentes de (co)variância, considerou-se a distribuição a priori qui-quadrado invertida e wishart invertida para as análises unicaracter e bicaracter, respectivamente.
As distribuições posteriores foram obtidas a partir de 500000 ciclos, retirandose amostras a cada 10 ciclos, após a eliminação dos 50 000 ciclos iniciais, totalizando 45000 amostras dos componentes de (co)variância. A partir destas amostras foram estimados a média a posteriori e os intervalos de credibilidade de 95 % para os componentes de (co)variância e parâmetros genéticos.
O monitoramento da convergência das cadeias geradas pelo amostrador de Gibbs foi realizado por meio do teste de Heidelberger e Welch (1983), disponível na biblioteca CODA (Convergence Diagnosis and Output Analysis), implementada no programa R (version 2.8.1).
A partir dos componentes de (co)variância foram preditos os valores genéticos dos animais utilizando as equações de modelos mistos de Henderson, implementado no sistema computacional MTSGAM.
Foram estimadas as correlações de Pearson e Spearman para os valores genéticos preditos e classificação dos animais, estas estimativas foram obtidas a partir dos resultados das análises unicaracter.
No processo de seleção utilizou-se os valores genéticos preditos dos animais para cada característica nas análises unicaracter. Realizou-se a seleção dois machos e quatro fêmeas de cada família. Com os valores genéticos dos animais selecionados foi estimada a resposta a seleção direta padronizada para o desvio padrão genético aditivo, utilizando a seguinte fórmula:

Para o cálculo da eficiência relativa da seleção indireta se utilizou a seguinte fórmula:
R(YX)p/RXp
Resultados e discussão
PARÂMETROS GENÉTICOS
Houve indicação de convergência para todas as cadeias de Gibbs dos componentes de (co)variância e parâmetros genéticos de todas as caracteristicas, tanto para a análise unicaracter quanto para bicaracter.
As medidas de associação genética entre as características foram positivas e altas, sugerindo que estas podem ser influenciadas pelo mesmo conjunto de genes (tabela II).
As características peso e ganho em peso diário apresentaram-se mais fortemente associadas que as demais. Os valores das correlações genética e fenotípica para as características foram superiores a 0,9. O mesmo ocorreu para comprimento total e padrão (tabela II).
As correlações genéticas estimadas entre o peso e os comprimentos total e padrão, apontam que a seleção para peso pode resultar em aumento do comprimento e o ganho em peso diário dos animais. A característica ganho em peso diário (GPD) apresentou-se fortemente associado com peso e altura (tabela II).
Estes resultados apontam que o uso de GPD como critério de seleção, além de impactar o peso adulto dos animais, pode resultar em incremento da altura do peixe de forma mais acentuada que as demais características (tabela VII). Este processo de seleção pode promover diferenciação na forma de corpo dos animais em relação àqueles selecionados por outros programas de melhoramento, em função desses programas utilizarem o peso como critério de seleção.
As estimativas das correlações genéticas entre as características obtidas no presente estudo foram inferiores àquelas encontrados por Nguyen et al. (2007) que apresentou valores superiores a 0,96 na associação com as características peso, comprimento e altura. Para as correlações fenotípicas, os valores encontrados nesse trabalho foram semelhantes, principalmente para a associação entre peso que em ambos os trabalhos foram de 0,87.
Ao observar os padrões de associação existente entre as características, verificouse concordâncias entre os resultados deste trabalho de Nguyen et al. (2007), que retrataram que a característica peso apresentou-se mais fortemente associado com os comprimentos total e padrão, que com a altura do peixe (tabela II). De forma semelhante, a característica altura apresentou-se mais fortemente associada com peso que com os comprimentos.
As correlações genéticas estimadas neste trabalho indicam que grande parte dos genes de ação aditiva que influenciam qualquer uma das características avaliadas também influencia as demais, sugerindo que a seleção para qualquer uma dessas características deve resultar em progresso genético nas outras.
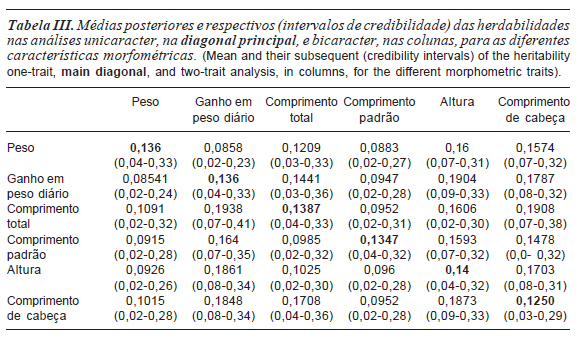
As herdabilidades estimadas, nas análises unicaracter, para todas as características apresentaram valores próximos a 0,14, com exceção da característica comprimento de cabeça que apresentou valor menor. Estas estimativas indicam que o grau de correspondência entre o valor fenotípico e o valor genético foi baixo para as características estudadas (tabela III).
A utilização de informações de apenas um ciclo de produção e de uma estrutura de parentesco contendo apenas informações entre pais e filhos e entre irmãos, pode ter reduzido a capacidade de explicação da variância total pelo componente genético aditivo, resultando em estimativas de herdabilidades de baixa magnitude para as características.
Os intervalos de credibilidade apresentaram limites inferiores, próximos de 0,04 e superiores próximos de 0,30. De acordo com estes resultados, as herdabilidades estimadas para as características peso, ganho em peso diário, comprimento total e padrão, altura e comprimento de cabeça tem probabilidade de assumir valores de baixa e ou média magnitude.
De maneira geral, observou-se grande oscilação nos valores das médias posteriores das herdabilidades das características nas análises bicaracter, havendo superestimação em relação à análise unicaracter como observado para altura, ou subestimação conforme verificado para peso. Porém, ao comparar os intervalos de credibilidade das herdabilidades verificouse elevada coincidência para as diferentes análises bicaracter e unicaracter, para todas as características (tabela III).
Em função da magnitude das estimativas das herdabilidades, observou-se que as características ganho em peso diário, comprimento de cabeça e altura, apresentaram maior importância relativa das diferenças genéticas herdáveis na variação total em relação as demais, e consequentemente maior resposta à seleção.
Para todas as características nas análises unicaracter e bicaracter as herdabilidades foram inferiores as encontradas por Nguyen et al. (2007) em tilápias do Nilo, da mesma variedade, utilizando a metodologia de máxima verossimilhança restrita para a estimação dos parâmetros genéticos. Bolívar e Newkirk (2002) encontraram valores de alta magnitude de herdabilidade (0,56) em tilápias do Nilo selecionadas pela taxa de crescimento. Porém, estes autores não consideraram os efeitos maternos e nestas condições os valores das herdabilidades podem ser superestimados.
Em condições de cultivo semelhantes e com a mesma variedade, Santos et al. (2011), encontraram valores de herdabilidades superiores aos estimados neste trabalho para a característica peso final. As diferenças nas estimativas estão relacionadas às peculiaridades dos modelos estatísticos e diferenças nos conjuntos de dados.
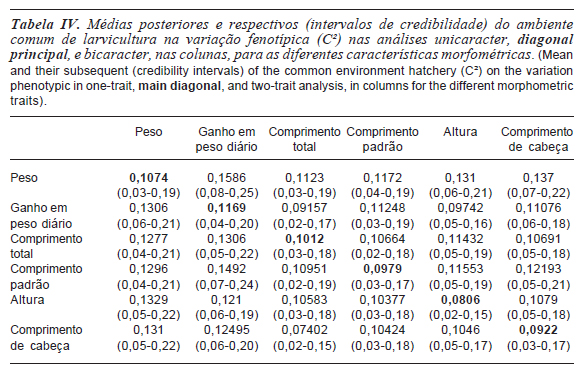
As importâncias relativas das diferenças do ambiente comum de larvicultura na variância total (C²) oscilaram entre 0,0806 a 0,1169 para as características analisadas a partir do modelo unicaracter. Enquanto nas análises bicaracter observou-se oscilação com amplitude de 0,0704 para comprimento total a 0,1586 para ganho em peso diário (tabela IV). Ao estimar a variação devido ao efeito de ambiente comum de alevinagem (W²) foi observada pequena participação relativa na variação total, apresentando valores inferiores a 3 % (tabela IV). As estimativas obtidas nas analises bicaracter confirmam esses resultados para peso, comprimento total, comprimento padrão e altura. Para ganho em peso diário e comprimento de cabeça as estimativas das médias posteriores apresentaram maior oscilação em função das características consideradas conjuntamente nas análises (tabela V).
Embora o impacto do ambiente de alevinagem estivesse mais próximo da data de mensuração das características utilizadas, apresentou menor importância relativa que o efeito de comum de larvicultura. A associação das diferenças relativas ao cuidado materno e ao número de irmãos e ao tempo que estes animais foram mantidos no mesmo hapa, pode ter tornado sua participação na variação fenotípica mais evidente.
Para o efeito do ambiente comum de família, Rutten et al. (2005), Ponzoni et al. (2005) e Santos et al. (2011), reportaram que a proporção da variância devido a esse efeito para as características corporais em tilápias variou de 12 a 26 % da variância total. Neste trabalho, o efeito do ambiente comum de família foi dividido em dois componentes, um devido ao ambiente comum de larvicultura e o outro relativo ao ambiente comum de alevinagem. A soma da participação relativa destes dois efeitos resultou em valores de média magnitude, o que demonstra o importante impacto das diferenças dos ambientes das famílias na variabilidade fenotípica.
AVALIAçãO GENÉTICA
Ao classificar os animais de acordo com os valores genéticos preditos para cada característica, foram observados valores de correlações de Pearson e Spearman superiores a 0,86 indicando que existe um relacionamento linear positivo entre as características e forte associação de ranking.
A classificação dos animais com base nos valores genéticos para ganho em peso diário apresentou-se fortemente associada com as classificações das demais características, com valores superiores a 0,76, evidenciando, pequena mudança no ranqueamento dos animais para o critério de seleção e as demais características estudadas (tabela V).
Segundo Crews Jr. e Franke (1998), coeficientes de correlações de Spearman de ordem inferiores a 0,70 podendo resultar em alterações na classificação dos animais, comprometendo a seleção dos animais.
A partir dos valores estimados para resposta à seleção direta, observou-se maior ganho genético relativo para GPD e peso, com valores superiores a 2 % ao ano. Verificou-se que o ganho genético para comprimento de cabeça foi negativo, o que implica que é possível reduzir o comprimenro de cabeça por meio de seleção (tabela VI).
Os ganhos genéticos estimados no presente trabalho foram inferiores aos relatados por Eknath et al. (2007), Bolívar e Newkirk (2002) e Santos et al. (2011). Contudo, estas diferenças pode estar associadas às intensidade de seleção e a forma de seleção. No trabalho de Santos et al. (2011) foram selecionados os melhores indivíduos das melhores famílias, enquanto que neste trabalho selecionou-se os melhores indivíduos de cada família.
Observou-se que ao selecionar os animais para GPD, a razão entre as respostas indireta/direta são superiores a 80 %. As respostas indiretas à seleção para comprimento de cabeça foram discrepantes da demais (tabela VII), pois em função da correlação genética positiva entre comprimento de cabeça e o critério de seleção, a seleção para aumento de GPD aumenta o comprimento o tamanho de cabeça, o que equivale à resposta contrária à seleção para redução do comprimento de cabeça. Estes resultados são esperados pois as respostas positivas aos comprimentos total e padrão, implicam em aumento no comprimento de cabeça. Porém, o que se deseja é que ocorra redução da participação relativa do comprimento de cabeça no comprimento do animal. Oliveira et al. (2012) relataram que após quatro anos de seleção da variedade GIFT no Brasil, a participação do comprimento de cabeça no comprimento do peixe está reduzindo. O que indica que, embora, haja associação genética positiva do comprimento da cabeça com o ganho em peso diário, as mudanças no comprimento do peixe são maiores que no comprimento da cabeça, ao selecionar-se para ganho em peso diário. Estes resultados estão, provavelmente, associados aos valores de correlações genéticas que são mais fortes entre o ganho de peso diário e os comprimento total e padrão, que aquele estimado para ao comprimento de cabeça e ganho em peso diário.
Em função das correlações genéticas existentes entre o ganho em peso diário e as demais características e das estimativas das herdabilidades, o processo de seleção utilizando como critério de seleção o ganho em peso diário, pode resultar em respostas indiretas à seleção, modificando a forma do peixe, aumentando o comprimento e altura, resultando em reprodutores que produzirão filhos com constituição genética que poderá resultar em melhora na qualidade de carcaça e rendimento de filé.
Conclusão
Os valores de herdabilidades estimados para ganho em peso diário apontam que velocidade de crescimento pode ser incrementada por meio da seleção.
As correlações genéticas estimadas indicam a existência de forte associação genética entre ganho em peso diário com peso final, altura e comprimento total. Dessa forma, a seleção para ganho em peso diário pode conduzir a ganhos genéticos indiretos para tais características.
Bibliografia
1. Bolívar, R.B. and Newkirk, G.F. 2002. Response to within family selection for body weight in Nile tilapia (Oreochromis niloticus) using a single-trait animal model. Aquaculture, 204: 371-381. [ Links ]
2. Crews Jr., D.H. and Franke, D.E. 1998. Heterogeneity of variances for carcass traits by percentage brahman inheritance. J Anim Sci, 76: 1803-1809. [ Links ]
3. Eknath, A.E.; Bentsen, H.B.; Ponzoni, R.W.; Rye, M.; Nguyen, N.H.; Thodesen, J. and Gjerde, B. 2007. Genetic improvement of farmed tilapias: Composition and genetic parameters of a synthetic base population of Oreochromis niloticus for selective breeding. Aquaculture, 273: 1-14. [ Links ]
4. FAO. 2012. Food agriculture organization. FAO yearbook. Fishery and aquaculture statistics. Aquaculture production. Roma, 2012. ftp://ftd.fao.org/FI/CDrom/CD_yearbook_2010/root/aquaculture/yearbook_aquaculture.pdf(20/11/ 2012). [ Links ]
5. Heidelberger, P. and Welch, P.D. 1983. Simulation run length control in the presence of an initial transient. Oper Res, 31: 1109-1144. [ Links ]
6. Nguyen, N.H.; Khaw, H.L.; Ponzoni, R.W.; Hamzah, A. and Kamaruzzaman, N. 2007. Can sexual dimorphism and body shape be altered in Nile tilapia by genetic means? Aquaculture, 272: 3846. [ Links ]
7. Oliveira, C.A.L.; Ribeiro, R.P.; Streit Jr, D.P.; Povh, J.A. e Resende, E.K. 2012. Melhoramento genético de peixes. Uma realidade para a piscicultura brasileira. Rev Panorama Aquicult, 22: 38-47. [ Links ]
8. Ponzoni, R.W.; Azhar, H.; Saadiah, T. and Norhidayat, K. 2005. Genetic parameters and response to selection for live weight in the GIFT strain of Nile tilapia (Oreochromis niloticus). Aquaculture, 247: 203-210. [ Links ]
9. Ponzoni, R.W. 2006. Genetic improvement effective dissemination: Keys to prosperous and sustainable aquaculture industries. In: Ponzoni, R.W.; Acosta, B.O.; Ponniah, A.G. Development of aquatic animal genetic improvement an dissemination programs. Worldfish Center. Malaysia. pp. 1-6. [ Links ]
10. Resende, E.K.; Oliveira, C.A.L.; Legat, A.P. e Ribeiro, R.P. 2010. Melhoramento genético animal: Uma visão crítica espécies aquáticas. Simpósio Brasileiro de Melhoramento Animal, VIII. Anais... Maringá- PR. Brasil. [ Links ]
11. Ribeiro, R.P. e Legat, A.P. 2008. Delineamento de programas de melhoramento genético de espécies aquicolas no Brasil. Documentos/ Embrapa Meio-Norte, no 184. [ Links ]
12. Rutten, M.J.M.; Komen, H. and Bovenhuis, H. 2005. Longitudinal genetic analysis of Nile tilapia (Oreochromis niloticus) body weight using a random regression model. Aquaculture, 246: 101-113. [ Links ]
13. Santos, A.I. 2009. Interação genótipo-ambiente e estimativas de parâmetros genéticos em tilápias (Oreochromis niloticus). Tese (doutorado). Universidade Estadual de Maringá. Programa de Pós-graduação em Zootecnia. [ Links ]
14. Santos, A.I.; Ribeiro, R.P.; Vargas, L.; Mora, F.; Filho, L.A.; Fornari, D.C. and Oliveira, S.N. 2011. Bayesian genetic parameters for body weight and survival of Nile tilapia farmed in Brazil. Pesqui Agropecu Bras, 46: 33-43. [ Links ]
15. Van Tassel, C.P. and Van Vleck, L.D. 1995. A manual for use of MTGSAM. A set of FORTRAN programs to apply Gibbs sampling to animal models for variance component estimation (DRAFT). U.S. Departament of Agriculture. Agricultural Research Service. [ Links ]
Recibido: 21-10-11
Aceptado: 12-6-13.