Mi SciELO
Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO -
 Accesos
Accesos
Links relacionados
-
 Citado por Google
Citado por Google -
 Similares en
SciELO
Similares en
SciELO -
 Similares en Google
Similares en Google
Compartir
Archivos de Zootecnia
versión On-line ISSN 1885-4494versión impresa ISSN 0004-0592
Arch. zootec. vol.60 no.232 Córdoba dic. 2011
https://dx.doi.org/10.4321/S0004-05922011000400015
Caracterización genética de tres líneas de tilapia del Nilo (Oreochromis niloticus)
Genetic characterization of three Nile tilapia (Oreochromis niloticus) strains
Lupchinski Jr., E.1*, Vargas, L.2, Lopera-Barrero, N.M.3, Ribeiro, R.P.2, Povh, J.A.3, Gasparino, E.2, Gomes, P.C.2 y Braccini, G.L.2
1Universidade Estadual de Rio Grande do Sul. Departamento de Zootecnia. Cidreira, RS. Brasil. *lupi0@yahoo.com.br
2Universidade Estadual de Maringá. Departamento de Zootecnia. Maringá, PR. Brasil.
3Universidade Federal de Mato Grosso. Instituto de Ciências Agrárias e Tecnológicas. Rondonópolis, MT. Brasil.
RESUMEN
El objetivo de este estudio fue analizar la diversidad genética de tres líneas de tilapia del Nilo (Oreochromis niloticus), mediante marcadores RAPDs. Se analizaron 90 individuos adultos (30 de cada línea) de dos piscifactorías ubicadas en las ciudades de Maringá (líneas Bouaké - B y GIFT G) y Guaíra (línea Chitralada - C), en el Estado del Paraná (Brasil). Los 13 oligonucleótidos seleccionados produjeron 72 fragmentos de los cuales 60 (83,3%) fueron polimórficos. Fueron observadas diferencias (p<0,05) en la frecuencia de 33 fragmentos, con 14 excluidos. Los resultados de variabilidad genética estimados por el porcentaje de fragmentos polimórficos y por el índice de diversidad genética de Shannon indicaron una alta variabilidad genética intra-poblacional. De acuerdo con el AMOVA, la mayor parte de la variación está dentro de cada línea. Este resultado se corroboró con los valores de FST, que mostraron una moderada diferenciación genética. También se constató que C y G fueron las líneas más semejantes genéticamente y que B y G presentaron menos genes en común. Los resultados de este estudio posibilitaran el correcto manejo re-productivo y genético de estas líneas de peces.
Palabras clave: Bouaké. Chitralada. GIFT. RAPD. Variabilidad genética.
SUMMARY
The aim of this study was to analyze the genetic diversity of three Nile tilapia (Oreochromis niloticus) strains, through the RAPD markers. Ninety adult individuals (30 of each strain) of two fish farms stations located in the Maringá (Bouaké - B and GIFT - G strains) and Guaíra (Chitralada C strain) cities, in the Paraná State (Brazil) were analyzed. The 13 selected primers yielded 72 fragments of which 60 (83.3%) were polymorphic. Differences (p<0.05) in the frequency of 33 fragments were observed, with 14 eliminated. The genetic variability results estimated by the percentage of polymorphic fragments and for the genetic diversity of Shannon index indicated a high intra-populational genetic variability. According with the AMOVA, most of the variation is within each strain. This result was corroborated with the FST values that showed a moderate genetic differentiation. It was also verified that C and G were the strains but similar genetically and that B and G presented less genes in common. The results of this study facilitated the correct reproductive and genetic management of these fish strains.
Key words: Bouaké. Chitralada. Genetic variability. GIFT. RAPD.
Introducción
En Brasil la producción total acuícola pasó de aproximadamente 30 000 toneladas en el inicio de los años noventa, hasta un total de 205 567 toneladas producidas en el año 2001 y 289 049 toneladas en el 2007 (FAO, 2009). El Estado del Paraná está entre los mayores productores nacionales de peces de agua dulce. Su producción fue de más de 19 700 toneladas en 2007, siendo representadas por 12 494 toneladas, lo que coloca el Estado como el tercer mayor productor nacional de tilapia (IBAMA, 2009).
Tilapia es el nombre común de aproximadamente 70 especies de peces clasificadas de la familia Cichlidae (Fitzsimmons, 2000). A pesar de ser oriunda del continente Africano y del Medio Oriente, la tilapia se tornó una de las especies más cultivadas en el mundo, siendo producida en más de 100 países (Romana-Eguia et al., 2004). En el Brasil, la producción de esta especie viene creciendo rápidamente en las últimas décadas desde su introducción al inicio de los años 70 (Cyrino et al., 2004) debido a características zootécnicas como su alta rusticidad, precocidad, aceptación de gran cantidad de alimentos, mejor conversión alimentaria y reproducción durante casi todo el año (Melo et al., 2006).
Por lo menos cinco líneas de tilapia del Nilo se introdujeron en el Brasil, destacándose la Bouaké y la Chitralada (Lovshin, 2000). La línea Bouaké, originaria de la Costa de Marfil, fue introducida en el país en 1971 en el Estado del Ceará, representada por apenas 60 individuos (Castagnolli, 1992). Por otro lado, la línea Chitralada, originaria de Egipto, comenzó a ser importada en 1996, cuando fueron introducidos 20 800 ejemplares en el Estado del Paraná (Zimmermann, 1999). El proyecto denominado GIFT (The Genetic Improvement of Farmed Tilapia GIFT) tuvo inicio en 1988, liderado por el órgano no gubernamental WorldFish Center (Gupta y Acosta, 2004; Li et al., 2006). La Estación Experimental de la Universidade Estadual de Maringá (UEM-CODAPAR) recibió los primeros ejemplares oriundos de 30 familias en marzo de 2005, a partir de un proyecto elaborado en conjunto con el WorldFish Center, y con el apoyo de la Secretaria Especial de Acuicultura y Pesca - SEAP Brasil (Lupchinski Jr., 2007).
El monitoreo genético de lotes de piscifactorías representa información de gran importancia para conseguir ganancias en la producción y en la conservación de peces (Feng et al., 2007). Esa evaluación es una etapa primordial, pues la variabilidad genética es fundamental para la implantación de programas de cría selectiva comercial, que tengan como objetivo la mejora genética y la producción de peces de crecimiento rápido, con mejores índices de conversión alimentaria y resistentes a enfermedades (Melo et al., 2006). Entre las técnicas moleculares, los marcadores RAPD son los más utilizados en estudios en acuicultura debido a su simplicidad, rapidez y costo relativamente bajo (Liu y Cordes, 2004).
El objetivo de este estudio fue analizar la diversidad genética de tres líneas de Oreochromis niloticus producidas comercialmente en el Brasil, mediante marcadores RAPDs.
Material y métodos
Las muestras de aletas caudales (aproximadamente 200-300 mg) se obtuvieron de 90 ejemplares adultos de tres líneas de Oreochromis niloticus (30 de cada línea) originarios de dos piscifactorías ubicadas en la ciudad de Maringá - 23o 25' S 51o 57' W (Universidade Estadual de Maringá CODAPAR: línea Bouaké y GIFT) y Guaíra -24o 04' S 54o 15' W (línea Chitralada) en el Estado del Paraná (Brasil).
Para extraer el ADN se utilizó la metodología descrita por Bardakci y Skibinski (1994), modificada por Povh et al. (2005). Fragmentos de aleta caudal se colocaron en micro-tubos con 550 µl de tampón de lisis (50 mM Tris-HCl pH 8,0, 50 mM EDTA, 100 mM NaCl, 1 % SDS) y 200 µg/ml de proteinasa K, y se incubaron en baño maría a 50oC durante 12 horas. El ADN se lavó con dos soluciones de fenol (250 µl) y tres de cloroformo (250 µl), se precipitó con dos veces y media de volumen de alcohol etílico absoluto helado y un décimo de volumen de acetato de sodio en relación al volumen recuperado (30 µl) y se incubó durante dos horas a -20oC. El ADN fue centrifugado, lavado con 2 ml de alcohol etílico 70 %, suspendido en 60 µl de tampón TE (10 mM Tris pH 8,0 y 1 mM EDTA) y tratado con 30µ/ml de RNAasa. Las muestras se incubaron durante 40 minutos en baño maría a 37oC y se conservaron a -20oC.
El ADN se cuantificó en espectrofotómetro Shimadzu (UV 1601, EE.UU.), en una longitud de onda de 260 nm y se diluyó en tampón TE a una concentración de 30 ng/ µl. La integridad del ADN se verificó en electroforesis horizontal usando un gel de agarosa al 1%, a 70 voltios durante 60 minutos, en tampón TBE 1X (500 mM Tris-HC1, 60 mM ácido bórico, 83 mM EDTA). El gel se tiñó con bromuro de etidio (0,5 µg/ml) durante 30 minutos y la imagen se capturó mediante el sistema fotográfico EDAS (Kodak 1D Image Analysis 3.5, EE.UU.).
Las condiciones de amplificación utilizadas fueron las descritas por Williams et al. (1990), con algunas modificaciones. El ADN se amplificó en un volumen de reacción de 25 µl, en el cual se utilizó tampón Tris-KCl 1X (Tris-HCl 20 mM pH 8,4 y KCl 50 mM), 2 mM MgCl2, 100 ng de oligonucleótido, 0,2 mM de cada dNTP, una unidad de Taq ADN polimerasa y 30 ng de ADN molde. Las reacciones de RAPD se amplificaron en un termociclador de PCR Eppendorf Mastercycler Gradient (EE.UU.), con el programa de 94oC por 5 min seguidos de 40 ciclos de 94oC por 1 min, 36oC por 1 min y 72oC por 2 min, con una extensión final de 72oC por 5 min. Fueron evaluados 28 oligonucleótidos del Kit Operon (Operon Technologies Inc. Alameda, CA, EE.UU.). Para evaluar las líneas se seleccionaron aquellos oligonucleótidos que presentaron características reproducibles y de consistencia adecuadas, mediante prueba de reproducibilidad (Paula, 2006). Los productos de amplificación se separaron en gel de agarosa al 1,7 %. La electroforesis se hizo con 70 voltios por cuatro horas y 30 minutos usando tampón TBE 1X. El gel se reveló utilizando el mismo protocolo en solución de bromuro de etidio.
El tamaño de los fragmentos se estimó por comparación con el patrón ADN Ladder de 100 pb (15 bandas con tamaño entre 100 y 2072 pb - Invitrogen® EE.UU.). La presencia o ausencia de fragmentos de tamaños moleculares idénticos se usó para la construcción de una matriz de similitud con base en el cálculo del coeficiente de similitud de Jaccard, codificando 1 como presencia de fragmento y 0 como su ausencia.
El índice de diversidad genética de Shannon (Lewontin, 1972) se estimó mediante la utilización del programa PopGene 1.31 (Yeh et al., 1999). El programa TFPGA 1.3(Miller, 1997) se utilizó para determinar el porcentaje de fragmentos polimórficos (critério de 95%), distancia e identidad genética (Nei, 1978) entre las líneas y la frecuencia de los fragmentos por el test exacto (Raymond y Rousset, 1995).
La divergencia genética entre las líneas fue calculada con base en el coeficiente de Jaccard, por el método de Monte Carlo, con el programa Mantel-Struct (Miller, 1999). El programa Arlequin 3.0 (Excoffieret al., 2005) se utilizó para determinar la diferenciación genética entre los lotes por médio de las estimaciones de FST (Weir y Cockerham, 1984) y para el análisis de variancia molecular (AMOVA) (Excoffieret al., 1992). Para analizar estos parametros las líneas fueron agrupadas dos a dos, en todas las combinaciones posibles, así distribuidas: Bouaké x GIFT; Bouaké x Chitralada y Chitralada x GIFT. La significación del FST fue obtenida por el test χ2 [χ2 = 2n FST (k-1);GL = (k-1) (s-1)], propuesto por Workman y Niswander (1970), en que: n es el número de individuos en los dos grupos, k es el número de alelos y s, el número de grupos. La magnitud de diferenciación genética entre los lotes fue determinada según la definición de Wright (1978), donde valores de FST entre 0,00 a 0,05; 0,05 a 0,15; 0,15 a 0,25 y > 0,25 indican respectivamente pequeña, moderada, alta y elevada diferenciación genética.
Resultados y discusión
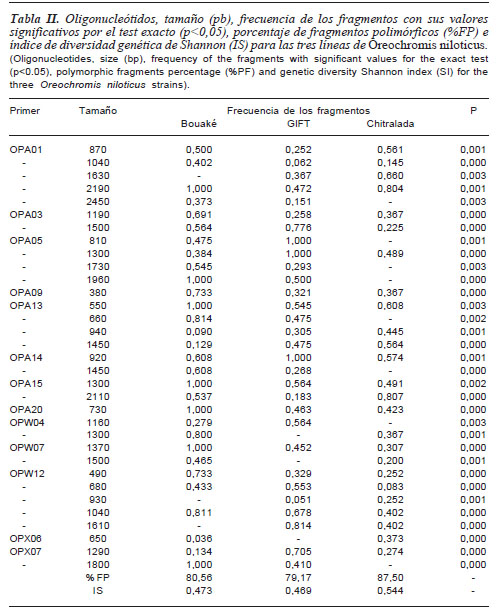
De los 28 oligonucleótidos evaluados, 13 se seleccionaron para ser analizados por presentar patrones de amplificación claros y reproducibles. En la tabla I es mostrada la secuencia de cada oligonucleótido, el porcentaje de guanina y citosina (G+C) de cada uno de ellos, los fragmentos amplificados, su polimorfismo y el tamaño en pares de bases.
Los resultados de la amplificación por los marcadores RAPDs, mostraron que el número total de fragmentos varió de tres (obtenidos con los oligonucleótidos OPA03, OPA14 y OPA15) a 9 (OPA01). El mayor fragmento (2800 pb) y el menor (380 pb) fueron obtenidos por la amplificación de los oligonucleótidos OPA20 y OPA09. Fueron encontrados un total de 72 fragmentos de los cuales 60 fueron polimórficos (83,30%) (tabla I). Según Telles et al. (2001), para la estimación de la diversidad genética por la técnica RAPD, el número de fragmentos obtenidos por el conjunto de oligonucleótidos utilizados en cualquier ensayo es más importante que el número de oligonucleótidos en cuestión. Los autores resaltaron que para bovinos, con aproximadamente 50 fragmentos seria posible estimar la variabilidad genética y obtener resultados confiables. Esto demuestra que el número de fragmentos encontrados en el presente estudio permitió una estima confiable de la variabilidad genética de las líneas de O. niloticus.
Igualmente, se observaron diferencias (p<0,05) en la frecuencia de 33 de los 72 fragmentos (tabla II), siendo determinadas características genéticas intrínsecas entre las tres líneas por la ausencia de fragmentos en algunas de ellas (Bouaké: OPA01 - 1630 pb, OPW12 - 930 pb/1610 pb; GIFT: OPW04 - 1300 pb; OPW07 - 1500 pb y OPX06 - 650 pb; Chitralada: OPA01 - 2450 pb, OPA05 810 pb/1730 pb/1960 pb; OPA13 - 660 pb; OPA14 - 1450 pb; OPW04 - 1160 pb y OPX07 - 1800 pb). Según Desvignes et al. (2001), -cuando una población pasa por un efecto cuello de botella muchos alelos con bajas frecuencias son eliminados. La presencia de alelos nulos en las líneas pudo presentarse en consecuencia de este efecto.
Se observaron 10 fragmentos limitantes, frecuencia 1,000 (siete en la línea Bouaké y tres en la GIFT), no presentando ninguno la línea Chitralada. La presencia de diversos fragmentos limitantes demuestra la presencia de alta variabilidad genética dentro de cada línea. Estos resultados son confirmados al analizar la variabilidad genética estimada por el porcentaje de fragmentos polimórficos y por el índice de diversidad genética de Shannon, donde fueron observados altos valores intra-poblacionales (tabla II). Por otro lado, fragmentos con baja frecuencia (menor que 0,1000) fueron observados en todas las líneas (dos en la línea Bouaké, dos en la GIFT y uno en la Chitralada). Si cualquiera de los alelos está en frecuencia mucho menor, como es el caso de los encontrados en este estudio, entonces su pérdida es más probable cuando existe un proceso de deriva genética.
Al comparar la variabilidad genética de las líneas Bouaké y Chitralada se verificó una alta diferenciación genética (% FP: 80,56% y 87,50%; IS: 0,473 y 0,544 respectivamente) la cual se corroboró con los resultados de divergencia genética (B x C= 0,374; B x G= 0,353 y C x G= 0,333). Esta alta diferenciación puede ser explicada por tres factores principales: el origen de cada línea, el número de individuos introducidos al país y el tiempo de introducción de cada línea.
La línea Bouaké fue importada de la Costade Marfil (África) (Castagnolli, 1992), en cuanto que la línea Chitralada fue importada de Tailandia (Asia) donde tuvo varias generaciones de cultivo (Kubitza, 2000), diferenciando geográficamente las dos líneas. Por otro lado, la línea Bouaké fue introducida al Brasil con apenas 60 ejemplares provenientes de la piscifactoría de Bouaké (Nugent, 1988). Diferentemente, la línea Chitralada se introdujo con 20 800 ejemplares procedentes del Asian Institute of Technology (AIT) (Zimmermann, 1999). El manejo genético y reproductivo de la línea Bouaké posiblemente se vio comprometido por el reducido número de reproductores disponibles, provocando el cruzamiento entre individuos emparentados por varias generaciones provocando reducción de la variabilidad genética y en consecuencia, la perdida de características zootécnicas de interés (Lima, 1999). De forma heterogénea, la manipulación genética y reproductiva de la línea Chitralada fue beneficiada por el manejo de un gran número de individuos. A pesar de no existir un registro del manejo reproductivo adoptado en el momento de la formación y conducción de cada línea, estos factores pueden haber contribuido con la diferenciación genética de las mismas.
Según Povh et al. (2005) el menor grado de polimorfismo de la línea Bouaké en relación a la Chitralada también puede ser explicado por el tiempo de introducción al Brasil de las mismas, debido a que la línea más antigua (en este caso la línea Bouaké) tiende a sufrir mayores efectos de consanguinidad y estrangulamiento genético, causados principalmente por el efecto fundador y por el manejo reproductivo específico de cada piscifactoría.
La perdida de variabilidad genética en la piscicultura siempre es esperada cuando existe un mal manejo reproductivo, debido al cruzamiento de individuos emparentados (Moreira et al., 2007) o a la formación de lotes sin suficiente variabilidad genética (Povh et al., 2008), lo que según Kang et al. (2006) consecuentemente aumenta el coeficiente de endogamia y reducirá el número efectivo de reproductores (Frost et al., 2006). Esta situación es bastante común en piscifactorías, ya que el método mas ampliamente usado en la formación de nuevos lotes es la selección de individuos con características favorables, lo que puede dar a lugar a un efecto cuello de botella (bottleneck effect) donde la variabilidad genética es reducida.
La menor variabilidad genética de la línea GIFT (%FP: 79,17%; IS: 0,469) al ser comparada con la Bouaké y la Chitralada pudo ser debida a que la GIFT fue constituida a partir del cruzamiento de cuatro líneas comerciales (provenientes de piscifactorías ubicadas en Filipinas, Israel, Singapur, Taiwán y Tailandia) y cuatro líneas salvajes (Egipto, Gana, Kenia y Senegal) de Oreochromis niloticus (Bentsen et al., 1998) mediante programas de mejoramiento genético durante 10 generaciones (Asian Development Bank, 2005), reduciendo el nivel de variabilidad genética como resultado del incremento de la homocigosis, que en consecuencia puede generar niveles variables e inevitables de consanguinidad.
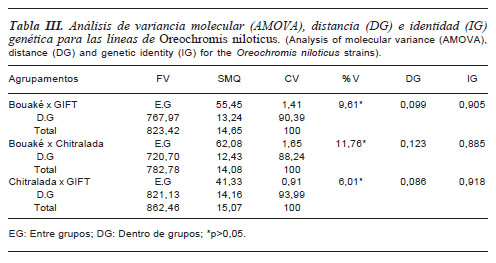
A pesar de las diferencias interpoblacionales existentes entre las líneas, la frecuencia alélica y la variabilidad genética estimadas demostraron que existe una alta diferenciación genética intra-poblacional, que indican que el establecimiento y manejo de sus lotes no interfirió de forma significativa en la pérdida de variabilidad. Confirmando esta afirmación, el análisis molecular (AMOVA) mostró una mayor variación dentro de cada línea. Entretanto, un menor porcentaje de variación entre las líneas fue observado, indicando que el pool genético de cada grupo es moderadamente semejante entre si (tabla III).
Los valores de FST según definición de Wright (1978), mostraron igualmente una moderada diferenciación genética entre las líneas, caracterizada por un valor menor para las líneas Chitralada y GIFT (0,060), un valor medio para las líneas Bouaké y GIFT (0,096) y un valor mayor para Bouaké y Chitralada (0,117). Al analizar el origen de las líneas Bouaké y Chitralada, era esperada una alta o elevada diferenciación genética entre las mismas (según la clasificación de Wright). Sin embargo fue observada una moderada diferenciación (tabla IV). Ya que el histórico de manejo de ambas líneas es desconocido y el tiempo de su introducción varía entre 12 y 37 años, es posible suponer que existan individuos híbridos fruto de deficiencias en el manejo reproductivo y genético de los lotes. No obstante, los valores de frecuencia, variabilidad genética, divergencia y AMOVA mostraron una alta diferenciación genética entre ambas, siendo posible este resultado estar relacionado a la endogamia.
Diversos autores han utilizado los valores de FST como parámetros de estimación de la variabilidad genética entre poblaciones de O. niloticus. De Souza (2007) al evaluar la caracterizar genéticamente dos lotes de reproductores (O. niloticus y línea Chitralada) encontró un valor de 0,627 indicando una elevada diferenciación genética. Bhassu et al. (2004) al analizar la estructura genética de poblaciones en Malasia encontraron un valor de 0,042 determinando una baja diferenciación. Valores todavía menores (0,035) fueron estimados por Hassanien y Gilbey (2005) para poblaciones naturales capturadas en Egipto.
Los valores estimados de identidad genética (IG) y distancia genética (DG) (tabla III) indicaron que las líneas Chitralada y GIFT fueron las más relacionadas genéticamente (IG: 0,918) y que las líneas Bouaké y Chitralada fueron las más divergentes (DG: 0,123). Las líneas Bouaké y GIFT tuvieron una distancia genética moderada (0,099). Esos resultados pueden ser explicados por el origen común de esas líneas (la Chitralada y GIFT fueron desarrolladas en Asia) a diferencia de la línea Bouaké que fue desarrollada en África. Estos resultados fueron similares a los encontrados por Massago et al. (2009), los cuales al analizar cuatro líneas de O. niloticus (GIFT, Chitralada, Supreme y Bouaké) encontraron una distancia genética menor entre las líneas GIFT y Chitralada. Igualmente, Rutten et al. (2004) al realizar la caracterización genética de cuatro líneas de O. niloticus encontraron una baja diferenciación entre las variedades GIFT y Chitralada de dos institutos de investigación, confirmando un origen genético común.
Otros autores han llevado a cabo estudios analizando líneas de O. niloticus. Appleyard y Mather (2002) al analizar cuatro líneas criadas en cautiverio en Fiji (Chitralada, Israel, Mossambicus y Roja Híbrida) observaron diferencias genéticas para todos los lotes, con una baja diversidad genética para la línea Mossambicus, característica que imposibilita su utilización en futuros programas de mejoramiento genético. Por otro lado, Povh et al. (2005) al analizar la diversidad genética de dos líneas de O. niloticus en dos generaciones, encontraron una menor variabilidad genética en la Bouaké (%FP: 18,9% para la generación de 1997 y 12,2% para la generación de 2002) cuando comparada con la Chitralada (%FP: 33,3% generación 1997 y 36,7% en la generación 2002), caracterizando la formación de dos grupos genéticamente distintos, coincidiendo con los resultados encontrados en el presente estudio. Vieira et al. (2005) analizando las mismas líneas, encontró valores de %FP de 73,53% para la línea Bouaké, valores semejantes a los encontrados en esta investigación.
Los resultados obtenidos en el presente estudio mediante el uso de la técnica RAPD permitieron obtener un perfil genético de las tres líneas estudiadas. La caracterización genética de líneas de peces puede servir como herramienta para el establecimiento de bases de selección. Como afirmaron Li et al. (2006), la mejora de la calidad genética de O. niloticus es fundamental para asegurar el futuro de la tilapicultura en varios países del mundo. Este aspecto puede ser utilizado para aumentar la variabilidad genética y explorar positivamente el efecto de la heterosis (Mather, 2001). Hassanien et al. (2004) analizando poblaciones de O. niloticus del río Nilo y de dos lagos Delta en Egipto observaron altos porcentajes de polimorfismo en tres poblaciones estudiadas (Assuit= 30,54%; Cairo= 33,5% y Qena= 44,84%) sugiriendo que la población de Qena tiene un gran potencial para su uso en programas de mejoramiento genético.
Por ser parte de los primeros estudios realizados en la línea GIFT desde su introducción en el Brasil, los resultados encontrados en este trabajo representan importantes informaciones para productores e investigadores relacionados con la producción de esta especie que actualmente tiene núcleos presentes en varios países de America Latina.
Conclusión
La variabilidad genética intra-poblacional fue alta en las tres líneas estudiadas, verificándose una semejanza genética entre la línea GIFT y Chitralada debida a su origen común. Fue observada una mayor diferenciación genética entre las líneas Bouaké y GIFT.
Bibliografía
Asian Development Bank. 2005. An impact evaluation of the development of genetically improved farmed Tilapia: and their dissemination in selected countries. Asian Development Bank. Mandaluyong. Filipinas. 14 pp. [ Links ]
Appleyard, S.A. and Mather, P.B. 2002. Genetic characterization of cultured Tilapia in Fuji using allozymes and random amplified polymorphic DNA. Asian Fish. Sci., 15: 249-264. [ Links ]
Bardakci, F. and Skibinski, D.O.F. 1994. Application of the RAPD technique in tilapia fish: species and subspecies identification. Heredity, 73: 117-123. [ Links ]
Bentsen, H.B., Eknath, A.E., Vera, M.S.P. de, Danting, J.C., Bolivar, H.L., Reyes, R.A., Dionisio, E.E., Longalong, F.M., Circa, A.V., Tayamen, M.M. and Gjerde, B. 1998. Genetic improvement of farmed tilapias: growth performance in a complete diallel cross experiment with eight strains of Oreochromis niloticus. Aquaculture, 160: 145-173. [ Links ]
Bhassu, S., Yusoff, K., Panandam, M., Embong, W.K., Oyyan, S. and Tan, S.G. 2004. The genetic structure of Oreochromis spp. (tilapia) populations in Malaysia as revealed by microsatellite DNA analysis. Biochem. Genet., 42: 217-229. [ Links ]
Castagnolli, N. 1992. Piscicultura de água doce. FUNEP. Jaboticabal. Brasil. 189 pp. [ Links ]
Cyrino, J.E.P., Urbinati, E.C., Fracalossi, D.M. and Castagnolli,N. 2004. Tópicos especiais em piscicultura de água doce tropical intensiva. TecArt. São Paulo. Brasil. 533 pp. [ Links ]
De Souza, M.E. 2007. Caracterização genética de reprodutores de tilápia: estratégias para a manutenção da variabilidade. Dissertação (Mestrado). Programa de Pós-Graduação em Recursos Pesqueiros e Aqüicultura. Universidade Federal de Pernambuco. 70 pp. [ Links ]
Desvignes, J.F., Laroche, J., Durand, S.D. and Bouvet, Y. 2001. Genetic variability in reared stocks of common carp (Cyprinus carpio) based on allozymes and microsatellites. Aquaculture, 194: 231-291. [ Links ]
Excoffier, L., Laval, G. and Schneider, S. 2005. Arlequin, v. 3.0: an integrated software package for population genetics data analysis. Evol. Bioinform. Online, 1: 47-50. [ Links ]
Excoffier, L., Smouse, P.E. and Quattro, J.M. 1992. Analysis of molecular variance inferred from metric distances among DNA haplotypes: application to human mitochondrial DNA restriction data. Genetics, 131: 479-491. [ Links ]
FAO. 2009. Food and Agriculture Organization of the United Nations. Regional review on aquaculture development 1. Latin American and the Caribbean. FAO Information Division. Roma. Italia. [ Links ]
Feng, Y., Zhang, P., Wang, K. and Xiang, J. 2007. Genetic variation of natural and cultured stocks of Paralichthys olivaceus by allozyme and RAPD. Chin. J. Oceanol. Limnol., 25: 78-84. [ Links ]
Fitzsimmons, K. 2000. Tilapia: the most important aquaculture species of the 21 century. In: Fitzsimmons, K. and J. Carvalho filho (Eds.). Proceedings from 5th International Symposium on Tilapia Aquaculture. Rio de Janeiro. Brasil. pp. 3-8. [ Links ]
Frost, L.A., Evans, B.S. and Jerry, D.R. 2006. Loss of genetic diversity due to hatchery culture practices in barramundi (Lates calcarifer). Aquaculture, 261: 1056-1064. [ Links ]
Gupta, M.V. and Acosta, B.O. 2004. From drawing board to dining table: The success story of the GIFT project. NAGA. Worldfish Center Quarterly, 27: 4-14. [ Links ]
Hassanien, H.A., Elnady, M., Obeida, A. and Itriby, H. 2004. Genetic diversity in Nile tilapia populations revealed by randomly amplified polymorphic DNA. Aquacult. Res., 35: 587-593. [ Links ]
Hassanien, H.A. and Gilbey, J. 2005. Genetic diversity and differentiation of Nile tilapia (Oreochromis niloticus) revealed by DNA microsatellites. Aquacult. Res., 36: 1450-1457. [ Links ]
IBAMA. 2009. Estatística da pesca 2007. Grandes regiões e unidades da federação. IBAMA. Brasília. Brasil. 151 pp. [ Links ]
Kang, J.H., Noh, J.K., Kim, J.H., Lee, J.H., Kim, H.C., Kim, K.K., Kim, B.S. and Lee, W.J. 2006. Genetic relationship between broodstocks of olive flounder, Paralichthys olivaceus (Temminck and Schlegel) using microsatellite markers. Aquacult. Res., 37: 701-707. [ Links ]
Kubitza, F. 2000. Tilápia: tecnologia e planejamento na produção comercial. 1a ed. F. Kubitza. Jundiaí. Brasil. 287 pp. [ Links ]
Lewontin, R.C. 1972. The apportionment of human diversity. Evolut. Biol., 6: 381-398. [ Links ]
Li, S.F., He, X.J., Hu, G.C., Cai, W.Q., Deng, X.W. and Zhou, P.Y. 2006. Improving growth performance and caudal fin stripe pattern in selected F6-F8 generations of gift Nile tilapia (Oreochromis niloticus L.) using mass selection. Aquacult. Res., 37: 1165-1171. [ Links ]
Lima, F.M. 1999. Estudo da variabilidade genética através de marcadores moleculares do tipo RAPD em algumas espécies e híbridos de tilápias (Pisces, Cichlidae). Dissertação (Mestrado). Departamento de Engenharia de Pesca. Universidade Federal do Ceará. Fortaleza. Brasil. 38 pp. [ Links ]
Liu, Z.J., and Cordes, J.F. 2004. DNA marker technologies and their applications in aquaculture genetics. Aquaculture, 238: 1-37. [ Links ]
Lovshin, L.L. 2000. Tilapia culture in Brazil. In: Costa-Pierce, B.A. and J.E. Rakocy (Eds.). Tilapia aquaculture in the Americas. The World Aquaculture Society. Baton Rouge. USA. pp. 133-140. [ Links ]
Lupchinski Jr., E. 2007. Avaliação da composição genética de linhagens de tilápia do Nilo (Oreochromis niloticus) e das gerações G0 e F1 da linhagem GIFT. Tese (Doutorado em Zootecnia). Programa de Pós-Graduação em Zootecnia. Universidade Estadual de Maringá. Maringá. Brasil. 76 pp. [ Links ]
Massago, H., Ribeiro, R.P., Lopera Barrero, N.M., Povh, J.A., Castagnolli, V. e Gomes, P.C. 2009. Diversidade genética de quatro linhagens de Oreochromis niloticus utilizando o marcador RAPD. Biosci. J., 25: 150-159. [ Links ]
Mather, P.B. 2001. Overview of fish genetics research at Queensland University of Technology. In: Gupta, M.V. and B.O. Acosta (Eds.). Fish genetics research in member countries and institutions of the International Network on Genetics in Aquaculture. ICLARM. Brisbane. Austrália. pp. 133-139. [ Links ]
Melo, D.C., Oliveira, D.A.A., Ribeiro, L.P., Teixeira, C.S., Souza, A.B., Coelho, E.G.A., Crepaldi, D.V. e Teixeira, E.A. 2006. Caracterização genética de seis plantéis comerciais de tilápia (Oreochromis) utilizando marcadores microssatélites. Arq. Bras. Med. Vet. Zootecn., 58: 87-93. [ Links ]
Miller, M.P. 1997. Tools for population genetic analyses (TFPGA): a Windows program for the analysis of allozyme and molecular population genetic data. Version 1.3. Flagstaff: Northern Arizona University. 33 pp. [ Links ]
Miller, M. 1999. MANTEL-ESTRUCT: a program for the detection of population structure via mantel tests. J. Hered., 90: 258-259. [ Links ]
Moreira, A.A., Hilsdorf, A.W.S., Silva, J.V. e Souza, V.R. 2007. Variabilidade genética de duas variedades de tilápia nilótica por meio de marcadores microssatélites. Pesq. Agropec. Bras., 42: 521-526. [ Links ]
Nei, M. 1978. Estimation of average heterozygosity and genetic distance from a small number of individual. Genetics, 89: 583-590. [ Links ]
Nugent, C. 1988. Session II, The status of wild and cultured tilapia genetic resources in various countries. In: Pullin, R.S.V. (Ed.). 2nd International Symposium on Tilapia in Aquaculture. Manilla. Philippines. pp. 20-21. [ Links ]
Paula, F.M. de. 2006. Diversidade genética de Prochilodus lineatus (Pisces Characiformes) das escadas de transposição de peixes das usinas hidroelétricas do Complexo Canoas - Rio Paranapanema. Tese (Mestrado em Genética e Biologia Molecular). Universidade Estadual de Londrina. Londrina. Brasil. 138 pp. [ Links ]
Povh, J.A., Moreira, H.L.M., Ribeiro, R.P., Prioli, A.P., Vargas, L., Blanck, D.V., Gasparino, E. e Streit Jr., D.P. 2005. Estimativa da variabilidade genética em tilápia do Nilo (Oreochromis niloticus) com a técnica de RAPD. Acta Scient. Anim. Sci., 27: 1-10. [ Links ]
Povh, J.A., Lopera-Barrero, N.M., Ribeiro, R.P., Lupchinski Jr., E., Gomes, P.C. y Lopes, T.S. 2008. Monitoreo genético en programas de repoblamiento de peces mediante marcadores moleculares. Cienc. Invest. Agr., 35: 5-15. [ Links ]
Raymond, M. and Rousset, F. 1995. An exact test for population differentiation. Evolution, 49: 1280-1283. [ Links ]
Romana-Eguia, M.R.R., Ikeda, M., Basiao, Z.U. and Taniguchi, N. 2004. Genetic diversity in farmed Asian Nile and red hybrid tilapia stocks evaluated from microsatellite and mitochondrial DNA analysis. Aquaculture, 236: 131-150. [ Links ]
Rutten, M.J.M., Komen, H., Deerenberg, R.M., Siwek, M. and Bovenhuis, H. 2004. Genetic characterization of four strains of Nile tilapia (Oreochromis niloticus L.) using microsatellite markers. Anim. Genet., 35: 93-97. [ Links ]
Telles, M.P.C., Monteiro, M.S.R., Rodrigues, F.M., Soares, T.N., Resende, L.V., Amaral, A.G. e Marra, P.R. 2001. Marcadores RAPD na análise de divergência genética entre raças de bovinos e número de locos necessários para a estabilidade da divergência estimada. Ciênc. Anim. Bras., 2: 87-95. [ Links ]
Vieira, V.P., Ribeiro, R.P., Vargas, L., Moreira, H.L.M., Povh, J.A. e Lopera-Barrero, N.M. 2005. Avaliação da variabilidade genética de linhagens de tilápia do Nilo (Oreochromis niloticus) com o uso do marcador de RAPD. Rev. Acadêm. Curitiba, 3: 41-49. [ Links ]
Weir, B.S. and Cockerham, C.C. 1984. Estimating F statistics for the analysis of population structure. Evolution, 38: 1358-1370. [ Links ]
Williams, J.G., Kubelik, A.R., Livak, J., Rafalski, J.A. and Tingey, S.V. 1990. DNA polymorphisms amplified by arbitrary primers are useful as genetic markers. Nucleic Acids Res., 18: 6531-6535. [ Links ]
Workman, P.L. and Niswander, J.D. 1970. Population studies on southwestern Indian tribes. II. Local genetic differentiation in the Papago. Amer. J. Hum. Genet., 22: 24-49. [ Links ]
Wright, S. 1978. Evolution and genetics of population. University of Chicago Press. Chicago, USA. 580 pp. [ Links ]
Yeh, F.C., Boyle,T.Y.Z. and Xiyan, J.M. 1999. PopGene, v. 131: Microsoft Window-based freeware for population genetic analysis. University of Alberta and Center for International Forestry Research. Alberta. USA. 29 pp. [ Links ]
Zimmermann, S. 1999. Incubação artificial: técnica permite a produção de tilápias do Nilo geneticamente superiores. Panorama da Aqüicultura, 9: 15-21. [ Links ]
Recibido: 21-8-08
Aceptado: 23-9-10.