Mi SciELO
Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO -
 Accesos
Accesos
Links relacionados
-
 Citado por Google
Citado por Google -
 Similares en
SciELO
Similares en
SciELO -
 Similares en Google
Similares en Google
Compartir
Archivos de Zootecnia
versión On-line ISSN 1885-4494versión impresa ISSN 0004-0592
Arch. zootec. vol.61 no.234 Córdoba jun. 2012
https://dx.doi.org/10.4321/S0004-05922012000200003
Polimorfismo genético da leptina e do receptor do hormônio do crescimento em caprinos
Genetic polymorphism of the leptin and receptor growth hormone in goats
Silva, N.M.V.1*, Ribeiro, M.N.2, Rocha, L.L.3, Gomes Filho, M.A.4 e Lima, A.P.S.5
1Departamento de Produção Animal. Universidade Federal Rural de Pernambuco. Recife/PE. Brasil. *nubiamsvieira@yahoo.com.br
2Departamento de Zootecnia. Universidade Federal Rural de Pernambuco. Recife/PE. Brasil. mn.ribeiro@uol.com.br
3Departamento de Fisiologia Animal. Universidade Federal Rural de Pernambuco. Recife/PE. Brasil. manoeladriao@yahoo.com.br
4Departamento de Zootecnia (UAST). Universidade Federal Rural de Pernambuco. Recife/PE. Brasil. laura_rocha77@yahoo.com.br
5Biotecnologia-Renorbio. Universidade Federal Rural de Pernambuco. Recife/PE. Brasil. patillima@gmail.com
Agradecimento ao Banco do Nordeste pelo financiamento deste trabalho.
RESUMO
Objetivou-se estudar a relação entre o polimorfismo no gene da leptina (LEP), especificamente o éxon 2, e o microssatélite do receptor do hormônio do crescimento (SSR-GHR) com as características de crescimento em animais das raças Anglo-Nubiana e Boer, a fim de identificar marcadores que possam ser úteis na seleção desses animais da espécie caprina de elevado mérito genético. Foram obtidas as frequências alélicas e heterozigosidade com auxílio do programa Toolkit. O teste para o equilíbrio de Hardy-Weinberg foi feito com auxílio do GENEPOP, no qual os marcadores se mostraram em desequilíbrio para as populações. Para a LEP os valores de heterozigosidade observada foram bem maiores do que os esperados e todos os animais apresentaram o mesmo padrão eletroforétrico com dois alelos (150 e 152 pb). No loco do GHR observou-se cinco alelos com tamanho variando de 90 a 125 pb. Para verificar a influência dos genótipos dos fragmentos polimórficos do GHR e da leptina sobre o desenvolvimento dos animais foram utilizados os pesos ao nascer (PN) e ao desmame (PD), para os quais foi feito análise de variância e teste de médias com auxílio do procedimento GLM do programa SAS. Observouse efeito significativo dos genótipos sobre os pesos ao nascer (PN) e à desmame (PD). Sugerese, o estudo destes polimorfismos em maior número de animais para confirmação do efeito sobre características de crescimento.
Palavras chave: Marcadores moleculares.
SUMMARY
This study aimed to evaluate the relationship between leptin gene polymorphism (LEP), specifically exon 2, and microsatellite of the growth hormone receptor (GHR-SSR) with the growth characteristics of Anglo-Nubian and Boer goats to identify useful markers for selecting animals with high genetic merit. The Toolkit program was used for obtaining the allele frequencies and heterozygosity. Hardy-Weinberg equilibrium test was performed with GENEPOP software. Markers were in desequilibrium in all populations studied. Observed heterozygosity values for LEP were greater than expected heterozygosity. All animals showed the same electrophoretic pattern with two alleles (150 and 152 bp). On GHR locus was detected five alleles ranging from 90 to 125 bp. Birth weight (BW) and weaning weight (PD) were used to evaluate the influence of polymorphic fragments on the animal growth. They were analyzed by GLM procedure of SAS software. The genotypes showed a significant effect on birth (BW) and weaning weight (PD). It is necessary to increase the number of samples to confirm the relationship among GHR and leptina polymorphic fragments GHR and growth characteristics.
Key words: Molecular markers.
Introdução
É notória a crescente preocupação com a qualidade da carne nas espécies de interesse econômico devido ao mercado em expansão e a crescente demanda por carnes mais magras e de melhor qualidade. A China, Índia, Austrália, Nova Zelândia e Turquia são os países com maior concentração de rebanhos, enquanto o Brasil vem ocupando o nono lugar (Correia, 2007). O rebanho caprino brasileiro está em torno de 9,450 milhões de cabeças, sendo a região Nordeste detentora de 8,637 milhões (IBGE, 2007). Embora o Nordeste possua condições edafoclimáticas favoráveis para a criação destes animais, (Madruga et al., 2005), o desenvolvimento do agronegócio da caprinocultura, quanto à sua tecnificação, ainda é inexpressiva, se comparada aos de bovinos e suínos. Dentre as raças caprinas com aptidão para produção de carne estão a Boer e Anglo-Nubiana, que segundo (Madruga et al., 2005), vem sendo utilizadas recentemente em cruzamentos com rebanhos SPRD (sem padrão racial definido) e também, com raças nativas como a Moxotó.
Para a cadeia produtiva da caprinocultura de corte alcançar um nível promissor e competitivo é indispensável o desenvolvimento de tecnologias que possam auxiliar a melhoria da qualidade e eficiência naprodução. É necessário profunda modificação de logística, para que ocorram mudanças no cenário nacional, em todas as classes envolvidas na produção de caprinos de corte, desde o pesquisador até o produtor.
Uso de dados moleculares como os marcadores bioquímicos e de DNA em programas de melhoramento genético ajuda o melhorista a alcançar o objetivo desejado e essas vantagens vão desde a seleção precoce de animais, por meio da análise de genes antes que ocorra sua expressão, até a seleção de características que são mensuradas após a morte do animal (Schierholt, 2005), como por exemplo, os parâmetros de qualidade da carne (força de cisalhamento e atividade da calpastatina) que requerem serem realizadas no final do ciclo produtivo.
Coutinho et al. (2007) em uma revisão sobre genômica animal, destacam que, dentre as espécies domésticas de interesse econômico, a caprina é a que tem sido menos estudada e seu genoma ainda não foi completamente estudado, com apenas 637 dbEST, ou seja, a Sequence Tag é uma subsequência pequena de cDNA transcrito, bem inferiores aos de espécies como bovina, ovina, suína, equina, canina e peixes.
O uso eficiente de marcadores moleculares deve considerar como base, o polimorfismo genético, bem como os aspectos técnicos dos métodos que os definem inclusive as vantagens e limitações de cada classe (Faleiro, 2007). Os marcadores moleculares destacam-se por serem altamente polimórficos, detectados em qualquer fase da vida do indivíduo e apresentarem características dominantes ou co-dominantes, além de não serem influenciados pelo ambiente. Uma das principais metas da pesquisa genômica tem sido a identificação de locos que controlam características quantitativas, sendo a análise de genes candidatos uma das abordagens mais utilizadas para esse fim.
A busca de genes candidatos para características ligadas à produtividade vem aumentando consideravelmente, sendo ferramenta valiosa para o melhoramento animal.
O gene candidato deve apresentar um ou mais polimorfismos nos animais parentais usados; deve-se verificar se a mutação é conservativa e em seguida, realiza-se estudos visando identificar associações com características de interesse nos programas de melhoramento (Schierholt, 2005).
Na espécie caprina são poucos os estudos realizados com essa finalidade como o de (Singh et al., 2009) com duas raças indianas, o que torna necessário ampliar os estudos, para identificação de genes candidatos com implicação em características de interesse econômico. Faz parte desta categoria de marcadores, o gene do hormônio do crescimento e o gene da leptina. Ambos exercem atividades biológicas no organismo, podendo estar associados a uma ou mais características de produção.
A caracterização do gene da leptina na região do éxon 2 tem sido analisada em diferentes espécies, como suína, bovina, bubalina, humana (Liefers et al., 2002; Zwierzchowski et al., 2001). Dois alelos (T e C) foram observados no gene da leptina com a enzima de restrição Hinf I, análise de restrição na espécie suína (Stratil et al., 1998). O padrão de polimorfismo foi associado com a deposição de gordura corporal e maior nível de RNAm da leptina no tecido adiposo em bovinos de corte (Buchanan et al., 2002). O presente estudo teve como objetivo estudar um seguimento do gene da leptina na região do éxon 2 e o microssatélite do receptor do hormônio do crescimento em duas raças de caprinos Anglo-Nubiana e Boer, investigando a ocorrência de polimorfismos e sua associação com características de crescimento, com a finalidade de gerar informações que possam auxiliar na seleção destes animais.
Material e métodos
Foram utilizados 62 caprinos, dos quais 30 da raça Boer (25 fêmeas e 5 machos) e 32 da raça Anglo-Nubiana (24 fêmeas e 8 machos) provenientes de diferentes fazendas dos municípios: Bezerros (16 Anglo-Nubiana) e Chã-Grande (15 Boer) do Estado de Pernambuco e do município Juazeiro (16 Anglo-Nubiana) e (15 Boer), Estado da Bahia.
Para as análises moleculares foram coletados cerca de 5 mL de sangue periférico de cada animal, por punção da veia jugular, utilizando-se tubos vacutainer contendo 7,5 mg de EDTA. Após a colheita, os tubos foram mantidos à -20oC até o momento da extração de DNA. No momento da coleta de material biológico também foram obtidos dados de peso ao nascer e ao desmame de cada indivíduo.
As análises foram realizadas no Laboratório de Fisiologia Animal Molecular Aplicada do Departamento de Morfologia da Universidade Federal Rural de Pernambuco.
A extração do DNA foi realizada de acordo com o protocolo descrito por Maniatis et al. (1989). Posteriormente a obtenção do DNA genômico realizou-se uma análise qualitativa.
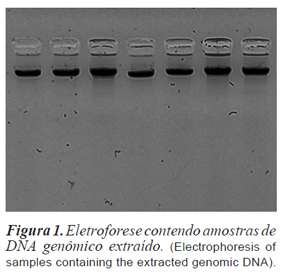
O DNA extraído mostrou-se íntegro, sem quaisquer sinais de contaminação e em boa quantidade, como pode ser observado na figura 1.
As reações de PCR foram realizadas em termociclador (Biocycler) utilizando água ultra-pura, tampão, dNTP, primers sintetizados para o gene da leptina na região do éxon 2 e para o gene do GHR.
OTIMIZAÇÃO DAS CONDIÇÕES DE AMPLIFICAÇÃO POR PCR DO ÉXON 2 DA LEP E DO GENE DO GHR
As genotipagens dos polimorfismos do éxon 2 do geneda leptina e do microssatélite do gene GHR (SSR-GHR) foram realizadas por PCR, utilizando oligonucleotídeos iniciadores (primers) descritos por Neuenschwander et al., 1996 para leptina: primer Forward - 5´- TGC AGT CTG TCT CCT CCA AA - 3´primer Reverse: 5´- CGA TAA TTG GAT CAC ATT TCT G -3´ e por Lucy et al. (1998) para o microssatélite do GHR: primer Forward - 5´ gtg ctc taa tct ttt ctg gta cca gg 3´ e primer Reverse - 5´ cct ccc caa atc aat tac att ttg tc 3PCR -leptina.
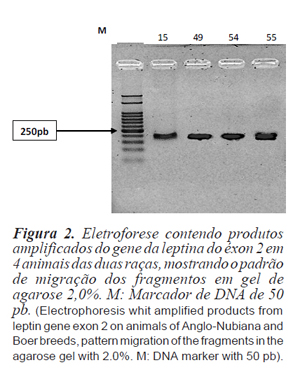
Para o éxon 2 do gene da leptina foi utilizado o PCR Optimizer Kit (Invitrogen), constituído por tampão com 200 mM de Tris-HCL e 50 mM de sulfato de amônio. Para a reação foi utilizado 2 µL de DNA genômico (20 ng/µL) adicionados a 23µL do mix de reação compostos pelos seguintes reagentes: 2,5 de um tampão de buffer; 2,5 µL de dNTP (2 mM), 1,5 µL MgCl2, 1µL (2 mM) de cada primer (10 pmoles/µL), 0,1µL (1U) de enzima Taq DNA polimerase (Invitrogen), para um volume final de 25 µL. O termociclador foi programado para 35 ciclos térmicos constituídos por: desnaturação inicial a 95oC por 2 minutos (1 ciclo); desnaturação a 95oC por 1 minuto; anelamento dos primers a 55oC por 1 minuto; extensão a 72oC por 1 minuto e extensão final a 72oC por 7 minutos (1 ciclo). Após amplificação, os amplicons foram visualizados em gel de agarose 2% para posterior digestão com enzima pela técnica PCR-RFLP, como pode ser visto na figura 2.
A analise da região do exón 2 revelaram o produto amplificado de 150 pb.
PCR-RFLP HINF I
A digestão com a enzima Hinf I (Fermentas), que reconhece sitio de restrição, 5↓G.ANTC-3´3´-CTNA↑G-5´, conduziu-se de acordo com o seguinte protocolo (cada reação): 1,5 µL tampão (10x Buffer R), 1µL enzima Hinf I (10 ng/mL), 5 µL PCR da leptina e 2,5 µL água ultra pura, tendo como produto final de 10 µL. Essa etapa foi realizada em termociclador a 37oC por 4 horas. Em segui da os produtos da digestão foram analisados em gel de poliacrilamida 5% para eficiência da enzima e avaliação dos tamanhos dos fragmentos utilizando um marcador de 10 bp (Invitrogen).
As amostras de PCR amplificadas foram submetidos à digestão Hinf-I e análise de RFLP mostrou um produto de 152 pb no exón 2 do gene da leptina em ambas as raças.
PCR-GHR
Para o microssatélite GHR a reação de amplificação (PCR) foi constituída por: 10 ng de DNA genômico; 10 ρmol de cada primer; 1,25 mM de dNTP mix; 2,5µL tampão PCR 10x concentrado (200 mM Tris-HCl pH 8,0; 50 mM KCl); 2 mM de MgCl2 e 0,2 µL e Taq DNA polimerase para um volume final de 25 µL. O termociclador foi programado para 35 ciclos térmicos constituídos por: desnaturação inicial a 94oC por 4 minutos (1 ciclo); desnaturação a 94oC por 1 minuto; anelamento dos primers a 60oC por 45 segundos; extensão a 72oC por 1 minuto e extensão final a 72oC por 4 minutos (1 ciclo). Após a PCR, os produtos da amplificação foram submetidos à eletroforese em gel de poliacrilamida 5%.
No gel de poliacrilamida foram utilizados 5 µL de cada produto de PCR e 5 µL do tampão de carregamento da amostra (NaOH, azul de bromophenol, xileno cianol em uma proporção de 1:1). Um marcador de peso molecular de 10pb (Invitrogen) foi utilizado nas eletroforeses a fim de se estimar o tamanho dos amplicons. A eletroforese foi conduzida em uma cuba vertical, a 1500 V, 60MA e 55W, com uma duração média de 1 hora e 30 minutos a uma concentração de 0,5% em tampão de TBE. A revelação dos géis de poliacrilamida foi feita com nitrato de prata.
ANÁLISES ESTATÍSTICAS
Foram obtidas as freqüências alélicas e heterozigosidade esperada (He) e observada (Ho) segundo Nei (1987) com auxílio do programa Toolkit (Park, 2001). O teste para o equilíbrio de Hardy-Weinberg foi feito com auxílio do GENEPOP versão 4.0.10, conforme Rousset (2008).
Para verificar a influência dos genótipos dos fragmentos polimórficos do GHR e da leptina sobre o desenvolvimento dos animais foram utilizadas as características peso ao nascer (PN) e peso ao desmame (PD) de animais alimentados com ração comercial. Foi feito análise de variância e teste de médias com auxílio do procedimento GLM do programa SAS (1999), de acordo com o seguinte modelo:

As médias foram submetidas ao teste de Tukey a 5% de probabilidade.
Resultados e discussão
Observou-se a amplificação de ambos os marcadores estudados nas duas raças avaliadas. O tamanho de alelos no loco do GHR variou de 90 a 125 pb enquanto a leptina apresentou-se com um padrão único de 150 e 152 pb.
O equilíbrio de Hardy-Weinberg foi feito com auxílio do GENEPOP versão 4.0.10, conforme Rousset (2008), onde os marcadores se mostraram em desequilíbrio para as populações.
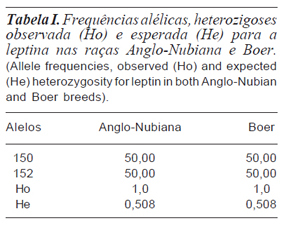
Na tabela I, encontra-se a freqüência alélica da leptina no éxon 2 nas raças Anglo-Nubiana e Boer estudas.
Os valores de heterozigosidade observada foram bem maiores do que a esperada, devido ao excesso de heterozigotos nessas populações. Todos os animais apresentaram o mesmo padrão eletroforétrico, com fragmentos de amplitude de 150 e 152 pb, gerando genótipo heterozigoto (150/152) em todos os animais (figura 3). Portanto na amostra estudada, ocorre a presença desse único genótipo no qual possui polimorfismo.
As amostras de PCR amplificados foram submetidos à digestão Hinf I e análise de RFLP mostrou um fragmento de 150 e 152 pb (figura 3), comprovando a eficácia da digestão pela Hinf I no exon 2 do gene da leptina em ambas as raças.
Singh et al. (2009) ao analisar o polimorfismo no éxon 2 do gene da leptina em caprinos indianos da raça Jamunapari e Barbari obtiveram na amplificação um produto homozigoto de 152 pb em ambas as raças.
Na figura 4, apresenta-se os produtos de PCR que foram amplificados do GHR em gel de poliacrilamida a 5%.
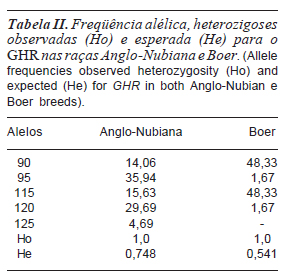
Os alelos do loco SSR-GHR apresentaram alelos de 90, 95, 115, 120 e 125 pb nos rebanhos analisados, mostrando-se bastante polimórfico. O alelo de 125 pb foi exclusivo do rebanho Anglo-Nubiana além do que no grupo Boer os alelos 95 e 120 ocorreram em freqüência muito baixa, como pode ser visto na tabela II.
O locus SSR-GHR foi um marcador de microssatélite desenvolvido para a espécie bovina. Neste estudo foram identificados cinco alelos com fragmentos de 94, 104, 106, 108 e 112 pb, descritos por Lucyet al. (1998). Leal (2009), utilizando este mesmo oligonucleotídeos iniciadores (primers) na espécie ovina com a raça Morada Nova e Santa Inês encontraram polimorfismo nas diferentes raças, independente do número de crias por parto. Contudo, quando se compararam animais de mesma raça com número de crias diferentes esse mostrou-se monomórfico.
Foram identificados os genótipos 90/ 115, 95/120 e 95/125 com freqüência de 62,9%, 32,26% e 4, 84%, para as raças estudadas, respectivamente, sendo o genótipo 95/125 específico da raça Anglo-Nubiana.
Os animais da raça Anglo-Nubiana com genótipo 95/120, 90/115 não apresentaram diferenças nas médias de pesos ao nascer. No entanto, observou-se diferenças significativas nos pesos ao desmame entre esses genótipos, sendo que os animais de genótipos 95/120 apresentaram peso superior aos demais (tabela III).
Na Raça Boer predominou o genótipo 90/115 (96,67%) e esses também apresentaram pesos ao nascer e ao desmame significativamente superiores aqueles de genótipo 95120.
O genótipo 95/125 exclusivo da raça Anglo-Nubiana apresentou a menor média para PN e PD com uma freqüência de 9,375%. A menor proporção desse genótipo sugere a possibilidade de seleção contra esse genótipo por apresentarem menor peso.
Em geral, observou-se predominância do genótipo 90/115 na raça Boer e 95/120 na raça Anglo-Nubiana, genótipos esses com maior desempenho em ambas as raças, o que sugere que a seleção está sendo direcionada possivelmente para esses genótipos pela maior capacidade produtiva que apresentam.
De uma maneira geral, a literatura é escassa em estudos com marcadores genéticos para características de produção em caprinos. Entretanto, em sistemas de produção de carne, a eficiência da seleção deve ser constantemente avaliada por estarem diretamente relacionadas à rentabilidade econômica da produção.
Os estudos com o GHR tem sido alvo em trabalhos de associação de alelos com as características quantitativas razão por este fazer parte do eixo somatotrópico e estar envolvido em grande variedade de parâmetros fisiológicos de interesse econômico (Ip et al., 2001).
Hale et al. (2000) estudando a presença do alelo S (11-TG) do receptor do hormônio de crescimento em novilhos castrados Angus criados sob condições comerciais, observaram diminuição na média de aproximadamente 17 kg na desmame e 23 kg no abate. Os resultados obtidos por Curi (2004) foram similares, mostrando o efeito negativo do alelo S sobre peso a desmame e peso de carcaça, sem, entretanto afetar área do músculo Longissimus dorsi e a deposição de gordura da carcaça.
Conclusões
As condições em que o trabalho foi realizado foi possível concluir que houve polimorfismo para o gene da leptina no n amostral nas raças Anglo-Nubiana e Boer estudadas. Estudos adicionais são necessários para verificar a real superioridade dos genótipos 95/120 para a raça Anglo-Nubiana e 90/115 para a raça Boer, observada no presente estudo.
Agradecimentos
Ao CNPq pela concessão de bolsa de estudo ao primeiro autor. Ao Programa de Pós-Graduação em Zootecnia da UFRPE e criadores, pela oportunidade.
Bibliografia
Buchanan, F.C., Fitzsimmons, C.J., Van Kessel, A.G., Thue, T.D., Winkelman-SIM, D.C. and Schmutz, S.M. 2002. Association of a missence mutation in the bovine leptin gene with carcass fat content and leptin mRNA levels. Genet Sel Evol, 34: 105-116. [ Links ]
Correia, F.W.S. 2007. Perfil setorial da caprinovino cultura no mundo, Brasil, Nordeste e Sergipe. Sebrae. http://www.biblioteca.sebrae.com.br/bds/BDS.nsf/49A7E70DA9FFD4FA832573840040EE7C/$File/PERFIL SETORIAL DA CAPRINOVINOCULTURA.pdf (10/05/2010). [ Links ]
Coutinho, L.L., Jorge, E.K., Rosario, M.F., Do Moura, A.S.A.M.T. e Ledur, M.C. 2007. Genômica Animal. http://www.cnpsa.embrapa.br/genomafrango/publica/20062007Zootec_Luiz%Coutinho_final2007.pdf (02/06/2008). [ Links ]
Curi, R.A. 2004. Relação entre os polimorfismos de genes envolvidos no controle do crescimento e na composição da carcaça e características de produção de bovinos de corte no modelo biológico superprecoce. Tese de Doutorado. Instituto de Biociências da Universidade Estadual Paulista. Campus de Botucatu. 126 pp. [ Links ]
Faleiro, F.G. 2007. Marcadores genético-moleculares aplicados a programas de conservação e uso de recursos genéticos. Embrapa Cerrados. Planaltina, DF. pp. 102. [ Links ]
IBGE. 2007. Pesquisa agropecuária municipal. http://www.sidra.ibge.gov.br/bda/pecua/ (12/03/2010). [ Links ]
Ip, S.C.Y., Zhang, X. and Leng, F.C. 2001. Genomic growth hormone gene polymorphisms in native Chinese chickens. Exp Biol Medicine, 226: 458-462. [ Links ]
Hale, C.S., Herring, W.O., Shibuya, H., Lucy, M.C., Lubahn, D.B., Keisler, D.H. and Johnson, G.S. 2000. Decreased growth in Angus steers with a short TG-microsatellite allele in the P1 promoter of the growth hormone receptor gene. J Anim Sci, 78: 2099-2104. [ Links ]
Leal, S.A.C. 2009. Caracterização genotipica de ovinos das raças Santa Inês e Morada Nova para o marcador microssatélite SSR-GHR. Relatório PIBIC-CNPq-UFRPE. [ Links ]
Liefers, S.C., Tepas, M.F., Veerkamp, R.F. and Vander Lende, T. 2002. Associations between leptin gene polymorphisms and production, live weight, energy balance, feed intake, and fertility in Holstein Heifers. J Dairy Sci, 85: 1633-1638. [ Links ]
Lucy, M.C., Johnson, G.S., Shibuya, H., Bodyd, C.K. and Herring, O. 1998. Polymorphic (GT)n microsatellite in the bovine somatotropin receptor gene promoter. J Anim Sci, 76: 2209-2210. [ Links ]
Madruga, M.S., Narain, N., Duarte, T.F., Souza, W.H., Galvão, M.S., Cunha, M.G. e Ramos, J.L. 2005. Características químicas e sensoriais de cortes comerciais de caprinos SRD e mestiços de Boer. Ciência Tecnologia Alimentos, 25: 713-719. [ Links ]
Maniatis, T., Fritsch, E.F. and Sambrook, J. 1989. Molecular cloning. A laboratory manual. V. 3, n. 1. Cold Spring Harbor Laboratory Press. New York. pp. 1-3. [ Links ]
Nei, M. 1987. Molecular evolutionary genetics. Columbia University Press. New York. 512 pp. [ Links ]
Neuenschwander, S., Rettenberger, G., Meijerink, E., Jorg, H. and Stranzinger, G. 1996. Partial characterization of porcine obesity gene (OBS) and its localization to Chromosome 18 by somatic cell hybrids. Anim Genet, 27: 275-278. [ Links ]
Park, S.D. 2001. Trypanotolerance in West African cattle and the population genetics effects of selection. Ph.D Thesis. Trinity College. University of Dublin. Ireland. [ Links ]
Rousset, F. 2008. Genepop'007: a complete reimplementation of the Genepop software for Windows and Linux. Mol Ecol Resources, 8: 103-106. [ Links ]
SAS®. 1999. User's Guide: Statistics. Version 8. SAS Institute Inc. Cary, NC. USA. [ Links ]
Schierholt, A.S. 2005. O gene da miostatina: Sequenciamento em suínos e análise filogenética. Tese. Universidade Federal de Viçosa. Programa de Pós-Graduação em Genética e Melhoramento. Viçosa. Minas Gerais. 75 pp. [ Links ]
Singh, S.K., Rout, P.K., Agarwal, R., Mandal, A., Singh, S.K., Shukla, S.N. and Roy, R. 2009. Characterization of exon 2 and intron 2 of Leptin gene in Indian goats. Anim Biotechnol, 20: 80-85. [ Links ]
Stratil, A., Kopecny, M., Moser, G., Schroffel Jr, J. and Cepioca, S. 1998. Hpa II and Rsa I PCR-RFLPs within an intron of porcine leptin receptor gene (LEPR) and its linkage mapping. Anim Genet, 29: 405. [ Links ]
Zwierzchowski, L., Opzader, J., Dymnicki, E. and Ozierzbici, P. 2001. An association of growth hormone, alpha-casein, beta-lactoglobulin, leptin and pit I loci polymorphism with growth rate and carcass traits in beef cattle. Paper and Reports. Anim Sci, 19: 65-77. [ Links ]
Recibido: 29-7-10
Aceptado: 22-9-11.