INTRODUCCIÓN
Los primeros coronavirus de procedencia humana se identificaron en la década de los 60. En el curso de un estudio sobre virus respiratorios realizado en Inglaterra en 1960, se recogió de un muchacho con resfriado una muestra, denominada B814, que contenía un virus capaz de conservar su infectividad en voluntarios a lo largo de pases sucesivos en cultivos de tráquea humana (cultivo de órganos), pero que no se replicaba de forma detectable en cultivos de células HeLa, fibroblastos humanos y células de riñón humanas y de primates, lo que lo distinguía de los virus respiratorios conocidos en la época, como adenovirus, virus de la gripe, rinovirus y otros1. Por esos mismo años, se aislaron en Chicago, también de muestras de resfriado común, cinco virus serológicamente diferenciables de los virus respiratorios conocidos2. Nuevas cepas similares fueron caracterizadas en los años siguientes, y se les adjudicaron las siglas OC en alusión a su dificultad para replicarse en cultivos celulares, lo que obligaba al uso de cultivos de órganos (OC = “organ culture”) para su aislamiento y propagación in vitro3. En 1968, un grupo de virólogos reconocieron que estas cepas y otras aisladas de animales debían constituir un grupo distinguible del de los myxovirus (en el que figuraba el virus de la gripe, y en el que habían sido incluidos inicialmente), y propusieron el nombre de coronavirus, en consideración al aspecto de los viriones, rodeados por una capa de proyecciones redondeadas4. Esta sugerencia fue recogida en el primer informe de la Comisión Internacional para la taxonomía de virus (ICTV), aparecido en 1971 (este y todos los informes del ICTV que se mencionan pueden encontrarse en la cita 5), que incluyó el género Coronavirus6, y posteriormente en su tercer informe, de 1975, se estableció la familia Coronaviridae; las cepas humanas se reunieron en la especie “Human respiratory coronavirus”, que a partir de 1976 (informe nº 4) y hasta 1991 (informe nº 13) pasó a denominarse “Human coronavirus”. El informe de 1995 (nº 14) reconoció dos especies humanas, estableciendo como prototipos las cepas 229E y OC43. El informe de 1996 (nº 15) creó el orden Nidovirales, en el que se ubicó la familia Coronaviridae, con una docena de especies, entre ellas las dos humanas reseñadas. Hasta la aparición del “Síndrome respiratorio agudo grave” (SARS, de “Severe acute respiratory sindrome”) en la provincia de Guangdong (China) en noviembre de 2002, las cepas humanas se consideraban causantes, mayoritariamente, de infecciones agudas leves del tracto respiratorio superior, es decir, formaban parte del heterogéneo conjunto de virus responsables del resfriado común; en pocos casos, se asociaron a otras manifestaciones clínicas, como otitis media en niños y neumonías, generalmente de buena evolución7. El virus responsable del SARS fue incluido como especie del género Coronavirusen el informe 22 del ICTV, aparecido en 2004. En el informe del año siguiente (nº 23) figuraba una nueva especie humana, “Human enteric coronavirus”, que se mantuvo hasta 2009; a semejanza de otros coronavirus que causan enteritis y diarrea en aves y mamíferos, se postuló que algunas enteritis humanas tendrían el mismo origen. En efecto, en heces de algunos enfermos de enteritis se han visualizado, mediante microscopía electrónica, viriones con estructura similar a la de los coronavirus, las cuales se designaron con las siglas CVLP (“coronavirus-like particles”), pero también se han observado en heces de individuos sanos; por otra parte, es posible la confusión con Torovirus, cuya morfología es similar, y que es un reconocido agente de gastroenteritis en humanos7. Actualmente, parece dudoso que existan coronavirus causantes de enteritis en humanos8, lo que justifica que la supuesta especie fuese desestimada (esto no excluye que los coronavirus respiratorios humanos pueden ocasionar síntomas intestinales). En el informe 25, de 2009, se establecieron tres géneros, Alphacoronavirus, Betacoronavirusy Gammacoronavirus; las especies humanas cuyas cepas tipo son 229E y NL63 se situaron en Alphacoronavirus, y las especies HKU1 y SARS en Betacoronavirus. El informe 26 de 2011 creó el género Deltacoronavirus, que al igual que Gammacoronavirussolo alberga especies patógenas de animales. El siguiente hecho relevante fue el reconocimiento de una nueva especie humana, responsable del Síndrome respiratorio de Oriente medio (MERS, “Middle East respiratory sindrome”). Los primeros casos de MERS fueron pacientes con neumonía grave, ocurridos en 2012 en Arabia Saudí y Jordania9; el nuevo coronavirus se incluyó en el género Betacoronavirusen 2015 (informe 30 del ICTV).
Hasta finales de 2019, se reconocían seis especies de coronavirus responsables de infecciones en humanos. Las especies tipificadas por las cepas HCoV-229E, HCoV-OC43, HCoV-NL63 y HCoV-HKU1 (HCoV son las siglas de “human coronavirus”), causantes de resfriados y rara vez de infecciones, y las emergentes SARS-CoV y MERS-CoV, con gran capacidad para causar infecciones del tracto respiratorio inferior, incluyendo neumonía atípica grave que puede evolucionar a insuficiencia respiratoria y síndrome respiratorio agudo potencialmente mortal8,10,11.
La pandemia de SARS iniciada a finales de 2002 se extendió a 29 países y el registro de la OMS de 1 de noviembre de 2002 a 31 de julio de 2003 contabilizó 8096 casos, con 774 fallecimientos (9,6 %); un esfuerzo global de contención consiguió detenerla10. Los registros de la OMS sobre los casos MERS, iniciados en septiembre de 2012, recogían a final de enero de 2020 un total de 2519, con 866 fallecimientos, lo que supone un 34,4 %; desde su inicio la enfermedad se ha extendido por los países de Oriente Medio, además de casos registrados en China, Tailandia, Inglaterra y Estados Unidos12. Tanto el SARS como el MERS han mostrado mayor gravedad en personas mayores que padezcan comorbilidades como diabetes no compensada, cardiopatías o hipertensión10; pero la capacidad de diseminación de SARS fue claramente superior a la mostrada hasta ahora por MERS11. Las diferencias entre ambos virus se explican porque, aunque incluídos en el mismo género, pertenecen a linajes filogenéticos distintos10. Los receptores celulares que utilizan son ectopeptidasas, pero mientras SARS-CoV se une a la enzima convertidora de angiotensina 2, MERS-CoV utiliza la dipeptidil-peptidasa 4 (DPP4)9. Es interesante señalar que DPP4 es escasa en las células del tracto respiratorio superior, lo que puede explicar la limitada capacidad de MERS-CoV para la transmisión de humano a humano9. La diferencia de receptores determina que SARS-CoV infecte a los neumocitos tipo I, mientras MERS-CoV infecta los neumocitos tipo II y las células bronquiales no ciliadas10. La replicación de MERS-CoV en células in vitroes más rápida y sus efectos citopáticos aparecen antes que los de SARS-CoV; MERS-CoV induce una respuesta inflamatoria más potente y muestra mayor capacidad para evadir las defensas de la inmunidad innata que SARS-CoV13, factores todos que deben relacionarse con las mayores tasas de mortalidad de MERS.
En diciembre de 2019 se registró en la populosa ciudad de Wuhan, capital de la provincia china de Hubei, un brote de neumonía de causa desconocida: la Comisión Municipal de Salud de Wuhan reportó un total de 27 casos, de los que siete se encontraban graves, con un cuadro clínico caracterizado sumariamente por fiebre, dificultad para respirar y lesiones infiltrativas de ambos pulmones14. El número de casos se incrementó con rapidez; los análisis de laboratorio excluyeron posibles agentes conocidos como adenovirus, gripe, SARS-CoV y MERS-CoV, hasta que el 9 de enero de 2020 se hizo público que se trataba de un nuevo coronavirus15. La identificación se realizó por secuenciación del RNA extraído de muestras de lavado broncoalveolar16,17; adicionalmente, el virus fue cultivado en células de epitelio respiratorio humano y en las líneas celulares Huh-7 y Vero E617. El nuevo coronavirus fue nombrado inicialmente “WH-Human 1 coronavirus” (WHCV), y con posterioridad 2019-nCoV16; finalmente, se le incluyó en la misma especie del SARS-CoV y se le denominó SARS-CoV-218; la enfermedad causada por SARS-CoV-2 se designó como COVID-19 (siglas de “Coronavirus disease 2019”)19. Ante la rápida expansión del virus a través de distintos países, el Director General de la OMS declaró la situación de pandemia el 11 de marzo de 202020. Los datos recogidos hasta el 3 de abril indican más 970.000 casos en todo el mundo, con una mortalidad promedio del 5,2 %21. En este artículo se revisan algunos de los aspectos más sobresalientes del SARS-CoV-2 y la patogenia, diagnóstico de laboratorio, tratamiento y prevención de COVID-19.
MÉTODOS
Se ha realizado un examen de publicaciones selectas sobre los distintos temas abordados en esta revisión, con las dificultades inmanentes de la superabundancia de información: a fecha de 14 de abril de 2020, el motor de búsqueda PubMed recoge 3.223 publicaciones en cuyo título aparezcan las siglas SARS-CoV-2 o COVID-19. La selección se ha realizado en base a criterios de calidad y de adecuación a los objetivos de la revisión, pero obviamente no ha pretendido ser exhaustiva ni puede considerarse un meta-análisis. Se ha recurrido también a otras fuentes de información, como libros, publicaciones de organismos oficiales y de sociedades científicas, sitios web comerciales, etc. Gran parte del material utilizado está solo en soporte electrónico previo a su publicación. Los autores son conscientes de que, en un breve margen de tiempo, nuevas investigaciones y una visión retrospectiva matizarán e incluso modificarán algunos de los conceptos, datos e interpretaciones vertidos el presente trabajo.
RESULTADOS Y DISCUSIÓN
Situación taxonómica de SARS-CoV-2
Los coronavirus son virus cuyo genoma es una molécula de RNA de cadena sencilla y polaridad positiva (lo que significa que la secuencia de bases es la misma que la de los RNAs mensajeros). Todos los virus con genoma RNA necesitan para su replicación de una enzima que no existe en las células, una RNA-polimerasa dependiente de RNA, es decir, una polimerasa que fabrica RNA tomando RNA como molde. Esta enzima debe estar, por tanto, codificada por un gen viral. En 2017 se propuso la creación de un superreino (“Realm”) denominado Riboviria, para incluir todos los taxones de virus que posean RNA-polimerasa dependiente de RNA; este superreino fue aceptado en el informe del ICTV de 2018b (nº 34), y contiene un phylum, dos subphyla, 6 clases, 10 órdenes, 7 subórdenes, 89 familias, 36 subfamilias, 387 géneros, 59 subgéneros y 2202 especies5. El orden Nidoviralespertenece a este superreino, pero no ha sido incluido, hasta el momento de redactar este artículo, en ningún phylum, subphylum ni clase. En el informe de 2018b, Nidoviralescontiene 7 subórdenes, entre ellos Cordinovirineae, con una sola familia, Coronaviridae, y dos subfamilias, de las cuales Orthocoronavirinaecontiene los cuatro géneros Alphacoronavirus, Betacoronavirus, Gammacoronavirusy Deltacoronavirus. En el género Betacoronavirushay 5 subgéneros (Figura 1); el subgénero Sarbecoviruscontiene una única especie, “Severe acute respiratory syndrome-related coronavirus”, en la que figuran los virus SARS-CoV y SARS-CoV-25,18.
Características del virión
Los coronavirus son viriones con envoltura, de aspecto esférico, con un diámetro de 120 nm; la nucleocápsida es de simetría helicoidal y contiene un genoma de RNA monocatenario, de polaridad positiva, que, con un tamaño de 27 a 32 kilobases, es el mayor entre los virus RNA22. Un esquema de la estructura del virión se muestra en la Figura 2.

Figura 2. Representación esquemática de un coronavirus. ssRNA, “single strand RNA” (RNA monocatenario)
La envoltura es una bicapa lipídica en la que se insertan tres proteínas distintas. La glicoproteína S forma trímeros que constituyen las espículas o peplómeros, que sobresalen hasta 20 nm de la superficie del virión23; cada monómero tiene una masa de unos 180 Kdal, y contiene dos subunidades, S1, que actúa como el ligando que se une a receptores de la superficie de la célula diana, y S2, que interviene en la fusión de la envoltura del virión con la membrana citoplásmica de la célula23,24. La glicoproteína M, de 25 Kdal, integrada en la envoltura, es la proteína más abundante en la envoltura del virión y cumple funciones en el ensamblaje de los viriones dentro de la célula infectada23,25,26. La proteína E, no glicosilada, es un pentámero, con monómeros de masa inferior a 8 Kdal; es la más escasa de las proteínas de envoltura, y también es crucial en el ensamblaje23,26.
La proteína N, de alrededor de 46 Kdal, se asocia al genoma para formar la nucleocápsida helicoidal23. El genoma (Figura 3) es una molécula de RNA monocatenario, de polaridad positiva, con la típica caperuza (“cap”) en el extremo 5’ y una cola de poliadenilato en el 3’27. Se han secuenciado los genomas de gran número de aislados; el genoma de referencia propuesto por GenBank posee 29.903 pares de bases16. Los genes que codifican para las cuatro proteínas estructurales figuran en el siguiente orden (del extremo 5’ al 3’): S, E, M y N; y están precedidos por los ORFs (marcos de lectura abierta) designados como 1a y 1b, que codifican para 16 proteínas no estructurales16. La comparación de los genomas de 101 cepas del virus reveló dos haplotipos, S y L, de los cuales S es la versión ancestral28.

Figura 3. Genoma del SARS-CoV-2. El nucleótido modificado del extremo 5’ constituye el cap. El conjunto de los ORFs 1a y 1b codifica para 16 proteínas no estructurales, entre ellas dos proteasas (los ORFs se traducen en poliproteínas que deben ser escindidas para liberar las proteínas individuales) y el complejo replicasa/transcriptasa que incluye la RNA-polimerasa dependiente de RNA. En el extremo 3’ se sitúa la “cola” de poliadenilato
Biología de los coronavirus
El primer paso de la infección celular por coronavirus es la unión del virión a receptores en la superficie celular. La glicoproteína S de SARS-CoV-2 se une al receptor celular, que es la enzima convertidora de angiotensina 2. La estructura de la proteína S y su interacción con el receptor han sido analizados con detalle29. La enzima convertidora de angiotensina 2 es también el receptor para SARS-CoV, pero la afinidad con la que se une la proteína S de SARS-CoV-2 es de 10 a 20 veces mayor, lo que puede explicar la mayor contagiosidad del nuevo coronavirus30. La interacción de la subunidad S1 con el receptor promueve la endocitosis del virión. El pH ácido y las proteasas endosomiales catalizan la escisión de las dos subunidades de S, y modifican S2 para que actúe como proteína de fusión, que facilita la fusión de las dos bicapas lipídicas, la envoltura del virión y la membrana de vesícula endocítica, liberando la nucleocápsida en el citoplasma de la célula infectada11. La proteasa de serina TMPRSS2 parece ser una enzima crucial en la activación de S2 para la fusión y entrada de SARS-CoV-2 en el citoplasma, por lo que los agentes inhibidores de TMPRSS2 son capaces de bloquear la infección y podrían tener aplicación terapéutica31.
Una vez en el citoplasma y tras la descapsidación, se inicia la traducción del RNA genómico, que al ser de polaridad positiva actúa como un mRNA. Se traducen los ORFs 1a y 1b, próximos al extremo 5’, con lo que la célula fabrica las poliproteínas pp1a (4.382 aminoácidos) y pp1ab (7.073 aminoácidos)11. Dos de los componentes de las poliproteínas tiene actividad proteasa y catalizan la escisión de las propias poliproteínas en proteínas individuales32, entre ellas la replicasa del virus, que es una RNA-polimerasa dependiente de RNA, y otras proteínas accesorias. Tomando como molde el RNA genómico, la replicasa fabrica la molécula complementaria completa (RNA genómico de polaridad negativa), que a su vez será tomada como molde (el denominado intermediario de replicación) para sintetizar las numerosas copias de genomas destinados a la progenie viral; y también RNAs subgenómicos, que se tomarán como moldes para los mRNAs correspondientes a los genes que codifican para las cuatro proteínas estructurales, de las cuales S y M son glicosiladas en el aparto de Golgi11,26.
La correcta distribución subcelular de las proteínas estructurales es necesaria para el ensamblaje de los componentes de los viriones hijos, que incluye la encapsidación selectiva de las copias de RNA genómico (los RNAs de polaridad negativa que se han sintetizado para actuar como moldes no son encapsidados); y los viriones hijos emergen del retículo endoplásmico en el interior de vesículas que se fusionan con la membrana citoplásmica, liberando la progenie viral11,23,25,26. La infección conduce finalmente a la muerte de la célula, posiblemente por apoptosis11; en cultivos celulares pueden observarse efectos citopáticos: redondeamiento de las células, que mueren y se desprenden de la monocapa17,33. Un esquema del proceso de replicación de los coronavirus se muestra en la Figura 4.

Figura 4. Replicación de SARS-CoV-2. El virión se une por su glicoproteína S (subunidad S1) al receptor celular (enzima convertidora de angiotensina 2), lo que induce la endocitosis. El pH ácido y las proteasas endosomiales escinden las subunidades S1 y S2 y modifican S2 para que actúe promoviendo la fusión de la envoltura viral y la membrana de la vesícula endocítica. La traducción parcial del RNA genómico conduce a la síntesis de la RNA polimerasa dependiente de RNA, que fabrica el intermediario de replicación (RNA de polaridad -), el cual actúa como molde para la síntesis de los RNAs genómicos de los viriones hijos. También se producen mRNAs para la síntesis de las proteínas estructurales. El ensamblaje de los distintos componentes sintetizados produce los viriones hijos, que emergen del retículo endoplásmico (RE) en el interior de vesículas, las cuales se fusionan con la membrana citoplásmica para liberar la progenie viral
Origen del SARS-CoV-2
Muchos coronavirus patógenos del ser humano y de otros vertebrados proceden de diversas especies de murciélagos. De hecho, más de un tercio del viroma de murciélagos secuenciado hasta la fecha consiste en coronavirus34. Tanto SARS-CoV como MERS-CoV derivaron de virus de murciélagos, aunque antes de llegar al hospedador humano pasaron por hospedadores intermediarios, la civeta de las palmeras (Paguma larvata) en el caso del SARS-CoV y el dromedario (Camelus dromedarius) en el del MERS-CoV34. En el caso del SARS-CoV-2, la secuencia de su genoma coincide en más del 96 % con la de un virus de murciélago, el SARSr-CoV RaTG13 (aislado en la provincia china de Yunnan)35; la identidad genómica con SARS-CoV es menor (alrededor del 79 %), y con MERS-CoV aún más baja (no mayor del 50 %)28. Por tanto, los análisis genómicos sugieren que SARS-CoV-2 y SARSr-CoV RaTG13 comparten un ancestro común; a partir de los murciélagos reservorios, el virus que finamente ha infectado al ser humano debió utilizar un hospedador intermediario, siendo candidatos tortugas, pangolines y serpientes36, aunque las evidencias más recientes favorecen al pangolín (Manis javanica)28,37.
El hecho de que en Wuhan existe desde 1956 un centro de investigación (“Key Laboratory of Special Pathogens and Biosafety, Wuhan Institute of Virology, Chinese Academy of Sciences”, web http://english.whiov.cas.cn/Home2016/), en el que se trabaja con coronavirus, motivó suspicacias en relación al posible origen del SARS-CoV-2; suspicacias que encontraron un argumento en el hecho de que, en 2015, científicos de dicho instituto, en cooperación con otros de USA y de Suiza, publicaron la creación de un coronavirus quimérico, con el fondo genético del SARS-CoV y la inserción de la proteína S de un coronavirus de murciélago, y describieron la gran capacidad de dicho virus quimérico para replicarse en cultivos celulares y su notable patogenicidad para el ratón38. Ya en el momento de su aparición, esta publicación motivó fuertes críticas en el sentido de que el balance riesgo/beneficios de este tipo de investigaciones podría ser peligrosamente negativo. Sin embargo, los análisis genómicos recientes han revelado como muy improbable que el SARS-CoV-2 sea un producto de laboratorio o un virus manipulado deliberadamente39.
Una pre-publicación que estuvo alojada en bioRxiv (“the pre-print server for biology”, un repositorio de acceso abierto para trabajos que aún no han superado la revisión por expertos), afirmaba la existencia de “extraña semejanza” entre insertos en la glicoproteína S del SARS-CoV-12 y dos proteínas del Lentivirus(VIH), concretamente, gp120 y Gag de VIH-1. La pre-publicación se instaló en el repositorio el 31 de enero y fue retirada el 2 de febrero, ante las numerosas críticas de la comunidad científica en relación a la tecnología empleada y a la interpretación errónea de los datos; un análisis exhaustivo dejó claro que los presuntos insertos son comunes en coronavirus, y entre otros están presentes en SARSr-CoV RaTG1337.
La capacidad del RNA genómico de los coronavirus para la recombinación ha sido reconocida desde hace tiempo23. Un ejemplo ilustrativo ha sido la detección de un coronavirus de murciélago, cuyo genoma posee un gen procedente de un Orthoreovirus; el gen p10 es funcional en el coronavirus y codifica para una proteína posiblemente implicada en la fusión de las células infectadas para formar sincitios40. Un estudio de seguimiento determinó que el coronavirus portador de un gen originario de un virus tan alejado filogenéticamente circula de forma persistente en los murciélagos hospedadores41. Por ello, no es sorprendente que antes de la aparición del SARS-CoV-2, diversos estudiosos de coronavirus predijeran la posibilidad de que de este grupo de virus emergiesen nuevos patógenos humanos3,10,11,42.
Epidemiología de COVID-19
La actual pandemia comenzó en China en diciembre de 2019 e inició su rápida expansión. El primer registro de la OMS, correspondiente al 20 de enero de 2020, computó 282 casos, de los cuales 60 se ubicaban en Wuhan, totalizando 278 en China, pero ya se detectaron dos casos en Tailandia, uno en Corea y otro en Japón. En el reporte del 23 de enero, aparece el primer caso en Estados Unidos, y en el del 25 del mismo mes, el virus llega a Europa (tres casos en Francia). La Figura 5muestra como el número de casos acumulados disparó su crecimiento a partir de la tercera semana de marzo. En España, el primer caso se detectó el 1 de febrero; la Figura 6presenta la evolución de la incidencia de nuevos casos, desde el 25 de febrero; el 31 de marzo se registró el máximo, con 9.222 casos nuevos, pero a partir de esta fecha la gráfica inicia un claro descenso.
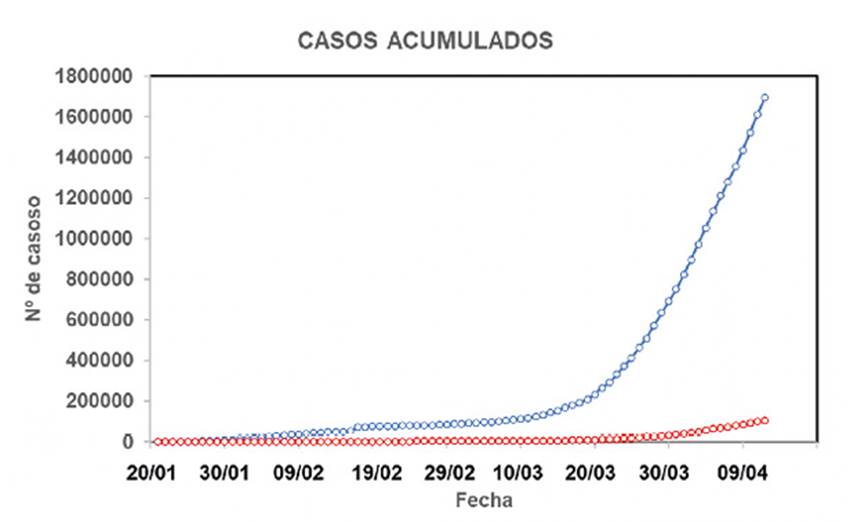
Figura 5. Casos confirmados de infección con SARS-CoV-2 (azul) y fallecimientos (rojo) en el mundo. Datos acumulativos tomados de los Reportsde la OMS sobre COVID-19 (https://www.who.int/emergencies/diseases/novel-coronavirus-2019/situation-reports)

Figura 6. Casos nuevos confirmados de infección por SARS-CoV-2 en España, hasta el 14 de abril de 2020. Datos tomados de RTVE (https://www.rtve.es/noticias/20200412/curva-contagios-muertes-coronavirus-espana-dia-dia/2010514.shtml)
El SARS-CoV-2 se transmite por contacto persona a persona y a través de secreciones de personas infectadas, principalmente gotitas respiratorias43. Las gotitas expulsadas al hablar, estornudar o toser tienen distintos tamaños; las mayores (> 5 µm) no suelen dispersarse más allá de un metro de distancia y no se mantienen en el aire más de 17 min, depositándose en el suelo o superficies de objetos; las menores (< 5 µm) permanecen en el aire por periodos largos de tiempo, formando aerosoles que pueden dispersarse a distancias mayores44. Por tanto, el contagio puede ocurrir por aspiración de las gotitas o por contacto con superficies contaminadas por ellas, que permita al virus accede a las mucosas de boca, nariz y ojos45. Experimentos realizados a lo largo de siete días, a 21-23 ºC y 40 % de humedad, revelaron que el virus puede mantenerse infectivo durante distintos periodos de tiempo, según la naturaleza de la superficie: 4 h sobre cobre, 24 h sobre cartón, 48 h sobre acero inoxidable y hasta 72 h sobre plástico46. Obviamente, este tipo de experimentos dependen mucho de los factores ambientales y del tamaño del inóculo, y no es prudente extrapolarlos a las circunstancias de la vida cotidiana. Un factor importante es la temperatura: a 56 ºC, el número de viriones infectivos se reduce un millar de veces en 10 min, y a 70 ºC en 1 min45. En términos generales, el virus es sensible a desinfectantes y antisépticos como alcohol de 70º, lejía casera, jabón, clorhexidina y povidona yodada45.
El número básico de reproducción, conocido como R0(número promedio de personas a las que una persona infectada transmitirá el virus) ayuda a predecir si una enfermedad infecciosa puede dar lugar a un brote epidémico importante, y también permite establecer la proporción de habitantes que deben ser inmunizados para crear una barrera o “cortafuegos” que impida la propagación de la enfermedad. Se han calculado distintos valores de R0para el SARS-CoV-2: entre 2,2 y 2,643, y de 1,9 a 6,547; no son los mayores valores para virus altamente contagiosos (el sarampión puede presentar valores entre 12 y 18), pero son mayores que los atribuidos a SARS-CoV y a MERS-CoV43. Dado que la estabilidad del SARS-CoV-2 en condiciones ambientales parece similar a la del SARS-CoV, la mayor contagiosidad del SARS-CoV-2 debe explicarse por otras causas, como la posibilidad de que las personas infectadas con SARS-CoV-2 eliminen y transmitan el virus mientras están asintomáticos y las altas cargas virales en el tracto respiratorio superior46. A este respecto, se ha descrito que hay una significativa carga viral en secreciones nasales y faringe, no solo de los enfermos, sino también de infectados asintomáticos, lo que confirma el papel de estos en la transmisión del virus48. Esta carga viral se debe a la capacidad del virus para replicarse en el tracto respiratorio superior desde el inicio de la infección, a diferencia del SARS-CoV que no posee esta capacidad, a pesar de que la mucosa expresa el receptor común para ambos virus49. Finalmente, debe considerarse también que la mayor afinidad de S1 del SARS-CoV-2 por el receptor30, ya mencionada anteriormente, es otro factor que contribuye a la mayor infecciosidad de este virus.
En cuanto a la tasa de mortalidad, los valores son variables según las zonas geográficas y a lo largo del tiempo. Partiendo de los datos de la Figura 5, los porcentajes de fallecimientos sobre el total de casos (siempre valores acumulados) fueron 2,17 el 1 de febrero, 3,42 el 1 de marzo, 4,93 el 1 de abril y 6,24 el 12 de abril. Varios factores pueden influir en los porcentajes de mortalidad: en primer lugar, el número de casos confirmados es siempre inferior al de casos reales de infección, ya que las pruebas de detección se hacen solo a individuos con síntomas, e incluso no a todos, según los países: es por tanto un parámetro inexacto y variable; además, la pandemia se ha extendido a países con una proporción alta de personas mayores, que suelen padecer comorbilidades que empeoran su pronóstico (ver la sección de patogenia); finalmente, es posible que algunas mutaciones en las cepas del virus influyan en su virulencia.
En el momento actual, no es posible predecir si COVID-19 adoptará un patrón estacional, semejante al de otras infecciones respiratorias virales epidémicas, si bien es cierto que se ha observado una relación lineal inversa entre la propagación del virus y los parámetros de temperatura y humedad45.
Patogénesis
Las manifestaciones clínicas de la infección por SARS-CoV-2 presentan un espectro que va desde la ausencia de síntomas hasta las formas más graves, que requieren cuidados intensivos y tienen una alta mortalidad. Según el informe del Centro de Coordinación de Alertas y Emergencias Sanitarias de 4 de abril de 2020, los síntomas más frecuentes son fiebre o reciente historia de fiebre (68,7% de los casos), tos (68,1%), dolor de garganta (24,1%), disnea (31%), escalofríos (27%), diarrea (14%) y vómitos (6%); cuando la infección se extiende a los pulmones, la neumonía (evidenciada por las imágenes radiográficas) puede ser moderada o grave; y en los casos con mala evolución clínica se presenta insuficiencia respiratoria que requiere ventilación mecánica, shock séptico, coagulación intravascular y fallo multiorgánico (incluyendo fallo renal)45.
La infección se inicia en la mucosa del tracto respiratorio superior, desde donde se extiende al inferior. Del pulmón, el virus pasa a la sangre y puede infectar órganos cuyas células expresen el receptor: corazón, riñón, tracto gastrointestinal50. Esto explica algunas de las complicaciones que pueden derivar de la infección: daño cardíaco, fallo renal, diarrea45,51.
Algunos autores diferencian tres fases en la enfermedad. En la fase I, el virus se replica en la mucosa respiratoria y ocurre la viremia; los síntomas son los propios de la infección respiratoria (tos seca), la viremia (fiebre) y, en su caso, la gastroenteritis (vómitos, diarrea); suele aparecer linfopenia. En la fase II, la infección llega al pulmón; continúan la tos y la fiebre; la neumonía puede ser leve o cursar con signos de gravedad (taquipnea, hipoxia); se acentúa la linfopenia y hay una elevación en los niveles de dímero D (un indicador de trastornos de la coagulación, que se eleva, entre otras situaciones clínicas, en las neumonías y estados de inflamación). A partir de aquí, la evolución puede ser buena, con eliminación del virus (detectable por la disminución de la carga viral) y paulatina desaparición de los síntomas, o el enfermo puede entrar en el estado crítico que caracteriza a la fase III: extrema dificultad respiratoria (el enfermo requiere respiración asistida) y un cuadro de respuesta inflamatoria sistémica (SIRS, de “Systemic Imflammatory Response Syndrome”), con su cohorte de signos (paso de fiebre a hipotermia, taquipnea, taquicardia, hipotensión) que puede llegar a un choque séptico (hipotensión refractaria, coagulación intravascular, isquemia en extremidades, fallo multiorgánico); se elevan los marcadores de inflamación (proteínas de fase aguda, ferritina)50,52.
Las primeras barreras defensivas frente a una infección viral corren a cargo de la inmunidad innata: ciertos componentes virales son reconocidos por receptores celulares, que ponen en marcha respuestas de producción de interferón de tipo I y de citocinas proinflamatorias. En el caso de los coronavirus, el propio RNA del genoma viral y los complejos de RNA bicatenario formados con el intermediario de replicación (-) y los RNAs de polaridad (+) son reconocidos por receptores intracelulares: TLR3 y TLR7, en el endosoma, y RIG-I/MDA543. Los interferones son moléculas inespecíficas capaces de detener la replicación viral en células infectadas. La respuesta inflamatoria tiene valor defensivo, ya que promueve la salida de leucocitos de los vasos sanguíneos y su acumulación en los tejidos infectados; pero también conlleva una agresión al propio tejido, consecuencia de la liberación de radicales citotóxicos por las células inflamatorias. Por ello, es crucial la regulación de la respuesta, ya que una inflamación excesiva incrementará la gravedad del proceso; la situación extrema es la inflamación generalizada (SIRS), que aparece como consecuencia de una liberación masiva de citocinas proinflamatorias (Interleucina 1, factor necrosante de tumores alfa, interleucina 6, interleucina 12, quimiocinas…), lo que se conoce como “tormenta de citocinas”53.
Las respuestas de inmunidad específica corren a cargo de los linfocitos B (respuesta de anticuerpos) y T (inmunidad celular). Mientras que la inmunidad innata actúa de inmediato, la específica, más eficiente en la eliminación de patógenos, tarda algún tiempo en desarrollarse, ya que implica complejas interacciones celulares y mecanismos de activación, proliferación y diferenciación de linfocitos específicos. No todos los anticuerpos que se producen protegen frente a la infección; algunos son capaces de neutralizar la infectividad de los viriones y de acelerar su eliminación, pero otros carecen de eficacia e incluso algunos pueden facilitar la entrada del virus en células que carezcan de receptores para él, pero que posean receptores para la parte inespecífica de las inmunoglobulinas, como ocurre con los macrófagos, neutrófilos y algunas poblaciones linfocitarias. De hecho, este mecanismo, en el que determinados anticuerpos facilitan la infección de células inmunitarias, ha sido descrito en enfermos de SARS y se ha relacionado con la derregulación de las respuestas54. En cuanto a la inmunidad celular, incluye la generación de células T citotóxicas que destruyen de forma específica a las células infectadas que están expresando antígenos virales53. Las células T también pueden contribuir a la “tormenta de citocinas”, aunque el hecho de que las fases II y III cursen con linfopenia sugiere que deben ser otras células (macrófagos, células dendríticas) los principales responsables de la respuesta inflamatoria55.
Hay una serie de factores que influyen significativamente en la gravedad de COVID-19 y sus tasas de mortalidad: la edad avanzada, y determinadas patologías previas (comorbilidades): enfermedad cardiovascular, diabetes descompensada, enfermedad pulmonar obstructiva crónica (EPOC), hipertensión, cáncer, situaciones de inmunosupresión45,52. La edad avanzada se asocia en muchos casos a las comorbilidades reseñadas; pero primariamente se asocia a la inmunosenescencia. En las personas de edad avanzada, hay un deterioro del sistema inmune, que se traduce en una capacidad disminuída para controlar infecciones, pero paradójicamente hay un estado basal proinflamatorio56. El repertorio de clones capaces de responder frente a nuevos antígenos se ve reducido, pero también disminuye la función de las células T reguladoras57, que producen citocinas antiinflamatorias. Macrófagos y neutrófilos pierden capacidades defensivas, pero hay una derregulación de los inflamasomas (complejos de multiproteínas que intervienen en la inflamación y la apoptosis)58. La linfopenia y el deterioro inmunitario en los ancianos dificultan mucho su recuperación de las situaciones de sepsis59, y hay que considerar que, adicionalmente, la propia infección por SARS-CoV-2 causa linfopenia, como ya se ha indicado.
En el extremo opuesto se encuentran los niños. Al parecer, las frecuencias de infección en niños y jóvenes menores de 18 años no son menores que las registradas en adultos, pero la evolución es mucho más leve, con tasas de mortalidad bajísimas45. Se ha propuesto que el sistema inmune infantil es muy eficaz en la eliminación de patógenos, a lo que se podría sumar la protección parcial debida a reacciones cruzadas con coronavirus causantes de algunos de los frecuentes resfriados que afectan a los niños, pero una interesante explicación se basa en la presencia de la forma soluble del receptor (enzima convertidora de angiotensina 2) en niveles mucho más altos en el plasma de niños que en el de adultos; esta forma soluble sería capaz de unirse a la proteína S del virión, que ya no podría acceder al receptor celular: quedaría neutralizado60. El hecho de que la infección sea predominantemente leve o incluso asintomática en la población infantil y juvenil sugiere que deben ocupar una notable proporción del conjunto, muy difícil de cuantificar, de infectados no diagnosticados, pero que son fuente de contagio61.
Finalmente, hay que tener presente la importancia de los factores genéticos individuales en el curso de la enfermedad; especial relevancia pueden tener los polimorfismos en el receptor del virus, que determinarán una mayor o menor afinidad y eficiencia de entrada del virus en las células, y polimorfismos en el receptor para la parte constante de inmunoglobulinas, que actuarían como anticuerpos facilitadores de la infección54.
Diagnóstico
El diagnóstico de laboratorio de la infección por SARS-CoV-2 no difiere esencialmente del de otros virus (Figura 7). Las técnicas de análisis directo buscan el cultivo e identificación del virus, o la detección de componentes suyos específicos (antígenos y secuencias del genoma), en muestras del enfermo. Las técnicas de análisis indirectos se basan en la detección de los anticuerpos específicos que el individuo infectado produce en respuesta a la presencia en su medio interno de los antígenos virales.

Figura 7. Diagnóstico de laboratorio. A, las técnicas de análisis directo detectan componentes del virus en la muestra del enfermo (secreciones respiratorias). La RT-PCR permite detectar secuencias específicas del genoma viral; los inmunoensayos identifican antígenos del virus, para lo que se usan anticuerpos monoclonales específicos. B, las técnicas de análisis indirectos buscan los anticuerpos específicos que el sistema inmune del enfermo produce en respuesta a los antígenos virales
Como ocurre con otros coronavirus, el cultivo no es útil como técnica de diagnóstico, ya que el virus no se replica bien en las líneas celulares más usuales8. El diagnóstico directo se reduce pues a la detección de secuencias génicas o de antígenos del virus. Las muestras utilizadas para ello son secreciones respiratorias de los enfermos: muestras nasofaríngeas, aspirado endotraqueal, broncoaspirado y lavado broncoalveolar62.
La detección de secuencias génicas se realiza casi exclusivamente por la reacción en cadena de la polimerasa (PCR). Esta técnica amplifica de forma exponencial fragmentos de DNA (amplicones) flanqueados por secuencias de bases conocidas, con las que han de hibridar oligonucleótidos sintéticos que actuarán como iniciadores (“primers”) de la síntesis de las copias del amplicón. Tras ciclos sucesivos de amplificación, se dispone de una cantidad suficiente como para detectarlo con gran sensibilidad. En las técnicas de PCR en tiempo real, la amplificación y la aparición de una señal de detección (generada por una sonda que reconoce una secuencia interna del amplicón, lo que refuerza la especificidad de la técnica) son simultáneas.
Como se ha descrito, la PCR amplifica y detecta DNA, pero no RNA. Por tanto, para el diagnóstico de coronavirus (igual que para el cualquier otro virus con genoma RNA) es preciso realizar una operación previa, que consiste en extraer el RNA presente en la muestra y, mediante una retrotranscriptasa (RT, enzima que, tomando RNA como molde, sintetiza DNA), fabricar el correspondiente DNA, al que se aplicará la PCR para averiguar si están presentes las secuencias específicas del virus. Este procedimiento se conoce como RT-PCR.
Los oligonucleótidos utilizados como “primers” y como sondas se diseñan de acuerdo con los datos de secuenciación del genoma del virus. Numerosas casas comerciales han desarrollado RT-PCRs para diagnóstico de COVID-2 y han publicado evaluaciones de sensibilidad y especificidad. Un resultado positivo para la presencia de un gen del SARS-CoV-2 se interpreta como probable infección; es recomendable confirmarlo mediante la detección de otro gen; por ello, son preferibles los diseños que permitan detectar simultáneamente dos genes62. Las técnicas de PCR destacan por su extremada sensibilidad. Una RT-PCR bien diseñada y adecuadamente ejecutada detecta RNA del SARS-CoV-2 desde los primeros días de la infección62. La ejecución de una RT-PCR puede consumir más de 4 h, y requiere instrumentación automatizada y personal debidamente entrenado.
Los métodos de amplificación isotérmica de ácidos nucleicos pueden ofrecer alternativas más rápidas y simples que la PCR. Alguno de estos procedimientos ya lleva años empleándose en el diagnóstico de la gripe63, y se prevé su pronta introducción como prueba rápida y portátil (pruebas POC, de “point-of-care”) en la detección de genes de SARS-CoV-2.
La detección de antígenos virales en las muestras de los pacientes requiere disponer de anticuerpos específicos como herramientas de diagnóstico. Normalmente se utilizan anticuerpos monoclonales, de origen murino, que constituyen un reactivo homogéneo y que se seleccionan buscando características óptimas de especificidad y afinidad. Existe una diversidad de inmunoensayos aptos para detectar antígenos, pero la inmunocromatografía ofrece buenas opciones de rapidez (la lectura se hace en pocos minutos) y simplicidad (no requiere aparatos ni personal especialmente entrenado). La detección de antígenos es menos sensible que la RT-PCR, pero la carga viral de SARS-CoV-2 en la nasofaringe suele ser suficientemente alta desde los primeros días de la infección como para compensar esa menor sensibilidad62, aunque la eficiencia del ensayo dependerá de la calidad del anticuerpo monoclonal usado y del diseño de la inmunocromatogafía. Las pruebas comercializadas detectan el antígeno N (la proteína de la cápsida) o el S.
Se han elaborado algoritmos diagnósticos para personas con síntomas de infección respiratoria, combinando radiografía de tórax, detección de antígeno (siempre que se trate de pruebas con sensibilidad suficiente) y RT-PCR62.
Por último, la búsqueda de anticuerpos específicos en suero o plasma de los pacientes presenta el inconveniente de que transcurre un tiempo de una semana o más entre el inicio de la infección y su aparición en niveles detectables por los inmunoensayos (Figura 8A). Durante este periodo, conocido como “ventana”, el individuo infectado es seronegativo (en cambio, la PCR y posiblemente la detección de antígenos serán positivas). Esta limitación se atenúa por el hecho de que el periodo de incubación, desde el momento del contagio hasta la aparición de los síntomas, se superpone en términos generales al periodo ventana. Por otra parte, los anticuerpos persisten durante bastante tiempo después de la curación (los individuos curados siguen siendo seropositivos cuando las pruebas directas ya se han negativizado); este inconveniente se contrarresta determinando la clase de inmunoglobulina a la que pertenecen los anticuerpos, ya que los de clase IgM, que son los primeros en aparecer, son gradualmente sustituídos por los de clase IgG (Figura 8A); por tanto, en los convalecientes y curados la IgM específica debe ser escasa o indetectable, siendo la IgG responsable de la seropositividad en estos casos. También hay que tener presente que los pacientes con inmunodeficiencias o inmunosuprimidos tendrán respuestas insuficientes62. Los inmunoensayos más utilizados para la detección de anticuerpos son las técnicas ELISA (Figura 8B) y las inmunocromatografías (Figura 8C), y ambas permiten detectar anticuerpos totales o individualizar las clases principales (IgM, IgA, IgG). Las técnicas ELISA ofrecen la ventaja de su fácil automatización, pero suelen ocupar más de una hora; las inmunocromatografías son rápidas, pero manuales. La búsqueda de anticuerpos totales o de IgG puede utilizarse en estudios retrospectivos, para evaluar la incidencia de la infección, así como para localizar individuos que puedan donar anticuerpos neutralizantes con aplicación en terapia (experimental) o profilaxis64.

Figura 8. Detección de anticuerpos específicos de virus en suero o plasma del paciente. A, cinética de la respuesta primaria: los anticuerpos son detectables varios días después de iniciada la infección; el periodo entre el inicio de la infección y la aparición de anticuerpos detectables es el “periodo ventana”, durante el cual el paciente está infectado pero es seronegativo; los anticuerpos que aparecen al principio de la respuesta pertenecen a la clase IgM, pero pronto aparecen los de clase IgG; en los convalecientes, la IgM desaparece pero persiste la IgG. B, esquema de un ELISA para detección de anticuerpos: la fase sólida (paredes de los pocillos) están recubiertas de antígeno; al incubar el suero del enfermo, los anticuerpos específicos se unen al antígeno; se añade una anti-gamma-globulina humana (un anticuerpo, obtenido en un animal, que reacciona con las inmunoglobulinas humanas) marcada con un enzima; al añadir un sustrato sintético, la enzima lo convierte en un producto coloreado; la intensidad del color es proporcional a la cantidad de anticuerpos específicos presentes en el suero problema. C, inmunocromatografía para detección de anticuerpos: la muestra de suero reacciona con antígenos solubles en el extremo de la tira, y los inmunocomplejos migran por capilaridad hacia las zonas reactivas, donde son retenidos por anticuerpos específicos para la IgM o la IgG humanas
El Ministerio de Sanidad de España ha suministrado indicaciones para la realización de las pruebas diagnósticas, clasificación de los casos (confirmados, probables, posibles), su notificación y la toma y envío de muestras65.
Tratamiento
No existe actualmente un tratamiento antiviral que haya mostrado eficacia contrastada para la COVID-19, pero hay numerosos ensayos de protocolos en marcha. Estos incluyen agentes análogos de nucleósidos, dirigidos contra la RNA-polimerasa dependiente de RNA para interferir en la replicación del virus; inhibidores de las proteasas virales, que impiden la escisión de las poliproteínas virales y, por tanto, bloquean la liberación del complejo que interviene en la replicación del genoma viral; y agentes primariamente antiparasitarios, que, por diversos mecanismos, ejercen acción antiviral frente al SARS-CoV-266.
Entre los análogos de nucleósidos, se ha propuesto el uso de dos análogos de guanina, favipiravir y ribavirin, y uno de adenina, remdesivir, de los que este último parece el más prometedor66. Remdesivir fue destinado inicialmente para combatir el Ebolavirus, pero es activo frente a coronavirus, incluyendo MERS-CoV, y se ha incluido en varios ensayos clínicos en curso67.
Lopinavir es un inhibidor de la proteasa del Lentivirus(VIH) que se administra en combinación con ritonavir, que ayuda a mantener los niveles plasmáticos de lopinavir al ralentizar su metabolización. Fue recomendado por las autoridades sanitarias chinas en el principio de la pandemia de COVID-19, pero sus resultados no han sido concluyentes, aunque sigue en ensayos clínicos67.
Las indicaciones de la cloroquina y la hidroxicloroquina fueron inicialmente como quimioterápicos para la malaria, que posteriormente se han utilizado como inmunosupresores en el tratamiento de enfermedades autoinmunes como lupus eritematoso y artritis reumatoide67. Ambos agentes tienen actividad antiviral frente a los coronavirus, explicada por la capacidad para inhibir la glicosilación del receptor celular para el virus (la enzima convertidora de angiotensina 2), lo que dificulta la unión de la espícula S; adicionalmente, inhiben la acidificación de las vesículas endocíticas, lo que bloquea la fusión y entrada del virus en el citoplasma68. Además, reducen la autofagia, interfieren con las vías de señalización activadas por los TLRs (“Toll-like receptors”, receptores de la inmunidad innata) y con la producción de citocinas, todo lo cual tiene un efecto antiinflamatorio indicado en la terapia de COVID-1969. Aunque ambas moléculas tienen efectos secundarios indeseables, la hidroxicloroquina es mejor tolerada.
La administración combinada de hidroxicloroquina y el antibiótico azitromicina ha mostrado una eficacia reforzada en la eliminación del virus en enfermos de COVID-1970. ¿Qué papel desempeña la azitromicina en este sinergismo? En principio consistiría en prevenir o tratar sobreinfecciones bacterianas que pudieran complicar la recuperación de los enfermos; pero los autores del ensayo aducen que, además, la azitromicina tiene actividad in vitrofrente a ciertos virus (Ebola, Zika)70. También pudiera ser relevante el hecho de que este antibiótico, de la familia de los macrólidos, posee actividad antiinflamatoria, que se ha aplicado en la terapia del asma alérgica71y en la prevención y tratamiento del rechazo crónico en el trasplante de pulmón72.
Otro agente antiparasitario, la ivermectina, se ha revelado recientemente como muy eficaz en la inhibición de la replicación del SARS-CoV-2 in vitro; anteriormente, ya había mostrado acción antiviral frente a diversos virus RNA, como los de la gripe, West-Nile y dengue, al parecer por interferir en el transporte de proteínas virales del citoplasma al núcleo de las células infectadas73. Será necesario evaluar su acción in vivo, y, como en el caso de la hidroxicloroquina, se cuenta con la ventaja de que se trata de moléculas ya usadas en terapia humana, por lo que sus perfiles de seguridad, dosificaciones y pautas de administración son conocidas. Una ventaja adicional de la ivermectina, en el caso de que se establezca su utilidad frente a COVID-19, es que también posee acción antiinflamatoria.
La administración de plasma de convalecientes de COVID-19, por su contenido en anticuerpos neutralizantes, y de anticuerpos monoclonales con el mismo fin, han sido propuestas como posibles opciones terapéuticas69,74.
Otros fármacos usados en el tratamiento de COVID-19 se dirigen a reducir la respuesta inflamatoria, para prevenir o tratar el SIRS; es el caso de los anticuerpos monoclonales tocilizumab y sarilumab, neutralizadores de IL-667. El uso de corticoides, anticoagulantes, y otras medidas terapéuticas, forma parte de los protocolos encaminados a luchar contra el SIRS y choque séptico.
Prevención
La vacunación es la manera más efectiva de protección frente a un agente infeccioso, siempre que la vacuna reúna una serie de condiciones: debe inducir inmunidad protectora, sin estimular reacciones inmunopatológicas indeseables (inflamación, hipersensibilidad, autoinmunidad), y deben ser funcionales en el rango de edad más amplio posible (desde niños a ancianos); a ello conviene sumar que la tecnología de producción no sea excesivamente compleja y las condiciones de almacenamiento y administración no planteen requerimientos especiales.
El desarrollo de vacunas preventivas de COVID-19 ha contado con la experiencia previa de investigaciones similares realizadas frente a SARS y MERS75. Por definición, todos los antígenos de un agente patógeno inducen respuestas específicas, pero es un hecho que no todos ellos inducen inmunidad protectora. A estos se les denomina “antígenos protectores” y deben estar presentes en la formulación de la vacunas. En el caso de SARS-CoV-2, la atención se ha dirigido a la proteína S que forma las espículas, ya que hay muchas probabilidades de que los anticuerpos dirigidos contra ella bloqueen la unión de S al receptor celular, impidiendo la infección66,76.
La urgencia en la necesidad de disponer de vacunas efectivas frente a COVID-19 no debe ir en detrimento de las necesarias evaluaciones de seguridad. Entre los riesgos a considera, figuran la posibilidad de inducir los ya mencionados anticuerpos facilitadores de la infección, que extiendan la infección a células carentes de receptores para el virus (entre ellas, células inmunitarias), o de anticuerpos no protectores que formen inmunocomplejos circulantes y, a través de la activación de la vía clásica del complemento, induzcan peligrosas reacciones inflamatorias, hechos de los que hay precedentes con vacunas candidatas para otras infecciones virales77.
Actualmente, no hay ninguna vacuna autorizada, pero hay numerosas en desarrollo, producidas con diversas tecnologías: desde las clásicas con viriones enteros inactivados o con virus atenuados, hasta las constituídas por proteína S recombinante, proteína S vectorizada por un virus recombinante atenuado, vacunas DNA y vacunas de mRNA76.















