Mi SciELO
Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO -
 Accesos
Accesos
Links relacionados
-
 Citado por Google
Citado por Google -
 Similares en
SciELO
Similares en
SciELO -
 Similares en Google
Similares en Google
Compartir
Archivos de Zootecnia
versión On-line ISSN 1885-4494versión impresa ISSN 0004-0592
Arch. zootec. vol.58 no.222 Córdoba jun. 2009
Inferência bayesiana na predição de valores genéticos do peso aos 365 dias de bovinos de corte
Bayesian inference to predict genetic values for 365 days weight in simmental, nellore and canchim cattle breeds
Madureira, A.P.1*, H.N. Oliveira2, G.J.M. Rosa3, L.F. Bezerra4 e L.F.A. Marques5
1FIOCRUZ. Av. Augusto de Lima, 1715. Barro Preto. CEP 30190-002. Belo Horizonte-MG. Brasil. *ana_paulamad@hotmail.com
2Departamento de Melhoramento e Nutrição Animal. Faculdade de Medicina Veterinária e Zootecnia UNESP-Botucatu. Caixa Postal 560. CEP 18618-000. Botucatu-SP. Brasil. hnunes@fca.unesp.br
3Department of Dairy Science. University of Wisconsin-Madison. 460 Animal Science Building 1675 Observatory Dr. Madison. Madison. Wisconsin, WI 53706. USA. grosa@wisc.edu
4Departamento de Genética. Faculdade de Medicina. USP. Av. Bandeirantes, 3900. Monte Alegre. CEP 14049-900. Ribeirão Preto/SP. Brasil. lafbezer@genbov.fmrp.usp.br
5Departamento de Zootecnia. Centro de Ciências Agrárias. UFES. Vitória. Alto Universitário s/n. CEP 29500.000. Alegre-ES. Brasil. lfernando@cca.ufes.br
RESUMO
Dados de animais Simental e Canchim, fornecidos por suas respectivas Associações Brasileiras de Criadores, e Nelore fornecidos pela Associação Nacional de Criadores e Pesquisadores foram utilizados na estimação de componentes de variância e na predição de valores genéticos para o peso aos 365 dias de idade. Os resultados obtidos pela inferência bayesiana foram comparados com aqueles obtidos pelos métodos da máxima verossimilhança restrita (REML) e melhor preditor linear não-viesado (BLUP), que se referem à metodologia mais comumente utilizada para a estimação e predição em melhoramento genético, respectivamente. Os dois métodos forneceram estimativas pontuais semelhantes, mas as distribuições marginais a posteriori obtidas com o método Bayesiano oferecem informação mais detalhada sobre os parâmetros dos modelos.
Palavras chave: Modelo animal. Parâmetros genéticos. Crescimento. Amostragem Gibbs.
SUMMARY
Data from purebred Simmental, Nellore and Canchim cattle breeds obtained from the respective Brazilian Associations of Breeders were used to estimate variance components and to predict genetic values for 365 days weight. The results obtained by Bayesian inference were compared to those from Restricted Maximum Likelihood (REML) and Best Linear Unbiased Prediction (BLUP), which are the most commonly used methods of estimation and prediction in animal breeding. The two methods presented similar point estimates but the study of the marginal posterior distributions in the Bayesian approach yields more detailed information about the parameters and other unknowns in the model.
Key words: Animal model. Beef cattle. Growth traits. Genetic parameters. Gibbs sampling.
Introdução
As raças bovinas Simental, Canchim e Nelore são muito usadas na pecuária de corte brasileira. A primeira é uma raça de origem européia, selecionada na origem como uma raça de dupla aptidão e explorada no Brasil como raça de Corte. A Raça Canchim formada no Brasil a partir da década de 1940 é proveniente de cruzamentos entre animais da raça Charolêsa, um raça européia selecionada para corte, e animais de raças zebuínas, oriundas da Índia, e que já naquela época apresentava uma grande distribuição no território nacional. A terceira é uma raça zebuína, oriunda da Índia e cuja adaptação às condições predominantes no Brasil, fez dela a raça com maior expressão numérica no Brasil. Animais da raça Nelore são utilizados em sistemas de produção comercial como animais puros e em cruzamentos com outras raças. Já os animais das outras duas raças são criados em rebanhos selecionadores ou multiplicadores e utilizados como linha paterna em cruza-mentos nos produtores comerciais. Criadores destas três raças vêm trabalhando na obtenção de animais que, utilizados como puros ou em cruzamentos se adaptem melhor às diversas condições ambientais do Brasil.
Neste sentido, existe a necessidade de se realizar a avaliação genética das raças, fornecendo assim aos criadores critérios que possam ser utilizados para seleção e melhoramento das mesmas, de acordo com o objetivo específico de cada uma das raças.
A metologia dos modelos mistos (Henderson, 1984) é a mais comumente utilizada para a análise de dados em melhoramento genético animal, quer para obtenção de estimativas dos componentes de variância por máxima verossimilhança restrita (REML), quer para a predição dos valores genéticos. Este método ficou conhecido pela sigla que representa em inglês o conjunto das propriedades de seus preditores (BLUP, melhor preditor linear não viesado). A análise bayesiana aparece como uma alternativa de grande flexibilidade, tanto em relação aos modelos que podem ser utilizados nas análises (Gianola e Fernando, 1986), quanto em relação às inferências que podem ser realizadas a partir dos resultados (Blasco, 2001). Uma análise bayesiana sob o modelo linear misto envolve tratar todos os parâmetros do modelo, incluindo os componentes de (co)variância, como variáveis aleatórias, e encontrar a distribuição a posteriori marginal dos mesmos (Wright et al., 2000).
Vários trabalhos, como os de Lôbo et al. (1997) e de Oliveira et al. (2000), vêm sendo realizados no Brasil com o objetivo de aplicar a análise bayesiana como metodologia na estimação de componentes de (co)variância e herdabilidades, e para compará-la ao método da máxima verossimilhança restrita. Outros autores mostram como a análise bayesiana permite a utilização de distribuições robustas, para o resíduo e efeitos aleatórios, como alternativas à distribuição gaussiana, em situações com a presença de valores discrepantes nas observações (Rosa, 1999; Rosa e Padovani, 2000, Pereira, 2001).
O objetivo deste trabalho é a aplicação da análise bayesiana na estimação de componentes de variância e na predição de valores genéticos para os pesos aos 365 dias de idade nas raças Simental, Nelore e Canchim, além da comparação destes resultados com aqueles obtidos pelo método REML e metodologia dos modelos mistos comumente utilizados em melhoramento genético animal. Os dados da raça Canchim foram os mesmos utilizados no trabalho de Oliveira et al. (2000).
Material e métodos
Os dados dos animais Nelore utilizados no presente trabalho são oriundos da Associação Nacional de Criadores e Pesquisadores (ANCP) e os das raças Simental e Canchim, das respectivas associações brasileiras de criadores. Para as raças Simental, Nelore e Canchim os arquivos de dados disponíveis para análise do peso aos 365 dias continham 16 564, 54 288 e 16 499 registros de genealogia e 11 180, 37 779 e 10 660 animais com dados de pesagem em idade próxima aos 365 dias, respectivamente.
Foi utilizado o software MTDFREML (Boldman et al., 1995) na estimação dos componentes de variância pelo método REML sob modelo animal e na predição dos valores genéticos pelo método BLUP. O modelo misto geral de análise incluiu os efeitos fixos de grupo de contemporâneo (animais nascidos na mesma estação de nascimento, de mesmo sexo, submetidos a um mesmo sistema de manejo e alimentação, de mesmo grupo genético, e pertencentes ao mesmo criador), classe de idade da vaca ao parto e o efeito da idade do animal na pesagem como covariável, e o efeito aleatório genético direto do animal.
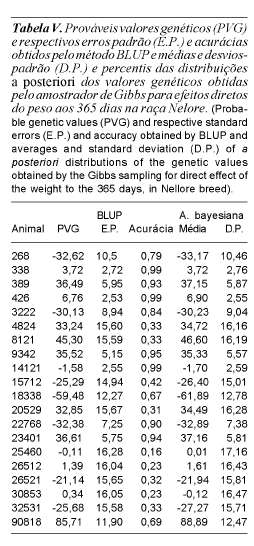
Com base nos valores genéticos preditos e erros padrão obtidos pelo BLUP, foram escolhidos alguns animais como amostra para a comparação das duas metodologias de análise. Foram selecionados 20 animais de cada raça assim distribuídos: três animais com valores genéticos baixos e negativos com acurácias altas, três animais com valores genéticos baixos e negativos com acurácias baixas, três animais com valores genéticos altos e positivos com acurácias altas, três animais com valores genéticos altos e positivos com acurácias baixas, três animais com valores genéticos médios com acurácias altas, três animais com valores genéticos médios com acurácias baixas, além do animal de mais alto valor genético e do animal de mais baixo valor genético.
O mesmo modelo de análise utilizado na estimação pelo método REML foi utilizado na análise bayesiana. O conjunto de programas MTGSAM (multiple-trait Gibbs sampler for animal models; ou modelos animais com características múltiplas usando a amostragem de Gibbs) para modelos lineares mistos, desenvolvido por Van Tassel e Van Vleck (1995), foi utilizado para obtenção das distribuições a posteriori dos componentes de variância, bem como das herdabilidades e dos valores genéticos dos animais. As distribuições a priori dos efeitos considerados no modelo são definidas no programa. Supõe-se para os efeitos fixos, uma distribuição uniforme (flat), denotando conhecimento a priori vago; e que a distribuição dos efeitos aleatórios é normal multivariada. Para os componentes de variância supõe-se distribuições Gamas invertidas independentes. Os valores a priori para a média dos componentes de variâncias foram obtidos mediante médias de estimativas descritas na literatura para esta característica. O grau de confiança nestas informações foi considerado baixo.
O esquema de amostragem Gibbs considerou 300 000 amostras, sendo desprezadas as primeiras 5000 e após esta, uma em cada 200 amostras foi armazenada para fins de inferências, num total de 1475 amostras disponíveis para estudo da distribuição a posteriori. As mesmas análises e pressu-posições foram feitas para as três raças.
Resultados e discussão
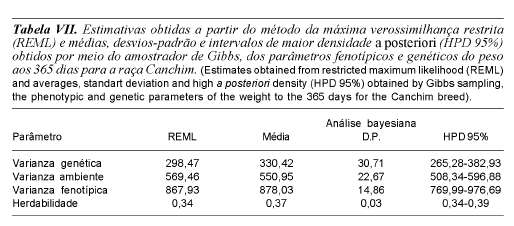
Os resultados das estimativas de componentes de variância obtidas pelo método REML e utilizando-se Inferência bayesiana estão apresentados nos tabelas I, IV e VII para as raças Simental, Nelore e Canchim, respectivamente. Pode-se verificar que as médias a posteriori obtidas pelo método Bayesiano são bastante próximas das estimativas pontuais obtidas pelo método REML, para as três raças. Em Wright et al. (2000), os autores notaram que a estimativa pontual para a variância genética aditiva obtida pelo método REML foi consideravelmente menor que a média a posteriori obtida por amostragem Gibbs. Estes resultados não são contraditórios uma vez que Wright et al. (2000) utilizaram um conjunto de dados com tamanho amostral bem menor do que os considerados no presente trabalho. Em situações com tamanhos amostrais relativamente menores, as distribuições a posteriori para os componentes de variância são geralmente mais assimétricas e, conseqüentemente, as respectivas médias são diferentes das modas, as quais, em geral, são mais próximas das estimativas de máxima verossimilhança. Além disso, é preciso ressaltar que o método REML encontra modas conjuntas e a Inferência bayesiana trabalha com distribuições marginais e elas não têm necessariamente que concordar, e de fato não concordam para os componentes de variância em distribuições multimodais, por exemplo.
A inferência bayesiana permite esta-belecer regiões de credibilidade para cada parâmetro ou combinação de parâmetros, as quais são facilmente construídas a partir das amostras obtidas no esquema de amostragem Gibbs. Assim, por exemplo, para herdabilidade do peso aos 365 dias, os intervalos de maior densidade a posteriori (HPD; do inglês highest posterior density), com 95% de probabilidade, foram obtidos entre 0,28 e 0,39 para a raça Simental; entre 0,38 a 0,44 na raça Nelore; e entre 0,34 e 0,39 para a raça Canchim.
Nas tabelas II, V e VIII são comparados os valores genéticos preditos e respectivas medidas de variabilidade pelos métodos BLUP e Bayesiano, respectivamente para as raças Simental, Nelore e Canchim. Pode-se observar que, além das médias a posteriori serem muito similares aos valores preditos (BLUP), os desvios padrão a posteriori são muito semelhantes aos erros padrão da predição. Como a distribuição é simétrica, o desvio padrão da distribuição posterior é um bom indicativo da acurácia.
Nas tabelas III, VI e IX estão os resultados das comparações entre quatro pares de animais, utilizando-se as respectivas distribuições marginais a posteriori. Na primeira situação, apresentam-se as diferenças entre o animal de maior valor genético e o de menor valor genético. Percebe-se, como esperado, que as diferenças são de grande magnitude em média (aproximadamente 143, 151 e 100 kg, para as raças Simental, Nelore e Canchim, respectivamente), mas a variação também é relativamente grande (de 46 a 247 kg, de 96 a 207 kg e de 39 a 152 kg, respectivamente as três raças). Os limites para os intervalos de maior densidade a posteriori com 95% de probabilidade foram 94,3 e 185,6 kg para a raça Simental; 122,9 e 183,6 kg para a raça Nelore e 75,9 e 129,3 kg para a raça Canchim.
Numa segunda situação, são apresen-tadas as diferenças entre dois animais de baixa acurácia. Esta comparação é praticamente inútil, dada a grande variação encontrada nas diferenças amostrais. Embora a diferença média seja de -0,81; 0,13 e 6,48 kg, os desvios padrão de 38,1; 24,15 e 25,09 kg, respectivamente para as raças Simental, Nelore e Canchim, são muito altos. Os intervalos de maior densidade a posteriori (95% de probabilidade) obtidos estão entre -62,8 e 59,6 kg para a raça Simental; entre -37,0 e 40,5 kg para a raça Nelore e entre -33,0 e 46,3 kg para a raça Canchim. Como estas distribuições mar-ginais apresentam alta densidade ao redor do valor zero (referente ao ponto onde não existe diferença entre os dois animais comparados), tem-se pouca evidência da superioridade de um animal em relação ao outro. A terceira situação refere-se à comparação de animais de alta acurácia. Ao contrário da situação anterior, esta comparação é bastante precisa e revela, para médias de -1,7; -3,2 e 17,4 kg, desvios padrão de 13,7; 3,67 e 4,7 kg, respectivamente. Os intervalos de maior densidade a posteriori (95%) estão entre -24,4 e 19,4 kg para a raça Simental; entre -8,7 e 3,1 kg para a raça Nelore e entre 9,8 e 24,6 kg para a raça Canchim.
Na quarta situação, são comparados dois animais de alta acurácia, um com alto valor genético e outro de baixo valor genético. Nesta situação a comparação também é bastante precisa, revelando médias de 32,2; 38,9 e 49,1 kg e desvios padrão de 14,8; 6,24 e 11,54 kg, respectivamente para as raças Simental, Nelore e Canchim. Os intervalos de maior densidade a posteriori (95%) estão entre 7,3 e 55,7 kg para a raça Simental; entre 28,0 e 48,4 kg para a raça Nelore e entre 29,1 e 67,1 kg para a raça Canchim. Para as três comparações, têm-se intervalos com maior massa de densidade para valores positivos, indicando a superioridade do valor genético de um animal em relação ao outro.
As densidades das distribuições a posteriori marginais para cada uma dessas situações são apresentadas nas figuras 1, 2 e 3, respectivamente para as raças Simental, Nelore e Canchim.
A tabela X apresenta as médias, desvios-padrão e percentis (2,5 e 97,5%) das distribuições a posteriori para animais de maiores valores genéticos, e para a média dos mesmos, nas raças Simental, Nelore e Canchim. As densidades das distribuições para cada uma das raças são apresentadas nas figuras 4, 5 e 6, respectivamente para as raças Simental, Nelore e Canchim.
As densidades marginais de interesse são facilmente estimadas a partir das amostras do esquema Gibbs na análise bayesiana. A partir das mesmas, são também obtidas as médias, desvios-padrão e intervalos HPD, como apresentados no trabalho, ou ainda outras quantidades a posteriori de interesse, como modas, medianas, percentis, etc. Já num contexto de inferência clássica, por exemplo, utilizando-se máxima verossimilhança, muitas vezes não é possível a obtenção de forma analítica explícita no estudo de alguns parâmetros ou funções paramétricas, ou na obtenção de intervalos de confiabilidade para os mesmos, e sua construção só é possível a partir de aproximações ou justificativas assintóticas.
Conclusões
As duas metodologias estatísticas utilizadas apresentaram, conforme esperado, estimativas pontuais similares. A abor-dagem bayesiana, embora demande maior recurso computacional, é mais flexível, fornece uma descrição mais detalhada dos parâmetros e permite a incorporação de informações a priori. O desenvolvimento de computadores mais potentes, especialmente com grande capacidade de armaze-nagem de informações deverá facilitar a utilização de metodologias bayesianas de análise, e permitirá comparar animais de maneira mais objetiva e interessante para os criadores.
Bibliografia
Blasco, A. 2001. The Bayesian controversy in animal breeding. J. Anim. Sci., 79: 2023-2046. [ Links ]
Boldman, K.G., L.A. Kriese and D.L. Van Vleck. 1995. A manual for use of MTDFREML - A set of programs to obtain estimatives of variances and covariances [DRAFT]. USDA-ARS. 114 p. [ Links ]
Gianola, D. and R.L. Fernando. 1986. Bayesian methods in animal breeding theory. J. Anim. Sci., 63: 217- 244. [ Links ]
Henderson, C.R. 1984. Applications of linear models in animal breeding. 1st ed. Canadian cataloguing in publication data. Canadá. 461 p. [ Links ]
Lôbo, R.B., H.N. Oliveira, L.A.F. Bezerra, E.N. Martins, A.L.R. Borjas, C.U. Magnabosco e A.R.V. Russo. 1997. Estimativa de componentes de (co)variância e herdabilidade para o peso aos 120 dias de idade na raça Nelore usando estatística bayesiana. Em: Reunião anula da Sociedade Brasileira de Zootecnia, 34, Juiz de Fora. Anais... SBZ. Juiz de Fora. p. 186-189. [ Links ]
Oliveira, H.N., R.B. Lôbo, G.J.M. Rosa e L.A.F. Bezerra. 2000. Análise bayesiana na estimação de componentes de covariância e predição de valores genéticos do peso aos 365 dias em bovinos. Canchim. Em: Reunião Anual da Sociedade Brasileira de Zootecnia, 37, 2000. ANAIS. Sociedade Brasileira de Zootecnia. Viçosa/MG. p. 191-191. [ Links ]
Pereira, I.G. 2001. Estudo de simulação e aplicação de modelos lineares mistos com distribuição normal contaminada no melhoramento genético animal. (Tese. Doutorado em Zootecnia). Botucatu, FMVZ/UNESP. 91 p. [ Links ]
Rosa, G.J.M. 1999. Robust mixed linear models in quantitative genetics: Bayesian analysis via Gibbs sampling. In: International Symposium on Animal Breeding and Genetics, 1999. Viçosa. Proceedings Viçosa. p. 133-160. [ Links ]
Rosa, G.J.M. e C.R. Padovani. 2000. Modelos lineares robustos utilizando-se a distribuição normal contaminada. Rev. Mat. Estatíst., 18: 65-81. [ Links ]
Van Tassel, C.P. and D.L. Van Vleck. 1995. A manual for use of MTGSAM - A set of FORTRAN programs to apply Gibbs Sampling to animal models for variance component [DRAFT]. USDA-ARS. [ Links ]
Wright, D.R., H.S. Stern and P.J. Berger. 2000. Comparing traditional and Bayesian analyses os selection experiments in animal breeding. J. Agric. Biol. Envir. S., 5: 240-256. [ Links ]
Recibido: 22-2-07
Aceptado: 17-9-08












 Curriculum ScienTI
Curriculum ScienTI