Mi SciELO
Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO -
 Accesos
Accesos
Links relacionados
-
 Citado por Google
Citado por Google -
 Similares en
SciELO
Similares en
SciELO -
 Similares en Google
Similares en Google
Compartir
Archivos de Zootecnia
versión On-line ISSN 1885-4494versión impresa ISSN 0004-0592
Arch. zootec. vol.60 no.231 Córdoba sep. 2011
https://dx.doi.org/10.4321/S0004-05922011000300043
Estrutura populacional e variabilidade genética da raça caprina Marota#
Population structure and genetic variability of the Marota goat breed
Barros, E.A.1, Ribeiro, M.N.2*, Almeida, M.J.O.3e Araújo, A.M.3
1Rua Ipiniras, 103. Cordeiro. Recife-PE. Brasil. eulalia.barros@bol.com.br
2Universidade Federal Rural de Pernambuco. UFRP. Departamento de Zootecnia. Recife-PE. Brasil, *maria.ribeiro@pesquisador.cnpq.br
3Embrapa Meio Norte-Piauí. Brasil
#Parte da dissertação do primeiro autor. Projeto financiado pelo CNPq.
RESUMO
A raça caprina Marota é parte do patrimônio genético do Brasil, formada por animais altamente adaptados ao semi-árido nordestino. Este estudo avaliou a estrutura genética do núcleo de conservação da raça Marota, mantida pela Embrapa Meio Norte. Foram estimados os parâmetros populacionais dos dados genealógicos de 663 animais nascidos entre os anos de 1995 a 2003. O coeficiente médio de parentesco (AR) e de consanguinidade (F) para a população foi de 0,11% e de 0,84%, respectivamente. O intervalo de gerações (IEG) foi de 5,28 anos e o tamanho efetivo médio (Ne) por geração foi de 222 animais, sendo que o número efetivo de animais fundadores (ƒe) e de ancestrais (ƒa) foi igual (48). Dentre os 214 ancestrais, apenas 22 foram responsáveis por 50% da variabilidade genética da população, o que indica perda de genes de origem. Observase baixa contribuição dos animais fundadores ao longo das gerações. Os valores do coeficiente de endogamia de Wright indicam subdivisão da população em linhagens. Em geral, a consangüinidade e os valores médios do coeficiente de parentesco são baixos para o rebanho avaliado.
Palavras chave: Conservação. Consanguinidade. Parentesco. Número efetivo.
SUMMARY
Marota breed goat is part of the Brazilian genetic patrimony, consisting by animals highly adapted to semi-arid region. This study evaluated genetic structure of Marota breed from Embrapa Meio Norte conservation nucleus. Pedigree records of 663 animals, which were born from 1995 to 2003, were used for population parameters estimation. Inbreeding coefficient (F) and average relationship coefficient (AR) of the population were 0.11% and 0.84% respectively. Generation interval (IEG) was 5.28 years and average effective size (Ne) per generation was 222 animals; being the effective number of founder animals (ƒe) and ancestral (ƒa) was the same (48). Among 214 ancestors evaluated, just 22 of them were responsible for 50% of the population genetic variability, which indicate loss of original genes. This study shows low contribution of the founder animals among the generations. Wright inbreeding coefficient indicates population subdivision in lineages. Inbreeding and average relationship coefficient (AR) are low in the evaluated herd.
Key words: Conservation. Inbreeding. Coancestry. Effective size.
Introdução
Todos os dias um número incerto de raças e variedades animal e vegetal desaparece ao ser trocado ou absorvido por outras com maiores taxas de produção. No intuito de procurar inverter esta tendência, se faz necessário perceber que os recursos genéticos de um país, ou região, constituem um patrimônio cultural e biológico único.
Nos países subdesenvolvidos essa situação é ainda mais séria, portanto, a adoção de medidas com objetivo de conservar seus recursos genéticos se faz necessário, já que os programas existentes ainda são incipientes. Assim como ocorreu nos países desenvolvidos, identificar os valores diferenciais e desenvolver raças nacionais através da seleção intra-rebanho é um dos maiores desafios para o Brasil.
Os primeiros caprinos que chegaram ao Brasil foram trazidos pelos colonizadores portugueses. Criados à margem de práticas zootécnicas, sem seleção direcionada para a produção, os caprinos de origem ibérica, se multiplicaram desordenadamente, passaram por um processo de seleção natural secular e deram origem aos vários tipos étnicos encontrados atualmente.
Nos últimos anos, devido a pouca valorização das raças nativas, vem ocorrendo uma mudança rápida na composição racial da população caprina brasileira, conseqüência da expressiva Introdução de raças exóticas, utilizadas intensivamente em cruzamentos (Figueiredo, 1988).
Atualmente, não se sabe o tamanho efetivo das populações de caprinos das raças nativas do nordeste, havendo zonas onde não existem mais populações desses animais em seu estado de pureza. Existem atualmente no semi-árido nordestino apenas alguns rebanhos de raças nativas, frutos de alguns criadores e empresas estaduais de pesquisa (Ribeiro e Pimenta Filho, 2003).
A raça Marota, também denominada Curaçá, é descendente dos tipos raciais trazidos pelos colonizadores, como todas as demais raças nativas de caprinos aqui existentes, apesar de não se ter ainda estudos que comprovem essa origem. Apesar de apresentar porte reduzido em relação às raças exóticas, os animais da raça Marota apresentam características raciais bem definidas e, pela sua própria origem, é bem adaptada a ambientes de baixa oferta de alimentos, além de serem dotados de grande rusticidade. Por falta de um conhecimento mais profundo sobre sua importância zootécnica e até mesmo histórico-cultural, essa raça está desde muitos anos seriamente ameaçada de extinção (Domingues, 1955; Araújo, 1979).
Alguns fatores podem promover a fragmentação de uma população, como o isolamento geográfico ou o uso de determinados animais de maneira mais intensa, tais fatores atuam na fixação ou até mesmo na perda de certos alelos junto a essas subpopulações. Mesmo havendo acasalamentos ao acaso dentro das sub-populações ocorrerá o favorecimento de determinados genótipos (Laat, 2002).
Este trabalho teve por objetivo avaliar o nível de ameaça e de conservação da variabilidade genética da raça Marota, através da monitoração do rebanho através de um conjunto de parâmetros para caracterizar a estrutura populacional (indivíduo e rebanho), visando com isso, fornecer informações para futuras melhorias nas práticas de gestão dessa população.
Material e métodos
Os dados utilizados no presente trabalho referem-se a genealogia de animais do campo experimental da Embrapa Meio-Norte no período de 1995 a 2003, totalizando 663 registros de nascimentos. Os dados genealógicos foram analisados utilizando-se o programa ENDOG (versão 4.5) (Gutiérrez y Goyache, 2005). Para caracterização da estrutura e diversidade genética da raça Marota, foram analisados os seguintes parâmetros:
1. Intervalo de gerações: idade média dos pais ao nascimento dos filhos. Pode-se obter para as quatro vias possíveis (paifilho, pai-filha, mãe-filho e mãe-filha). Foi calculado de acordo com Hill (1979), através da fórmula:


2. Número de gerações completas: geração mais distante em que todos os ancestrais são conhecidos.
3. Número de gerações equivalentes: somatório dos termos (1/2)n de todos os ancestrais conhecidos, em que n é o número de gerações que separa o indivíduo de cada ancestral conhecido.
4. Número efetivo de animais fundadores (ƒe): representa o número de animais com igual contribuição que produziria a mesma variabilidade genética encontrada na população estudada (Lacy, 1989). Obtido por:

5. Número efetivo de ancestrais (ƒa): representa o número mínimo de animais necessários para explicar a diversidade genética da população estudada (Boichard et al., 1997). Calculado de maneira similar ao ƒe:


6. Coeficiente de consanguinidade individual (F) (Wright, 1931): probabilidade de dois indivíduos apresentarem cópia do mesmo alelo por conta de um ascendente em comum. Foi calculado para todo e pedigree e para as diferentes gerações utilizando-se o algoritmo proposto por Meuwissen e Luo (1992). O aumento da consanguinidade (.FΔ) foi calculado para cada geração através da clássica fórmula:

7. Número efetivo da população (Ne) (Wright, 1931): número de indivíduos que acasalando ao acaso e com a mesma chance de deixarem filhos gerassem a mesma taxa de consanguinidade observada na população em estudo. Obtido por:

8. Coeficiente de parentesco médio (AR) (Goyache et al., 2003): é a dupla probabilidade de dois alelos tomados ao acaso, um para o animal e outro para a população no pedigree (incluindo o do animal), serem idênticos por descendência. O AR de cada indivíduo no pedigree é calculado como a média dos coeficientes na linha correspondente ao indivíduo em relação a matriz de parentesco A.
9. Índice de fixação ou estatísticas F (Wright, 1978): teoria que parte do princípio da existência de uma metapopulação constituída de várias subpopulações que podem ser definidas em função dos diferentes sexos, áreas geográficas, fazendas, linhagens, famílias, etc. As estatísticasF, FIS, FST e FIT, são definidas respectivamente, redução de heterozigosidade entre subpopulações, redução da heterozigosidade dentro das subpopulações e redução da heterozigosidade no total da população. As estatísticas F são calculadas de acordo com Cabellero e Toro (2002):

Resultados e discussão
As estimativas dos intervalos de gerações (IEG) para as quatro passagens gaméticas são apresentadas na Tabela I. Observa-se que em todas as passagens os valores foram muito próximos, não havendo diferenciação entre o IEG de machos e fêmeas, isso significa que a reposição de reprodutores e matrizes está sendo feita ao mesmo tempo no rebanho. Em rebanhos alvo de programas de conservação essa medida é adequada, pois permite o uso igualitário entre os dois sexos e quanto maior melhor, contanto que haja controle da consanguinidade.
O IEG médio para caprinos é de 2,3 anos (Léon et al., 2005), no entanto, em rebanhos que tem instalados programas de melhoramento, esse valor tende a ser menor, principalmente pela via paterna. Tal fato ocorre devido à breve permanência dos animais dentro do rebanho somado a entrada precoce desses animais na vida reprodutiva, esses dois fatores promovem o intervalo entre gerações menores, em rebanhos que adotam esse tipo de manejo.
O IEG médio obtido foi de 5,28 anos, esse resultado já era esperado, pois como os animais estudados pertencem a um núcleo de conservação, os intervalos de gerações tendem a ser mais elevados. Isso ocorre porque os animais são mantidos no rebanho pelo maior tempo possível, principalmente aqueles considerados fundadores, como forma de garantir sua maior participação na constituição genética da população. No entanto, tal medida precisa estar associada a um bom manejo reprodutivo, através do controle dos acasalamentos, evitando assim aumentos da consanguinidade. Lima et al. (2007) em estudos com caprinos nativos no Brasil, relataram IEG médio de 3,01 anos, valor bem abaixo do encontrado no presente trabalho.
Na tabela II, é apresentado o resumo da estrutura populacional do rebanho estudado. Foram determinados a partir da população base, aqueles responsáveis pela variabilidade genética encontrada no rebanho, ou seja, o número de animais fundadores e ancestrais.
O número efetivo de fundadores (ƒe) apontou um valor bem abaixo do número total de animais pertencentes à população fundadora. Esse resultado já era esperado, uma vez que, de acordo com a análise prévia do pedigree, verificou-se que poucos foram os animais usados como reprodutores ao longo dos anos, em relação ao rebanho base.
O número efetivo de ancestrais (ƒa) encontrado foi igual ao ƒe (48). Quando esse tipo situação ocorre, significa que os animais que contribuíram para formação da raça continuam atuando de maneira efetiva no rebanho atual, ou seja, não houve nenhum animal, além dos considerados fundadores, que tenha contribuído de forma efetiva para a composição genética do rebanho. Porém o ideal é que esses números efetivos sejam os mais próximos possíveis da população fundadora, o que não ocorreu neste estudo. Observaram-se baixos valores de ƒe e ƒa, quando comparados com os números das populações base e referência, fazendo com que a o rebanho se desenvolvesse a partir de estreita base genética, levando a um gargalo genético com perdas de genes de origem.
A contribuição dos ancestrais para a formação da população referência pode ser vista na figura 1. Observa-se que um número pequeno de ancestrais explica grande parte da variabilidade genética da população estudada.
Do total de animais pertencentes à população base apenas 22 explicam 50% da variabilidade total encontrada no rebanho, indicativo do uso desequilibrado de alguns reprodutores. Essa situação é reflexo dosbaixos valores do ƒe e ƒa encontrados, o ideal seria que todos os animais contribuíssem de igual maneira ao longo das gerações.
Valera et al. (2005) estudando a raça de cavalo Andaluz, constataram que apenas 6 animais concentraram 50% do total de variabilidade da população. Já Goyache et al. (2003) avaliando a raça ovina Xalda atribuíram essa mesma porcentagem de variabilidade a 13 animais. Ficando claro também nesses estudos, as contribuições desequilibradas entre os fundadores que lhes deram origem.
O desequilíbrio entre as contribuições dos ancestrais e fundadores pode ser verificado ao analisar-se o número de descendentes deixados por esses animais.
Na tabela III encontram-se os quatro ancestrais e fundadores que mais contribuíram para variabilidade genética da população. Todos esses animais são nascidos no ano em que se iniciaram os registros e, nesse mesmo ano, a relação macho: fêmea encontrava-se em desequilíbrio. Poucos machos foram usados na reprodução de maneira mais intensa, fato que justifica a presença marcante de machos como os principais contribuintes da população.
O animal que mais contribuiu para o rebanho deixou 53 descendentes. No entanto, esse foi um caso isolado, já que a maior parte dos ancestrais (94%) contribuiu com menos de 10 descendentes. Dos quatro principais animais listados como ancestrais três também foram classificados como animais fundadores. Assim sendo, todos os animais classificados como fundadores também foram considerados ancestrais já que seus respectivos números efetivos foram iguais. Situação mais crítica foi encontrada por Royo et al. (2007) em pôneis da raça Astucón, onde o principal ancestral foi responsável por 22,9% da variabilidade genética total encontrada, indicando o uso intenso de apenas um animal.
Mesmo com o uso intenso de determinados reprodutores, os valores médios de consanguinidade (F) e de parentesco médio (AR) encontrados no rebanho Marota ao longo dos anos foram baixos. Vale ressaltar que a quase inexistência de consanguinidade individual não se deve apenas ao fato de não haver animais consanguíneos dentro do rebanho, mas também, pela dificuldade de estimar este parâmetro de maneira precisa, uma vez que, durante os primeiros anos o volume de informação das relações de parentesco existentes dentro do rebanho foi escasso.
Como pode ser visto na figura 2, a elevação de F no ano de 2001 coincide com o surgimento dos poucos animais endogâmicos na população, porém, não ultrapassa 0,5%, pois a partir desse momento os valores voltam a diminuir.
Quanto ao valor médio de AR, o mesmo segue a tendência da consanguinidade. A estreita relação existente entre os dois parâmetros fica clara na figura 2.
Os coeficientes de parentesco médio individuais (AR) não ultrapassaram o valor de 0,043, valor esse, referente ao animal que mais contribuiu para o rebanho. Esses baixos valores podem ser usados como ferramenta para prever e controlar possíveis incrementos da consanguinidade a longo prazo.
Os baixos valores de AR tanto individual quanto populacional demonstram que o rebanho estudado encontra-se em boa situação genética, aumentando as chances de reprodução entre indivíduos não aparentados ou pouco aparentados. Portanto, o conhecimento do AR dos reprodutores é essencial para que se faça a escolha mais adequada dos animais a serem usados na reprodução. A forma mais eficiente para o controle da consanguinidade a longo prazo é dar preferência sempre ao uso de reprodutores com baixos valores de AR. Todos os trabalhos sobre raças em situação de risco apontam para a necessidade da maior utilização de animais fundadores de baixo AR para reprodução, a exemplo de Valera et al.(2005) e Gutiérrez et al. (2005).
Valores de FeAR mais elevados foram relatados por Goyache et al. (2003) com ovinos da raça Xalda, com valores de F e AR de 1,54 e 1,79%, respectivamente. Já Lima et al. (2007) em estudo com as raças caprinas nativas, Canindé, Azul e Moxotó, apresentaram valores de F de 6%, 7% e 5%, respectivamente. Na Tabela IV, encontramse os valores médios de consanguinidade (F) e do coeficiente médio de parentesco (AR) de acordo com as gerações estudadas. Observa-se aumento de F e AR ao longo das gerações. O número de animais por geração é bem irregular. A distribuição dos animais pertencentes do total de 663 animais 39, 56 e 4,7% foram classificados para as gerações 0, 1 e 2, respectivamente.
Na a primeira geração, não foi registrado consanguinidade e tal fato ocorre porque essa geração é composta pelos primeiros filhos dos animais fundadores. Porém, nos animais da segunda geração observa-se presença de consanguinidade, mesmo que em níveis baixos. Quanto menor o número de animais e mais avançada a geração, maior será a probabilidade de haver incrementos de F e de AR, notadamente em rebanhos pequenos, como é o caso, já que em fases mais avançadas, já se tem controle dos pedigrees o que permite a estimativa da consanguinidade, o que não é possível para os animais fundadores, isto é, aqueles animais sem pedigrees conhecidos.
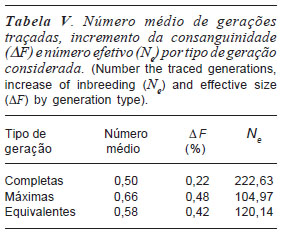
O incremento da consaguinidade e o número efetivo calculados para os diferentes tipos de gerações traçadas encontramse na tabela V. Os valores de ΔF encontramse baixos para as diferentes gerações. Isto é conseqüência do alto número efetivo, já que este parâmetro é inversamente proporcional a consanguinidade.
Verifica-se que, quanto menor o número de ancestrais conhecidos, menor a probabilidade de ser detectado aumento de consanguinidade, exatamente pela ausência das informações de parentesco. Tal fato justifica o menor valor de ΔF encontrado nas gerações completas. Por outro lado, quanto maior for o número de ancestrais conhecidos, maior será a probabilidade de se obter elevadas taxas de consangüinidade. Por conta disso, nas gerações máximas foram encontrados valores mais elevados de ΔF. Para as gerações equivalentes, os valores encontrados foram intermediários, uma vez que seu cálculo considera todos os ancestrais conhecidos e não somente os conhecidos ou mais remotos.
Os valores de Ne oscilaram concomitantemente as oscilações ocorridas nas taxas de consanguinidade, já que este parâmetro é calculado em função de .F e, ambos são inversamente proporcionais.
Vale ressaltar que em populações onde informações de pedigree são escassas, essas diferentes estimativas podem ser úteis para indicar os limites superior, inferior, e reais do Ne na população analisada (Gutiérrez e Goyache, 2005).
A situação do rebanho quanto à possível ocorrência de subdivisão foi avaliada e os valores obtidos para os coeficientes de consanguinidade dentro das subpopulações (FIS) e da população (FIT) foram de -0,22 e de -0,008, respectivamente. Os valores negativos e próximos a zero indicam que os níveis de variabilidade genética do rebanho é alta, já que tais valores indicam um excesso de animais heterozigotos, tanto dentro das linhagens paternas (subpopulações) quanto para a população como um todo (metapopulação). Esse resultado está em desacordo a estreita base genética encontrada no rebanho, que deveria levar a altos níveis de consangüinidade. Uma explicação para isso pode ser dada pela possível Introdução de animais de fora no rebanho.
Para o índice FST o valor calculado foi de 0,178, demonstrando que há diferenciação entre as linhagens paternas com formação de subpopulações, o que era esperado já que parte 50% da diversidade genética do rebanho foi determinada pela contribuição de poucos animais conforme observa-se na figura 1. Entretanto, 82,7% da variabilidade genética total do rebanho correspondem as diferenças entre os indivíduos.
Conclusão
O uso de alguns animais de forma mais intensa contribuiu para a baixa representação dos animais fundadores no pedigree do rebanho ao longo das gerações.
O efeito de gargalo genético é observado devido ao baixo número de animais ancestrais que contribuíram para fundação do rebanho estudado.
Os valores das estatísticas F de Wright indicam a existência de subdivisão da população promovido pelo uso excessivo de algumas linhagens paternas.
Os baixos valores de consangüinidade e parentesco médio podem ser úteis para o estabelecimento de um plano de gestão genética para o rebanho.
Bibliografia
Araújo, A,B. 1979. À margem da caprinocultura cearense. Pecuária. 89: 21-22. [ Links ]
Boichard, D., Maignel, L. and Verrier, E. 1997. The value of using probabilities of gene origin to measure genetic variability in a population. Genet. Sel. Evol., 29: 5-23. [ Links ]
Caballero, A. and Toro, M.A. 2002. Analysis of genetic diversity for the management of conserved subdivided populations. Conserv. Genet., 3: 289-299. [ Links ]
Domingues, O. 1955. A cabra na paisagem do Nordeste. Secção de fomento agrícola do Ceará. Fortaleza. 55 pp. [ Links ]
Figueiredo, E.H. 1987. Recursos genéticos e programas de melhoramento na espécie caprina no Brasil. In: 7oCongresso Brasileiro de Reprodução Animal. Belo Horizonte. Brasil. [ Links ]
Goyache, F., Gutierrez, J.P., Fernandez, I., Gomez, E., Alvarez, I., Diez, J. and Royo, L.J. 2003. Using pedigree information to monitor genetic variability of endangered populations: the Xalda sheep breed of Asturias as an example. J. Anim. Breed. Genet., 120: 95-103. [ Links ]
Gutierrez, J.P., Marmi, J., Goyache, F. and Jordana, J. 2005. Pedigree information reveals moderate to high levels of inbreeding and a weak population structure in the endangered Catalonian Donkey breed. J. Anim. Breed. Genet., 122: 378-386. [ Links ]
Gutierrez, J.G. and Goyache, F. 2005. A computer program for analysing pedigree information. ENDOG. J. Anim. Breed. Genet., 122: 172-176. [ Links ]
Hill, W.G. 1979. A note on effective population size with overlapping generations. Genetics, 92: 317-322. [ Links ]
Laat, D.M. 2001. Contribuição genética dos fundadores e ancestrais na raça Campolina. (Dissertação Mestrado). Universidade Federal de Minas Gerais. [ Links ]
Lacy, R.C. 1989. Analysis of founder re-presentation in pedigrees: founder equivalents and founder genome equivalents. Zoo Biol., 8: 111-123. [ Links ]
Lima, P.J.S., Souza, D. L., Pereira, G.F. e Torreao, J.N.C. 2007. Gestão genética de raças caprinas no estado da Paraíba. Arch. Zootec., 56: 623626. [ Links ]
Meuwissen, T.H.E. and Luo, Z. 1992. Computing inbreeding coefficients in large populations. Genet. Sel. Evol., 24: 305-313. [ Links ]
Ribeiro, M.N. y Pimenta Filho, E.C. 2003. Impacto de la Introducción de razas europeas a Brasil sobre la producción caprina. In: Reunion Nacional sobre Caprino Cultura. Benemérita. Universidad Autónoma de Puebla. pp. 215-223. [ Links ]
Royo, L.J., Álvarez, I., Gutiérrez, J. P., Fernández, I. and Goyache, F. 2007. Genetic variability in the endangered Asturcón pony assessed using genealogical and molecular information. Livest. Sci., 107: 162-169. [ Links ]
Valera, M., Molina, A., Gutiérrez, J.P., Gómez, J. and Goyache, F. 2005. Pedigree analysis in the Andalusian horse: population structure, genetic variability and influence of the Carthusian strain. Livest. Prod. Sci., 95: 57-66. [ Links ]
Wright, S. 1978. Evolution and the genetics of populations: variability within and among natural populations. University of Chicago. Chicago. [ Links ]
Wright, S. 1931. Evolution in Mendelian populations. Genetics, 16: 97-159. [ Links ]
Recibido: 4-3-09
Aceptado: 26-11-09