Mi SciELO
Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO -
 Accesos
Accesos
Links relacionados
-
 Citado por Google
Citado por Google -
 Similares en
SciELO
Similares en
SciELO -
 Similares en Google
Similares en Google
Compartir
Nefrología (Madrid)
versión On-line ISSN 1989-2284versión impresa ISSN 0211-6995
Nefrología (Madr.) vol.31 no.1 Cantabria 2011
Diagnóstico molecular de la poliquistosis renal autosómica dominante
Molecular diagnosis of autosomal dominant polycystic kidney disease
R. Torra Balcells1, E. Ars Criach2
1 Servicio de Nefrología. Enfermedades Renales Hereditarias. Fundació Puigvert. Departament de Medicina. Universitat Autònoma de Barcelona.
REDinREN. Instituto de Investigación Carlos III. Barcelona.
2 Laboratorio Biología Molecular. Fundació Puigvert. Departament de Medicina. Universitat Autònoma de Barcelona.
REDinREN. Instituto de Investigación Carlos III. Barcelona
Dirección para correspondencia
RESUMEN
La poliquistosis renal autosómica dominante (PQRAD) es la enfermedad renal hereditaria más frecuente. Su prevalencia estimada es de 1 cada 800 personas. Los pacientes con PQRAD constituyen un 8% aproximadamente de la población en diálisis o trasplante renal. El diagnóstico de la enfermedad es radiológico y/o genético. La posibilidad de realizar un diagnóstico genético directo de la PQRAD es actualmente una realidad en nuestro país, aunque por las características del gen PKD1 no es un análisis sencillo ni económico. Debe estudiarse cada caso de forma individualizada con el fin de determinar la idoneidad de realizar un estudio genético y determinar qué tipo de estudio es el adecuado. El diagnóstico genético es de especial interés para los donantes vivos, para casos neonatales y para casos esporádicos. El diagnóstico genético permite ofrecer diagnóstico prenatal o preimplantacional en familias con casos severos de la enfermedad y también permitirá tratar la enfermedad, cuando exista un tratamiento específico, en aquellos casos dudosos que sin confirmación genética no serían candidatos a tratamiento.
Palabras clave: PQRAD, Poliquistosis renal autosómica dominante, Diagnóstico genético, PKD1, PKD2, Mutaciones.
ABSTRACT
Autosomal dominant polycystic kidney disease (ADPKD) is the commonest renal inherited disorder. Its estimated prevalence is 1 in 800 individuals. ADPKD patients constitute 8% of the population on dialysis or renal transplantation. The diagnosis of the disease can be made using radiological or genetic procedures. In Spain, we are now able to perform a direct genetic diagnosis of the disease, however it is neither an easy test nor cheap. This is why every case should be considered by means of an individualized approach to determine the appropriateness of genetic testing and to determine which genetic test is more adequate. Genetic testing in ADPKD is especially interesting for living donors, neonatal and sporadic cases. Genetic testing offers the chance to perform prenatal or preimplantation testing of embryos in families with severe disease. Also, the guarantee of a doubtless diagnosis will permit to treat sporadic cases, when treatment becomes available.
Key words: ADPKD, Autosomal dominant polycystic kidney disease, Genetic diagnosis, PKD1, PKD2, Mutations.
Introducción
Clínica y epidemiología
La poliquistosis renal autosómica dominante (PQRAD) es la enfermedad renal hereditaria más frecuente. Su prevalencia estimada es de uno cada 800 personas. Los pacientes con PQRAD constituyen un 8% aproximadamente de la población en diálisis o trasplante renal. Se caracteriza por la progresiva aparición de quistes renales que suele conducir a insuficiencia renal crónica terminal (IRCT), generalmente en la edad adulta. La mayoría de pacientes suelen tener quistes hepáticos pero sólo una minoría desarrolla una enfermedad poliquística hepática masiva. También es poco frecuente la presencia de aneurismas cerebrales en estos pacientes (10% aproximadamente). Se ha descrito una agrupación familiar de aneurismas cerebrales1,2, por lo que actualmente se recomienda realizar un angiorresonancia craneal cuando algún miembro afectado de la familia ha presentado aneurismas cerebrales o un episodio compatible con ruptura de los mismos. Existe una notable variabilidad clínica tanto interfamiliar como intrafamiliar, siendo mucho mayor la primera de ellas3. El espectro fenotípico de la enfermedad oscila entrecasos severos intraúteroy pacientes ancianos con función renal normal.
Bases genéticas
Es bien conocido que la PQRAD es una enfermedad de causa genética. Presenta heterogeneidad genética, es decir, hay más de un gen causante de la enfermedad. Los genes responsables son: PKD1 (localizado en el cromosoma 16: 16p13.3)4 y PKD2 (localizado en el cromosoma 4: 4p21)5. Mutaciones en el gen PKD1 dan lugar al 85% de los casos de PQRAD, mientras que mutaciones en el gen PKD2 dan lugar al 15% restante6. La severidad de la enfermedad es superior en los casos causados por mutaciones en PKD1;la edad media de inicio de diálisis es de 54,3 años para las personas conPKD1 y de 74 para las personas con PKD27. Para una misma edad, el tamaño renal es significativamente superior si el gen causante es PKD1 que si es PKD2, lo cual, según los resultados del estudio CRISP, justifica plenamente la distinta evolución de la enfermedad8. De todas maneras existe una notable variabilidad respecto a la edad de inicio de diálisis para cada uno de los genes9.
Gen PKD1
El gen PKD1 está constituido por 46 exones y abarca una región genómica de 54 kb. Se transcribe en un ARNm de aproximadamente 14 kb con una secuencia codificante de 12.909 pb (figura 1A)10,11. El análisis de este gen resulta muy complejo puesto que la región contenida entre los exones 1 y 33 ha sufrido una duplicación intracromosómica a lo largo de la evolución humana de manera que contiene 6 seudogenes (PKD1P1-P6), localizados entre 13 y 16 Mb proximalmente al gen PKD1. Estos seudogenes se expresan pero tienen codones de parada de la traducción, por lo que se prevé que darán lugar a proteínas de tamaño reducido no funcionales. Comparando las secuencias de estos seudogenes con PKD1 podemos ver que los seudogenes contienen mutaciones pero que presentan una identidad de secuencia del 98-99% en las regiones homólogas a PKD1. Las mínimas diferencias entre las secuencias de estos seudogenes y PKD1 han resultado claves para el diseño de estrategias que permiten la amplificación selectiva de PKD1 evitando la amplificación de estos seudogenes12-14.
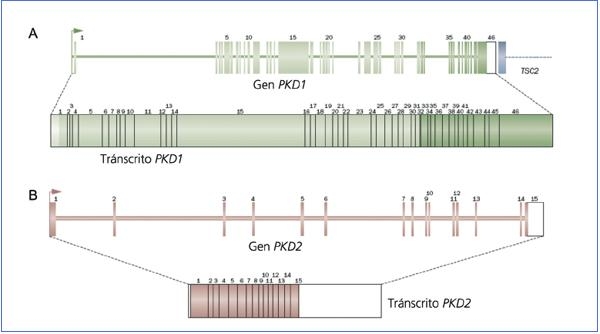
Adaptada de Harris PC y Rossetti. Nat Rew 2010.
Figura 1. Estructura de los genes PKD1 (A) y PKD2 (B) y sus tránscritos.
El gen PKD1 codifica para una proteína integral de membrana denominada poliquistina 1.
Gen PKD2
El gen PKD2 contiene 15 exones y abarca una región genómica de 68 kb con una secuencia codificante de 2.904 pb5,15. Este gen codifica para la proteína poliquistina 2 que es un canal de calcio de la familia de los TRP (transient receptor potencial) por lo que también se denomina TRPP2 (figura 1B).
Ambas poliquistinas se localizan, entre otros emplazamientos, en los cilios primarios. Por ello, al igual que todas las enfermedades quísticas renales, se considera que la PQRAD es una ciliopatía. Parece que las poliquistinas tienen una función relacionada con la detección de flujo16, presión17, modulación de la duplicación del centrosoma y/o regulación del ciclo celular18,19. También se han implicado en la preservación de la polaridad planar20. Todos estos mecanismos pueden estar implicados en la quistogénesis, pero el mecanismo clave y preciso aún no queda claro.
Diagnóstico de la poliquistosis renal autosómica dominante
Dado que la PQRAD es una enfermedad autosómica dominante con una elevada penetrancia, los descendientes de un progenitor afectado tienen una probabilidad del 50% de presentar la enfermedad. Y dada también esta elevada penetrancia sería una rareza que la enfermedad saltara, clínicamente, una generación. Actualmente, la enfermedad se suele diagnosticar al haber un familiar afectado y realizar una ecografía para descartar/confirmarla. El diagnóstico de la enfermedad es en la actualidad radiológico y/o genético.
Diagnóstico radiológico
La ecografía es la técnica más utilizada para el diagnóstico de la enfermedad en los sujetos con riesgo de presentarla. Los criterios clásicos para el diagnóstico de PKD1 son los descritos por Ravine et al. en 199321 y más recientemente se han publicado los criterios ecográficos para PQRAD en ausencia de conocimiento del gen causante de la enfermedad22.
En un 10% de los casos no hay antecedentes familiares de la enfermedad23,24. En estos casos, la enfermedad se diagnostica de forma casual, pero a menudo es difícil precisar si es realmente una PQRAD. La población general desarrolla de forma habitual quistes renales, de forma progresiva con la edad, de manera que el diagnóstico se hace más complejo en casos sin antecedentes familiares en individuos adultos o de edad avanzada. A pesar de la relativa precisión de los criterios ecográficos éstos resultan poco sensibles y poco específicos en jóvenes y en individuos de edad avanzada con una forma leve de la enfermedad.
La TAC y la RNM son técnicas con un coste económico superior que la ecografía, pero son más sensibles y permiten ver quistes de menor tamaño (2 mm en comparación con 10 mm en la ecografía). No existen criterios radiológicos para el diagnóstico de PQRAD por TAC o RNM.
Las dificultades diagnósticas por medios radiológicos en determinados casos de PQRAD, como jóvenes y pacientes sin antecedentes familiares, han hecho necesario el desarrollo del diagnóstico genético.
Diagnóstico genético
Análisis de ligamiento
La localización de los genes PKD1 y PKD2 permitió ya en la década de los noventa realizar el diagnóstico genético de la PQRAD mediante análisis de ligamiento. Este estudio es indirecto y se basa en el análisis de marcadores genéticos localizados en la región del gen PKD1 y del gen PKD2 (figura 1). Permite determinar si es el haplotipo PKD1 o el PKD2 el que segrega con la enfermedad en una determinada familia y, en consecuencia, cuál es el gen responsable de la enfermedad en dicha familia. Actualmente se dispone de las bases de datos en las que se localizan un mínimo de 15 marcadores tipo microsatélite útiles para el ligamiento a PKD1 y de 8 marcadores para PKD2. El gran inconveniente de este tipo de diagnóstico es que sólo se puede aplicar a los casos familiares y, debido a la heterogeneidad genética de la enfermedad, además del consultante debemos disponer de varios familiares afectados y varios no afectados, a los que se les haya hecho un estudio radiológico para conocer con exactitud su estado respecto a la enfermedad. Además es imperativo que alguno de los miembros de la familia tenga un diagnóstico clínico 100% certero de PQRAD (a este familiar se le denomina probando o caso índice). Sólo determinadas familias son lo suficientemente amplias como para que el estudio confirme el ligamiento a uno de los genes y descarte el ligamiento al otro gen. En algunos casos todos los familiares afectados comparten tanto el haplotipo PKD1 como el PKD2 y ninguno de los familiares no afectados son portadores de éstos, por lo que el estudio no es informativo (no permite definir si es PKD1 o PKD2 el responsable de la enfermedad) (figura 2). Pero no sólo el tamaño de la familia o la informatividad de los marcadores utilizados complican esta aproximación diagnóstica sino que hay otros fenómenos como: la presencia de mutaciones de novo, el mosaicismo, la presencia de alelos hipomórficos, etc. Todo esto indica que se debe ser muy cauto con este tipo de aproximación diagnóstica.

Figura 2. Análisis de ligamiento para PKD1 y PKD2.
Análisis mutacional
A pesar de existir distintas posibles aproximaciones para el estudio mutacional de los genes PKD1 y PKD2, la secuenciación de todos sus exones es la técnica más empleada en la actualidad. Existen muchos estudios que han demostrado la gran heterogeneidad alélica de estos genes, de manera que una misma mutación no se encuentra en más del 2% de familias. Este hecho dificulta enormemente la búsqueda de mutaciones por lo que se deben analizar de forma sistemática todos los exones del gen PKD1 y PKD2, lo cual, dado el tamaño y complejidad de PKD1, es relativamente costoso. Para analizar la región de PKD1 homóloga a los seudogenes se han diseñado los cebadores de la reacción de amplificación (PCR, polymerase chain reaction) en las secuencias en las que PKD1 difiere de estos seudogenes. Así, inicialmente se amplifica esta región genómica que incluye los exones del 1 al 33 en 5 PCR largas y a continuación, se utilizan estos productos iniciales como molde en la subsiguiente amplificación de estos 33 exones13. Los 13 exones de PKD1 restantes y los 15 exones de PKD2 se pueden amplificar de forma convencional a partir del ADN genómico del paciente. Una vez secuenciados los 46 exones de PKD1 y/o los 15 de PKD2 es muy importante realizar una correcta evaluación de la posible patogenicidad de las distintas variantes de secuencia identificadas.
Para ello una herramienta de gran utilidad es la base de datos desarrollada y mantenida por la Mayo Clinic (http://pkdb.mayo.edu/cgi-bin/mutations.cgi) que incluye todas las variantes de secuencia descritas en estos genes. En la tabla 1 podemos ver los tipos de mutaciones que causan la PQRAD. Tal como puede observarse en la figura 3A existen mutaciones claramente patogénicas, mientras que un porcentaje considerable de variantes de secuencia identificadas en estos genes son probablemente neutras. Las variantes que se prevé darán lugar a una proteína truncada (de tamaño inferior a la proteína salvaje) y las que afectan a las secuencias canónicas de splicing 100% conservadas, generalmente se consideran claramente patogénicas y no requieren ningún estudio adicional. En cambio, las variantes en pauta (in frame) que no interrumpen el marco de traducción de la proteína (variantes de cambio de aminoácido, deleciones/inserciones de número de bases múltiplo de 3, mutaciones en regiones no codificantes) requieren una evaluación más profunda. Si a este hecho le añadimos que cada paciente tiene una media de 10 variantes neutras en el gen PKD1 podemos empezar a vislumbrar la gran dificultad diagnóstica de la enfermedad en muchos casos. Existen herramientas bioinformáticas que se utilizan para clasificar estas variantes de secuencia y determinar así su patogenicidad12,25-28. Hasta el momento en la base de datos constan 436 mutaciones diferentes para PKD1 y 115 para PKD2. En la figura 2B podemos ver los distintos tipos de variantes de secuencia identificadas tanto para PKD1 como para PKD2. A veces de trata de grandes deleciones que eliminan hasta 10 genes próximos a la región 5' sin consecuencias fenotípicas aparte de la PQRAD. Por otra parte, en la región 3' pueden producirse deleciones que abarcan también el gen TSC2 (causante de uno de los dos tipos de esclerosis tuberosa) y dan lugar a un síndrome de genes contiguos que clínicamente se traduce en poliquistosis renal de presentación precoz y esclerosis tuberosa. Como se puede observar el porcentaje de cambios de secuencia con alta probabilidad de ser patogénicos es muy superior en PKD2 que en PKD1 (figura 2 A). Asimismo, el porcentaje de mutaciones tipo missense (cambio de sentido) es muy superior en PKD1, lo cual dificulta el diagnóstico al tener que demostrar la patogenicidad de las mismas (figura 2 B).
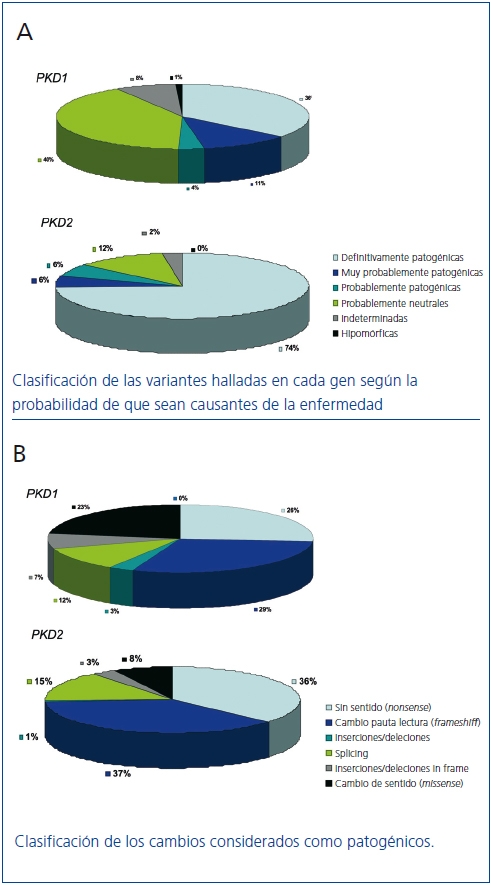
Figura 3. Clasificación de las variantes de secuencia halladas en los genes PKD1 y
PKD2 según la base de datos de mutaciones en PKD1 y PKD2
(http://pkdb.mayo.edu/cgi-bin/mutations.cgi).
En total, se pueden llegar a identificar variantes de secuencia con una elevada probabilidad de ser mutaciones patogénicas en un 91% de familias. De éstas, un 65% son mutaciones que truncan la proteína por lo que pueden ser directamente utilizadas para el diagnóstico pero el 26% son variantes in frame que requieren un cauteloso análisis antes de su aplicación clínica29. La segregación de estas variantes in frame en una determinada familia es de gran ayuda de cara a establecer de forma definitiva su patogenicidad. También es muy importante tener en cuenta si alteran aminoácidos altamente conservados en las proteínas homólogas. Asimismo, la inclusión de estas mutaciones en bases de datos permitirá una mayor precisión en la determinación de su patogenicidad cuando se detecten de nuevo las mismas mutaciones en otras familias.
Para otros genes en lugar o además de herramientas bioinformáticas se pueden realizar estudios funcionales para determinar la repercusión funcional de una variante de secuencia y, por lo tanto, su patogenicidad. Aunque teóricamente es factible para PKD2 y para algunas mutaciones en PKD1 se trata de técnicas extremadamente laboriosas, no aplicables para la rutina diagnóstica, no suficientemente sensibles ni específicas. Por lo tanto, a pesar de que un ensayo funcional sería ideadle gran ayuda, las herramientas bioinformáticas van probablemente a tener mucho mayor peso en el diagnóstico genético de la PQRAD que los estudios funcionales.
Dificultades añadidas en el diagnóstico genético mediante detección de mutaciones en la poliquistosis renal autosómica dominante
Individuos con poliquistosis renal autosómica dominante y sin mutación detectada
En aproximadamente un 9% de pacientes con PQRAD no se halla ninguna mutación patogénica ni en PKD1 ni en PKD229. Estos casos suelen tener una forma más leve de la enfermedad y con mayor frecuencia no poseen antecedentes familiares de la enfermedad. Así, la no detección de mutaciones no es solamente una falta de sensibilidad de la técnica sino que en realidad podría tratarse de otra enfermedad. Por una parte, pueden existir cambios en los intrones que afecten el splicing (podrían detectarse con la secuenciación intrones o estudio de mutaciones partir de ARN), puede haber mutaciones en regiones reguladoras como el promotor (por el momento no se han descrito mutaciones en estas regiones), o puede haber cambios que conlleven una alteración de la proteína que sea valorada como no patogénica in silico (herramientas bioinformáticas) pero que produzca una alteración sutil de la proteína que condicione la enfermedad. Por otra parte, se han descrito familias no ligadas a PKD1 ni a PKD2 aunque al analizar a fondo estas familias se cuestiona que el análisis de ligamiento sea correcto30. Y, por último, hay enfermedades quísticas con un fenotipo/curso clínico similar que pueden comportarse como fenocopias, causadas por los siguientes genes: HNF1b, PRKCSH, SEC63 o PKHD1.
Mosaicismo
El mosaicismo (la coexistencia en un individuo de poblaciones celulares normales y mutadas) es común en enfermedades genéticas con una elevada tasa de mutaciones de novo. En la PQRAD, sólo un 10% de los casos son de novo por lo que no se espera que este fenómeno sea muy frecuente. El mosacismo puede ser germinal (cuando afecta únicamente a la línea de células germinales), somático (si las poblaciones celulares genéticamente distintas son únicamente somáticas) o gonosómico (cuando implica tanto a células somáticas como germinales).
Se han descrito dos familias PQRAD con mosaicismo31,32. Para detectarlo es necesario disponer de la primera generación de la familia con uno o más familiares afectados por la enfermedad (caso de novo). El mosaicismo puede explicar la gran variabilidad fenotípica dentro de una familia y debe tenerse en cuenta su posible existencia cuando se ofrece consejo genético. Así, un paciente con padres aparentemente sanos y considerado, por lo tanto, como un caso de novo o esporádico, puede tener hermanos afectados si uno de sus progenitores padece un mosaicismo germinal (tiene células germinales con la mutación y sin la mutación). La presencia de mosaicismo será uno de los motivos por los cuales el análisis de ligamiento no será concluyente y puede ser negativo para PKD1 y PKD2. Por otra parte, cuando existe mosaicismo somático pueden presentarse niveles distintos de alelo mutado en los diferentes tejidos, por lo que una determinación en sangre periférica no será representativa de lo que está ocurriendo en los riñones31.
Correlación genotipo-fenotipo y alelos hipomórficos
Con los datos actuales existe una pobre correlación genotipo-fenotipo tanto para PKD1 como para PKD2. Aunque algún trabajo correlaciona algún tipo o localización de mutaciones, los resultados son insuficientes para establecer un pronóstico en función de lamutación detectada33,34.
Se creía que las mutaciones missense en los genes PKD eran inactivantes pero, hace poco28,35 se ha observado que algunos de estos cambios dan lugar a alelos hipomórficos o de penetrancia incompleta que se comportan como si la PQRAD se tratara de una enfermedad recesiva. También en modelos animales de PQRAD se ha observado este fenómeno36.
Recientemente, en familias con PQRAD se han detectado miembros con afectación muy diversa por la enfermedad debido a la presencia de alelos hipomórficos28,35. Así pues, individuos con los dos alelos PKD1 hipomórficos en trans presentan un fenotipo severo, mientras que los individuos de la familia con sólo uno de estos alelos presentan una forma mucho más leve de la enfermedad o incluso no se llegan a detectar quistes renales. Así pues, en estas familias la enfermedad puede etiquetarse de forma errónea como poliquistosis renal recesiva o como casos esporádicos de PQRAD. Tanto en un caso como en otro el error tendrá unas graves consecuencias de cara al consejo genético y dará lugar errores en un análisis de ligamiento. Para determinar la patogenicidad de estos alelos hipomórficos que dan lugar a un cambio de aminoácido en la poliquistina 1 debemos acudir, en la actualidad, a las herramientas bioinformáticas de las que disponemos.
Se calcula que entre un 43 y un 50% de la variabilidad clínica de la PQRAD, en cuanto a la edad de los pacientes con insuficiencia renal crónica terminal (IRCT), radica en efectos genéticos modificadores37-39. En la actualidad, no se conoce el grado de implicación de los alelos hipomórficos en la variabilidad fenótipica de la PQRAD.
Estado actual del diagnóstico genético de la polquistosis renal autosómica dominante
En general, no se recomienda realizar un estudio genético de PQRAD cuando el diagnóstico clínico y por la imagen es claro, pues se trata de un estudio económicamente costoso que en muchos casos no aporta información relevante. En la tabla 2 se resumen las indicaciones actuales del diagnóstico genético de la PQRAD.
La realización de un estudio genético para determinar si el gen causante de la enfermedad en el paciente es PKD1 o PKD2 es cuestionable, pues existe una gran variabilidad clínica dentro de cada gen40, aunque es evidente que ser portador de una mutación en el gen PKD2 conlleva un mejor pronóstico que tenerla en PKD141.
La ausencia de indicación extensiva del diagnóstico de la PQRAD se debe al coste de la técnica y a la complejidad actual para determinar si los cambios de secuencia del ADN que se detectan son en realidad mutaciones patogénicas. A medida que se abaraten las técnicas de secuenciación y se pueda determinar mejor la patogenicidad de los cambios detectados la técnica podría llegar hacerse de forma mucho más generalizada.
Por otra parte, en la actualidad no existe un tratamiento efectivo para la PQRAD, pero existen diversos ensayos clínicos en marcha. Es de esperar que, en unos años, dispongamos de tratamiento para la enfermedad y entonces será imprescindible que no existan dudas diagnósticas para tratar a un paciente. Cabe la posibilidad que las terapias deban iniciarse en edades jóvenes en las que el diagnóstico por la imagen resulta poco concluyente.
Conclusiones
La posibilidad de realizar un diagnóstico genético directo de la PQRAD es actualmente una realidad en nuestro país, aunque por las características del gen PKD1 no es un análisis sencillo ni económico. Debe estudiarse cada caso de forma individualizada con el fin de determinar la idoneidad de realizar un estudio genético y determinar qué tipo de estudio es el adecuado. Este tipo de diagnóstico es de especial interés para los donantes vivos, para casos neonatales y para casos esporádicos. El diagnóstico genético permite ofrecer diagnóstico prenatal o preimplantacional en familias con casos severos de la enfermedad y también permitirá tratar la enfermedad, cuando exista un tratamiento específico, en aquellos casos dudosos que sin confirmación genética no serían candidatos a tratamiento.
Referencias Bibliográficas
1. Belz MM, Hughes RL, Kaehny WD, Johnson AM, Fick-Brosnahan GM, Earnest MP, et al. Familial clustering of ruptured intracranial aneurysms in autosomal dominant polycystic kidney disease. Am J Kidney Dis 2001;38(4):770-6. [ Links ]
2. Chauveau D, Pirson Y, Verellen-Dumoulin C, Macnicol A, Gonzalo A, Grunfeld JP. Intracranial aneurysms in autosomal dominant polycystic kidney disease. Kidney Int 1994;45(4):1140-6. [ Links ]
3. Torra R, Darnell A, Estivill X, Botey A, Revert L. Interfamilial and intrafamilial variability of clinical expression in ADPKD. Contrib Nephrol 1995;115:97-101. [ Links ]
4. Hughes J, Ward CJ, Peral B, Aspinwall R, Clark K, San Millan JL, et al. The polycystic kidney disease 1 (PKD1) gene encodes a novel protein with multiple cell recognition domains. Nat Genet 1995;10(2):151-60. [ Links ]
5. Mochizuki T, Wu G, Hayashi T, Xenophontos SL, Veldhuisen B, Saris JJ, et al. PKD2, a gene for polycystic kidney disease that encodes an integral membrane protein. Science 1996;272(5266):1339-42. [ Links ]
6. Torra R, Badenas C, Darnell A, Nicolau C, Volpini V, Revert L, et al. Linkage, clinical features, and prognosis of autosomal dominant polycystic kidney disease types 1 and 2. J Am Soc Nephrol 1996;7(10):2142-51. [ Links ]
7. Hateboer N, Dijk MA, Bogdanova N, Coto E, Saggar-Malik AK, San Millan JL, et al. Comparison of phenotypes of polycystic kidney disease types 1 and 2. European PKD1-PKD2 Study Group. Lancet 1999;353(9147):103-7. [ Links ]
8. Grantham JJ, Cook LT, Torres VE, Bost JE, Chapman AB, Harris PC, et al. Determinants of renal volume in autosomal-dominant polycystic kidney disease. Kidney Int 2008;73(1):108-16. [ Links ]
9. Torra R, Badenas C, Pérez-Oller L, Luis J, Millán S, Nicolau C, et al. Increased prevalence of polycystic kidney disease type 2 among elderly polycystic patients. Am J Kidney Dis 2000;36(4):728-34. [ Links ]
10. Hughes J, Ward CJ, Peral B, Aspinwall R, Clark K, San Millan JL, et al. The polycystic kidney disease 1 (PKD1) gene encodes a novel protein with multiple cell recognition domains. Nat Genet 1995;10(2):151-60. [ Links ]
11. Polycystic kidney disease: the complete structure of the PKD1 gene and its protein. The International Polycystic Kidney Disease Consortium. Cell 1995;81(2):289-98. [ Links ]
12. Rossetti S, Consugar MB, Chapman AB, Torres VE, Guay-Woodford LM, Grantham JJ, et al. Comprehensive molecular diagnostics in autosomal dominant polycystic kidney disease. J Am Soc Nephrol 2007;18(7):2143-60. [ Links ]
13. Rossetti S, Chauveau D, Walker D, Saggar-Malik A, Winearls CG, Torres VE, et al. A complete mutation screen of the ADPKD genes by DHPLC. Kidney Int 2002;61(5):1588-99. [ Links ]
14. Phakdeekitcharoen B, Watnick TJ, Germino GG. Mutation analysis of the entire replicated portion of PKD1 using genomic DNA samples. J Am Soc Nephrol 2001;12(5):955-63. [ Links ]
15. Veldhuisen B, Saris JJ, De Haij S, Hayashi T, Reynolds DM, Mochizuki T, et al. A spectrum of mutations in the second gene for autosomal dominant polycystic kidney disease (PKD2). Am J Hum Genet 1997;61(3):547-55. [ Links ]
16. Nauli SM, Alenghat FJ, Luo Y, Williams E, Vassilev P, Li X, et al. Polycystins 1 and 2 mediate mechanosensation in the primary cilium of kidney cells. Nat Genet 2003;33(2):129-37. [ Links ]
17. Sharif-Naeini R, Folgering JH, Bichet D, Duprat F, Lauritzen I, Arhatte M, et al. Polycystin-1 and -2 dosage regulates pressure sensing. Cell 2009;139(3):587-96. [ Links ]
18. Battini L, Macip S, Fedorova E, Dikman S, Somlo S, Montagna C, et al. Loss of polycystin-1 causes centrosome amplification and genomic instability. Hum Mol Genet 2008;17(18):2819-33. [ Links ]
19. Li X, Luo Y, Starremans PG, McNamara CA, Pei Y, Zhou J. Polycystin-1 and polycystin-2 regulate the cell cycle through the helix-loop-helix inhibitor Id2. Nat Cell Biol 2005;7(12):1202-12. [ Links ]
20. Happe H, Leonhard WN, Van der WA, Van de WB, Lantinga-Van L, I, Breuning MH, et al. Toxic tubular injury in kidneys from Pkd1-deletion mice accelerates cystogenesis accompanied by dysregulated planar cell polarity and canonical Wnt signaling pathways. Hum Mol Genet 2009;18(14):2532-42. [ Links ]
21. Ravine D, Gibson RN, Walker RG, Sheffield LJ, Kincaid-Smith P, Danks DM. Evaluation of ultrasonographic diagnostic criteria for autosomal dominant polycystic kidney disease 1. Lancet 1994;343(8901):824-7. [ Links ]
22. Pei Y, Obaji J, Dupuis A, Paterson AD, Magistroni R, Dicks E, et al. Unified criteria for ultrasonographic diagnosis of ADPKD. J Am Soc Nephrol 2009;20(1):205-12. [ Links ]
23. Reed B, McFann K, Kimberling WJ, Pei Y, Gabow PA, Christopher K, et al. Presence of de novo mutations in autosomal dominant polycystic kidney disease patients without family history. Am J Kidney Dis 2008;52(6):1042-50. [ Links ]
24. Rossetti S, Strmecki L, Gamble V, Burton S, Sneddon V, Peral B, et al. Mutation analysis of the entire PKD1 gene: genetic and diagnostic implications. Am J Hum Genet 2001;68(1):46-63. [ Links ]
25. Santin S, García-Maset R, Ruiz P, Giménez I, Zamora I, Pena A, et al. Nephrin mutations cause childhood- and adult-onset focal segmental glomerulosclerosis. Kidney Int 2009;76(12):1268-76. [ Links ]
26. Tan YC, Blumenfeld JD, Anghel R, Donahue S, Belenkaya R, Balina M, et al. Novel method for genomic analysis of PKD1 and PKD2 mutations in autosomal dominant polycystic kidney disease. Hum Mutat 2009;30(2):264-73. [ Links ]
27. García-González MA, Jones JG, Allen SK, Palatucci CM, Batish SD, Seltzer WK, et al. Evaluating the clinical utility of a molecular genetic test for polycystic kidney disease. Mol Genet Metab 2007;92(1-2):160-7. [ Links ]
28. Rossetti S, Kubly VJ, Consugar MB, Hopp K, Roy S, Horsley SW, et al. Incompletely penetrant PKD1 alleles suggest a role for gene dosage in cyst initiation in polycystic kidney disease. Kidney Int 2009;75(8):848-55. [ Links ]
29. Rossetti S, Consugar MB, Chapman AB, Torres VE, Guay-Woodford LM, Grantham JJ, et al. Comprehensive molecular diagnostics in autosomal dominant polycystic kidney disease. J Am Soc Nephrol 2007;18(7):2143-60. [ Links ]
30. Paterson AD, Pei Y. Is there a third gene for autosomal dominant polycystic kidney disease? Kidney Int 1998;54(5):1759-61. [ Links ]
31. Consugar MB, Wong WC, Lundquist PA, Rossetti S, Kubly VJ, Walker DL, et al. Characterization of large rearrangements in autosomal dominant polycystic kidney disease and the PKD1/TSC2 contiguous gene syndrome. Kidney Int 2008;74(11):1468-79. [ Links ]
32. Connor A, Lunt PW, Dolling C, Patel Y, Meredith AL, Gardner A, et al. Mosaicism in autosomal dominant polycystic kidney disease revealed by genetic testing to enable living related renal transplantation. Am J Transplant 2008;8(1):232-7. [ Links ]
33. Rossetti S, Burton S, Strmecki L, Pond GR, San Millan JL, Zerres K, et al. The position of the polycystic kidney disease 1 (PKD1) gene mutation correlates with the severity of renal disease. J Am Soc Nephrol 2002;13(5):1230-7. [ Links ]
34. Rossetti S, Chauveau D, Kubly V, Slezak JM, Saggar-Malik AK, Pei Y, et al. Association of mutation position in polycystic kidney disease 1 (PKD1) gene and development of a vascular phenotype. Lancet 2003;361(9376):2196-201. [ Links ]
35. Vujic M, Heyer CM, Ars E, Hopp K, Markoff A, Orndal C, et al. Incompletely penetrant PKD1 alleles mimic the renal manifestations of ARPKD. J Am Soc Nephrol 2010;21(7):1097-102. [ Links ]
36. Lantinga-van L, I, Dauwerse JG, Baelde HJ, Leonhard WN, Van de WA, Ward CJ, et al. Lowering of Pkd1 expression is sufficient to cause polycystic kidney disease. Hum Mol Genet 2004;13(24):3069-77. [ Links ]
37. Tazon-Vega B, Vilardell M, Perez-Oller L, Ars E, Ruiz P, Devuyst O, et al. Study of candidate genes affecting the progression of renal disease in autosomal dominant polycystic kidney disease type 1. Nephrol Dial Transplant 2007;22(6):1567-77. [ Links ]
38. Fain PR, McFann KK, Taylor MR, Tison M, Johnson AM, Reed B, et al. Modifier genes play a significant role in the phenotypic expression of PKD1. Kidney Int 2005;67(4):1256-67. [ Links ]
39. Paterson AD, Magistroni R, He N, Wang K, Johnson A, Fain PR, et al. Progressive loss of renal function is an age-dependent heritable trait in type 1 autosomal dominant polycystic kidney disease. J Am Soc Nephrol 2005;16(3):755-62. [ Links ]
40. Torra R, Badenas C, Perez-Oller L, Luis J, Millan S, Nicolau C, et al. Increased prevalence of polycystic kidney disease type 2 among elderly polycystic patients. Am J Kidney Dis 2000;36(4):728-34. [ Links ]
41. Barua M, Cil O, Paterson AD, Wang K, He N, Dicks E, et al. Family history of renal disease severity predicts the mutated gene in ADPKD. J Am Soc Nephrol 2009;20(8):1833-8. [ Links ]
![]() Dirección para correspondencia:
Dirección para correspondencia:
Roser Torra Balcells y E. Ars Criach,
Fundació Puigvert.
Departament de Medicina.
Universitat Autònoma de Barcelona.
REDinREN. Instituto de Investigación Carlos III,
Cartagena 340-450, 08025, Barcelona
rtorra@fundacio-puigvert.es
ears@fundacio-puigvert.es
Enviado a Revisar: 9 Nov. 2010
Aceptado el: 12 Nov. 2010














