INTRODUCCIÓN
Cronobacter es un género de bacterias gramnegativas perteneciente a la familia Enterobacteriaceae cuyos miembros se consideran patógenos oportunistas transmitidos por alimentos. Este género (anteriormente denominado Enterobacter sakazakii y reclasificado en 2007) está integrado en la actualidad por siete especies: C. sakazakii, C. turicensis, C. malonaticus, C. muytjensii, C. dublinensis, C. universalis y C. condimenti (1). Cronobacter sakazakii se ha implicado en el desarrollo de infecciones neonatales graves que incluyen meningitis, sepsis y enterocolitis necrotizante (ECN) (2). También, aunque con mucha menos frecuencia, se han reportado otras enfermedades, como conjuntivitis, infecciones del tracto urinario, amigdalitis, diarrea y osteomielitis (3,4). Estas infecciones típicamente afectan a los recién nacidos más pequeños y vulnerables, con tasas de mortalidad que oscilan entre el 40 % y el 80 % en el caso de los prematuros (menos de 37 semanas de edad gestacional) y/o los recién nacidos de bajo peso al nacer (menos de 2500 g) (5,6). La combinación de un sistema inmunológico y una microbiota intestinal poco desarrollados supone para los recién nacidos un mayor riesgo de infección en comparación con otros grupos de edad (7,8). En el caso de los supervivientes de la infección por C. sakazakii, estos pueden desarrollar secuelas crónicas graves como alteraciones neurológicas (4,9,10).
La enfermedad se ha relacionado frecuentemente con los preparados en polvo para lactantes (PPL) y se puede presentar, por tanto, en forma de brotes. Aunque todavía se desconoce el reservorio habitual de Cronobacter, el hecho de haberse aislado de una gran variedad de alimentos y ambientes (en instalaciones de producción de leche en polvo y otros PPL, así como en otras plantas de producción de alimentos, hogares y hospitales) refleja su carácter ubicuo (4,6,11-13).
La capacidad que posee un microorganismo para originar una enfermedad (patogenicidad) depende de los factores de virulencia. En el caso de Cronobacter, la información sobre la patogenicidad y los factores de virulencia de los aislados obtenidos de diversas fuentes es todavía relativamente escasa, si bien algunas cepas producen compuestos similares a las enterotoxinas. Además, parecen existir diferencias entre la virulencia de los aislados clínicos, ambientales y de alimentos (1,4). Los estudios de secuenciación pangenómica han demostrado una alta capacidad de discriminación o diferenciación de las cepas de cada especie de Cronobacter, lo que genera una visión más precisa de las distancias genéticas que existen entre los aislados obtenidos en cada estudio (14,15).
Esta revisión tiene como objetivo proporcionar una actualización sobre la patogenicidad de Cronobacter para la población pediátrica a la luz de la información de la genómica bacteriana, que trata de identificar y rastrear los genotipos asociados a la enfermedad. La aplicación de estas estrategias contribuirá a reducir el riesgo de contaminación por Cronobacter en los entornos de producción de los PPL, mejorando así la seguridad alimentaria y protegiendo la salud infantil.
TAXONOMÍA
El género Cronobacter se consideró inicialmente como la especie Enterobacter sakazakii (1,9). Posteriormente se comprobó mediante secuenciación de diferentes genes que los aislados de E. sakazakii formaban varios grupos genéticamente distintos que podrían ser especies diferentes. El género Cronobacter está integrado en la actualidad por siete especies (1,16) que, según su relevancia clínica, se agrupan de la siguiente manera:
− El grupo 1 comprende C. sakazakii y C. malonaticus, que constituyen la mayoría de los aislados clínicos de pacientes de todos los grupos de edad.
− El grupo 2 comprende C. turicensis y C. universalis, que se han asociado raramente a casos clínicos, aunque C. turicensis se ha aislado también de lactantes asintomáticos (8), lo que puede indicar su posible relevancia clínica.
− El grupo 3 comprende C. dublinensis, C. muytjensii y C. condimenti, que son principalmente comensales ambientales y probablemente tienen poca o ninguna importancia clínica (1,4,9).
De las siete especies mencionadas, C. sakazakii es la especie predominante entre las que causan enfermedad (8). Cronobacter malonaticus se clasificó inicialmente como una subespecie de C. sakazakii debido a sus similitudes genéticas pero en la actualidad se considera una especie diferente debido a la presencia de genes específicos para el metabolismo del malonato (16). Más recientemente se ha comprobado que C. malonaticus, al igual que C. sakazakii, puede causar infecciones en niños al invadir y causar daños en las células del epitelio intestinal (8). No obstante, C. sakazakii tiene en su genoma más secuencias que codifican proteínas (marcos de lectura abiertos) en comparación con otras especies de Cronobacter, lo que posiblemente indica su mayor capacidad de disponer de proteínas relevantes para resistir el estrés y para la patogenicidad (16,17).
PATOGENIA
Cronobacter se considera un patógeno oportunista. La importancia de Cronobacter se debe a su asociación con infecciones neonatales graves (enterocolitis necrotizante, sepsis y meningitis), que en algunos casos se asocian a secuelas graves o la muerte. Las infecciones se han relacionado frecuentemente con la contaminación de los PPL y se pueden presentar, por tanto, en forma de brotes, además de como infecciones esporádicas (4,8).
La combinación de un sistema inmunológico poco desarrollado y un intestino con una barrera epitelial inmadura predispone a algunos niños recién nacidos a la colonización e infección por microorganismos patógenos (7,8). De hecho, la infección y el desarrollo posterior de la enterocolitis necrotizante (ECN), una enfermedad que se desarrolla cuando el tejido del revestimiento interno del intestino delgado o grueso se daña, inflama y necrosa, se ha relacionado con la colonización de células inmaduras del intestino del niño por C. sakazakii (8) (Fig. 1). Cronobacter sakazakii no solo puede suprimir la maduración de las células dendríticas (CD) sino que también puede invadir las CD para sobrevivir y diseminarse, evadiéndose así del sistema inmunológico (8) (Fig. 1). Se ha observado que los macrófagos y los neutrófilos desempeñan un papel importante en la eliminación de C. sakazakii al regular la producción de CD, pero la respuesta inmunitaria es poco eficaz debido a la naturaleza intracelular de C. sakazakii (8) (Fig. 1). La ECN puede desarrollarse debido a citoquinas proinflamatorias liberadas durante la infección, que estimulan la producción de óxido nítrico que daña la mucosa intestinal (8) (Fig. 1). Una mayor alteración de las capas intestinales puede permitir que C. sakazakii llegue al torrente sanguíneo y/o al cerebro, lo que puede causar sepsis y/o meningitis, respectivamente (4).
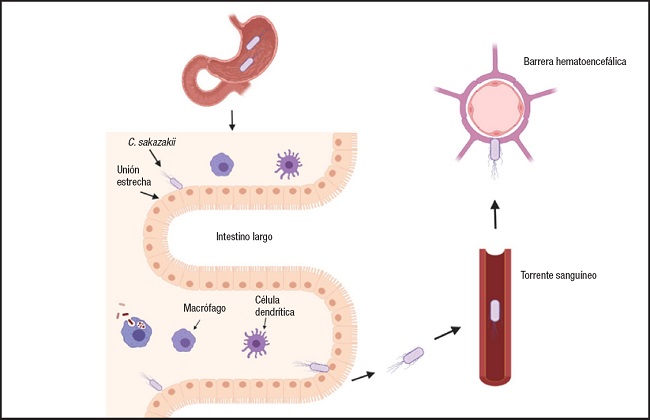
Figura 1. Descripción general de la patogenia de Cronobacter sakazakii en un lactante. Este diagrama ilustra el tránsito de C. sakazakii a través del estómago y el intestino delgado hasta llegar al intestino grueso. En el intestino grueso, C. sakazakii puede evadir las células del sistema inmunitario para alterar la capa epitelial, lo que podría causar enterocolitis necrotizante, y entrar en el torrente sanguíneo para causar sepsis. Desde el torrente sanguíneo, C. sakazakii puede atravesar la barrera hematoencefálica y causar meningitis. La imagen fue creada con BioRender.com (8). Copyright© 2021: Ke, Parreira, Goodridge and Farber (8).
ECOLOGÍA
Cronobacter sakazakii es un microorganismo ubicuo y se ha aislado de una amplia variedad de fuentes, incluidos alimentos de origen vegetal y animal como los productos lácteos y los PPL en general (12,13,18), así como la leche materna (1). Igualmente se ha aislado en todo tipo de ambientes, como plantas de producción de PPL y de alimentos en general, hospitales y hogares (5,6). Los PPL se pueden contaminar con C. sakazakii debido a la falta de higiene y el flujo de trabajo en las instalaciones de producción, como la filtración inadecuada en el proceso de secado por pulverización (3,19). También C. sakazakii se ha encontrado en otros ambientes, como los hospitales, tanto en el aire como en el polvo y en el cuerpo humano, por lo que el manejo cuidadoso de los PPL puede no ser suficiente para eliminar el riesgo de infección (1). Por ejemplo, aproximadamente el 9 % de las hierbas aromáticas y medicinales analizadas en un estudio estaban contaminadas con C. sakazakii (20). Por tanto, como C. sakazakii está muy extendido en la naturaleza, las fuentes de contaminación pueden variar y sus reservorios no están claros (4).
EPIDEMIOLOGÍA
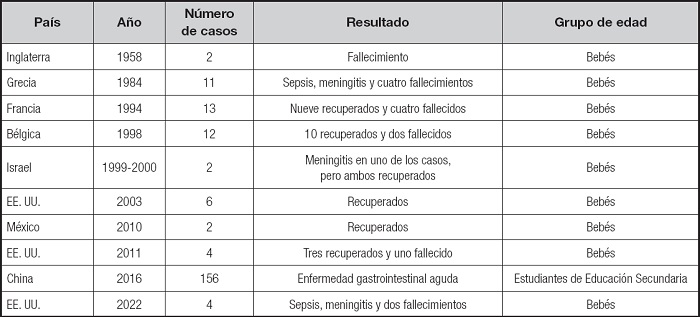
Los lactantes, especialmente los de menos de 2 meses de edad, de bajo peso al nacer (de menos de 1500 a 2500 g) y/o prematuros (menos de 37 semanas de edad gestacional) son el principal grupo de edad asociado con brotes de infecciones de C. sakazakii (6,15). No obstante, los brotes de infecciones causadas por C. sakazakii son poco frecuentes (8) (Tabla I). En algunos casos, los brotes de infecciones por Cronobacter se han asociado a unidades de cuidados intensivos neonatales. Las infecciones se han vinculado directamente a PPL reconstituidos que pueden estar contaminados intrínsecamente o que se contaminan durante la preparación y administración. Una característica común en algunos de estos brotes es el abuso de temperatura del alimento preparado, que permitiría el crecimiento bacteriano (8).
Cronobacter se ha aislado de tráqueas de niños y de sondas de alimentación de recién nacidos alimentados tanto con leche materna como con PPL (8). Por lo tanto, se deben investigar también otras fuentes como causa de los brotes y no solo los PPL. No obstante, los PPL son una fuente importante de infección por C. sakazakii en los bebés (6) porque los ingredientes sensibles al calor (p. ej., vitaminas) se agregan después de la esterilización del PPL y la contaminación puede ocurrir en el entorno de la fabricación (11). Una vez empaquetados los PPL contaminados, C. sakazakii puede permanecer viable hasta 2 años (21). Además, el uso de agua a menos de 70 °C para rehidratar los PPL podría aumentar la probabilidad de contaminación (22,23). El manejo inadecuado y/o la falta de conocimiento sobre cómo reconstituir adecuadamente los PPL pueden contribuir a su contaminación con C. sakazakii y a la supervivencia del microorganismo. Se recomienda que el PPL reconstituido se use dentro de las 2 horas posteriores a la preparación y se deseche si no se termina en una sola toma (22,23).
A principios de la década que comenzó en los 2000, la FAO y la OMS colaboraron en una evaluación de riesgos para mejorar la seguridad microbiológica de los PPL. Salmonella y Cronobacter se clasificaron como patógenos de categoría A, lo que significaba una clara evidencia de causalidad en la contaminación de los PPL (22,23). Los patógenos de la categoría B, incluidos los géneros Escherichia, Klebsiella y Citrobacter, presentaban una cierta posibilidad de contaminación, mientras que los patógenos de la categoría C, como los géneros Clostridium, Staphylococcus y Listeria, no estaban implicados en la contaminación de los PPL (22,23). Se elaboró un modelo de evaluación de riesgos para C. sakazakii en PPL para cuantificar los riesgos específicos para los lactantes (http://tools.fstools.org/esakmodel/ESAKRAModelWizard.aspx). El modelo FAO/OMS considera cuatro riesgos potenciales de los PPL: 1) la población inicial de C. sakazakii, 2) la temperatura de reconstitución, 3) los métodos de manipulación y 4) el período de alimentación.
VIRULENCIA
La información sobre la diversidad genética, patogenicidad y virulencia de los aislados de Cronobacter obtenidos de diversas fuentes es todavía relativamente escasa, si bien algunas cepas producen compuestos similares a las enterotoxinas (1,4). Además, parecen existir diferencias entre la virulencia de los aislados clínicos, ambientales y de alimentos. Estas diferencias pueden originarse por el hecho de que los microorganismos poseen genes accesorios que se pueden perder y ganar frecuentemente como resultado de los procesos de recombinación genética (17). En el caso de C. sakazakii, el análisis genómico de gran número de cepas de diferentes orígenes ha permitido comprobar que existen muchos genes específicos de las cepas humanas (3297 genes), alimentarias (2570 genes) y ambientales (1968 genes) (17) (Fig. 2).
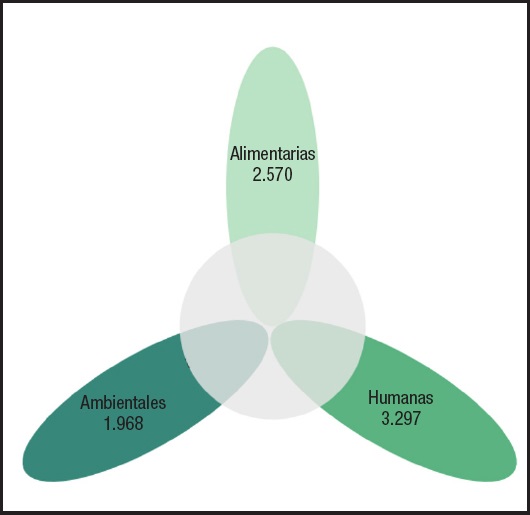
Figura 2. Número de genes que se encuentran exclusivamente en grupos de cepas de Cronobacter sakazakii de diferentes fuentes (humanas, 3297 genes; alimentarias, 2570 genes; ambientales, 1968 genes) (17) (©The Authors, 2019. Open Access).
FACTORES DE VIRULENCIA
Cronobacter sakazakii tiene muchos genes relacionados con la supervivencia y la virulencia, pero la función de algunos de ellos aún se está investigando.
Hfq, también conocido como Host Factor 1, es una proteína reguladora implicada en la patogénesis, la comunicación y la supervivencia de varios patógenos transmitidos por los alimentos, incluyendo C. sakazakii (8). Se ha comprobado que el gen hfq de C. sakazakii regula los genes de virulencia y que su ausencia reduce la tasa de infección de células Caco-2, la supervivencia en macrófagos y la resistencia al estrés oxidativo (8).
Las proteínas de la membrana externa (OMP, Outer Membrane Proteins) desempeñan un papel importante en la virulencia de C. sakazakii. Se ha comprobado que las dos OMP, OmpA y OmpX, son esenciales para la adhesión y la invasión de las células intestinales por C. sakazakii (24). También se ha visto que OmpA facilita la invasión de la barrera hematoencefálica por C. sakazakii (8).
Las enterotoxinas y endotoxinas de C. sakazakii siguen siendo funcionales durante la fabricación y la vida útil de los PPL, lo que puede relacionarse con la patogénesis de la infección por C. sakazakii (14). Las enterotoxinas de C. sakazakii pueden tener una función similar a la de los lipopolisacáridos y estimular una respuesta inflamatoria en el huésped, que puede conducir a la ECN (24), mientras que sus endotoxinas pueden ayudar a la translocación del patógeno a través de las barreras intestinal y hematoencefálica para causar bacteriemia y meningitis (11).
El ácido siálico se encuentra en la leche materna, los PPL, la mucina intestinal y el cerebro humano, ya que es un compuesto bioactivo necesario para el desarrollo del cerebro (11,16). La secuenciación del genoma completo de C. sakazakii reveló que, dentro del género Cronobacter, dicha especie es la única que puede utilizar el ácido siálico como fuente de carbono, ya que posee el clúster de genes nanAKTR (4,16,24). La capacidad de C. sakazakii para metabolizar el ácido siálico puede ser un factor de supervivencia en los PPL y de invasión del intestino y el cerebro, pero se necesitan más investigaciones para validar esta hipótesis (14).
ADAPTACIÓN AL ESTRÉS
Cronobacter sakazakii es un enteropatógeno resistente capaz de sobrevivir a la desecación, el calor y el bajo pH, condiciones que se dan durante los procesos de fabricación de alimentos y en el tracto gastrointestinal humano (19,21,25). Además, su resistencia frente a los componentes del tracto gastrointestinal, incluidos los ácidos estomacales y los líquidos biliares, puede ser un factor clave en el desarrollo de enfermedades en todos los grupos de edad, y especialmente en los lactantes debido a su menor acidez estomacal.
Los lactantes tienen una acidez gástrica y una concentración de sales biliares menores en comparación con los adultos (19,21,25), lo que puede facilitar la supervivencia de los patógenos transmitidos por los alimentos en el tracto gastrointestinal (19,21,25). Las células de C. sakazakii pueden sobrevivir a valores de pH tan bajos como 4,5, una concentración de sales biliares del 5 %, una concentración de sal de hasta el 10 %, una temperatura de hasta 60 °C y más de 20 días de desecación (19,21,25). De hecho, las condiciones subletales en el tracto gastrointestinal de los lactantes pueden exacerbar la resistencia de C. sakazakii a los compuestos antimicrobianos producidos por las bacterias intestinales o los probióticos (8). Además, la exposición a condiciones ácidas y térmicas subletales puede aumentar la resistencia al calor de C. sakazakii durante la rehidratación de los PPL utilizando agua a temperaturas por debajo de 64 °C (25), lo que apoya la recomendación de la OMS de rehidratar los PPL utilizando agua que se encuentre a más de 70 °C (23).
GENÉTICA DE POBLACIONES Y PATOGENICIDAD
Dado que Cronobacter es un organismo ubicuo, se requieren sistemas de tipificación que permitan identificar los genotipos tanto en las investigaciones epidemiológicas como en las ambientales.
TIPIFICACIÓN MEDIANTE SECUENCIACIÓN DE MÚLTIPLES LOCI (MLST)
La genética de poblaciones estudia la variabilidad genética de las poblaciones bacterianas a partir de las frecuencias alélicas de varios genes o «loci», y trata de explicar esta variabilidad en términos de mutación, selección, recombinación genética, etc. Uno de los métodos de análisis más precisos y reproducibles de la genética de poblaciones bacteriana es la tipificación mediante secuenciación de múltiples loci (multi-locus sequence typing, MLST). Para cada uno de los loci estudiados, toda secuencia distinta (incluso una sola base) constituye un alelo diferente. Los alelos correspondientes a todos los loci estudiados presentes en las distintas cepas constituyen su tipo de secuencia o ST (sequence type), perfil alélico o genotipo (14). La tipificación de Cronobacter para comprender su diversidad ha llevado al desarrollo de un sistema específico de MLST que está disponible en línea (https://www.pubMLST.org/cronobacter/). El sistema MLST de C. sakazakii reveló la existencia de clones estables, algunos de los cuales pudieron rastrearse durante un período de 50 años, y que procedían de de una amplia gama de países y fuentes (26).
De especial interés es la existencia de ST integrados principalmente por aislados clínicos, o por aislados de PPL y clínicos, o por aislados de origen no clínico. Esto puede reflejar diferencias ecológicas del organismo. Según Ogrodzki y Forsythe (26), en la base de datos de MLST de Cronobacter la mayoría de las cepas son C. sakazakii (68 %) y C. malonaticus (13 %). De un total de 236 ST identificados en C. sakazakii, los predominantes en las muestras clínicas son los ST 1, 4, 8 y 12. ST4 es el ST más numeroso (208 cepas de un total de 1126). Más de la mitad de los aislados con perfil ST4 proceden de niños (53 %) (26). En estudios realizados con muestras ambientales de plantas de producción de leche en polvo (12) o con los propios alimentos infantiles comercializados (13), también se ha encontrado que predominan los genotipos ST4 y ST1. Al analizar la capacidad de formar biofilm, se ha observado que ST4 es predominante entre las cepas que forman biofilm, lo que podría explicar su presencia mayoritaria en el ambiente de las plantas de producción de alimentos infantiles (13).
SECUENCIACIÓN DEL GENOMA COMPLETO (WGS)
Los estudios de secuenciación del genoma completo (WGS, whole genome sequencing) han demostrado una alta discriminación o diferenciación de las cepas de cada especie de Cronobacter, lo que genera una visión más precisa de las distancias genéticas que existen entre los aislados obtenidos en cada estudio (1,4,15). La WGS se utiliza tanto para identificar la especie de Cronobacter como para establecer identidades y detectar los genes asociados a la resistencia a antibióticos, a la virulencia, a la formación de biofilm, a la resistencia al estrés, etc., lo que permite establecer vínculos epidemiológicos más precisos. Por ejemplo, mediante WGS se identificó recientemente en C. sakazakii un operón de resistencia al arsénico y un grupo de genes del sistema de secreción de tipo 4 que contenía un gen de fosfolipasa D, un factor de virulencia bien conocido (27). En otro estudio realizado en Chile se identificaron cinco cepas de Cronobacter, dos de ellas C. sakazakii ST1 y dos C. sakazakii ST31, y una cepa de C. malonaticus ST60 (28). Todos los aislados eran resistentes a la cefalotina, y se detectaron 19 genes de resistencia a antibióticos y 32 genes asociados a la virulencia (proteínas flagelares, proteínas de la membrana externa, quimiotaxis, hemolisinas, invasión, activador del plasminógeno, colonización, regulador transcripcional, supervivencia en macrófagos, uso de ácido siálico y genes de sistemas toxina-antitoxina) (28).
CONCLUSIONES
El uso de metodologías moleculares como la MLST y la WGS combinadas mejora la identificación del patógeno y proporciona información precisa sobre su prevalencia y diversidad genética, que es relevante para la toma de las decisiones que se deben adoptar para aumentar la seguridad de los alimentos infantiles. Dado que los alimentos infantiles en polvo son consumidos por grupos considerados vulnerables, la presencia de factores de virulencia y resistencia a antibióticos en cepas de Cronobacter aisladas de estos productos debe analizarse con mayor detención por la relación directa que tienen con la severidad de la enfermedad asociada a este patógeno. Por todo ello, es necesario que los organismos públicos de investigación y las empresas productoras de alimentos infantiles en polvo realicen más actividades de I+D+i para mejorar el control preventivo de la contaminación de estos alimentos.