Mi SciELO
Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO -
 Accesos
Accesos
Links relacionados
 Citado por Google
Citado por Google -
 Similares en
SciELO
Similares en
SciELO  Similares en Google
Similares en Google
Compartir
Gaceta Sanitaria
versión impresa ISSN 0213-9111
Gac Sanit vol.17 no.6 Barcelona nov./dic. 2003
NOTA METODOLÓGICA
Aplicación de las redes neuronales artificiales para
la estratificación de riesgo de mortalidad hospitalaria
J. Trujillano a /J. March b /M. Badia a /A. Rodríguez a /A. Sorribas b
aUnidad de Cuidados Intensivos. Hospital Universitario Arnau de Vilanova de Lleida. Lleida.
bDepartamento de Ciencias Médicas Básicas. Universidad de Lleida. Lleida. España.
Correspondencia: J. Trujillano. Hospital Arnau de Vilanova. Unidad de Cuidados Intensivos. Avda. Rovira Roure, 80.
28198 Lleida. España.
Correo electrónico: jtruji@cmb.udl.es
Recibido: 31 de marzo de 2003.
Aceptado: 15 de septiembre de 2003.
(Application of artificial neural networks for risk stratification of hospital mortality)
| Resumen | Abstract |
Introducción
La construcción de sistemas de clasificación de pacientes (o de ajuste de riesgos) permite comparar la efectividad y la calidad de hospitales y los servicios hospitalarios, aportando información útil para la toma de decisiones de gestión y sobre el manejo de los pacientes1. Los sistemas de ajuste de riesgos para estratificar la gravedad de los pacientes respecto a un resultado clínico, se construyen, en general, a partir de variables asistenciales y utilizando técnicas estadísticas basadas en la regresión logística (RL)2.
Las redes neuronales artificiales (RNA) son sistemas de cálculo que se asemejan a las redes neuronales biológicas al utilizar nodos (neuronas) interconectados. Estos nodos reciben la información, realizan operaciones sobre los datos y transmiten sus resultados a otros nodos. El procedimiento consiste en entrenar a las RNA para que aprendan patrones complejos de relaciones entre las variables predictoras y de resultado y que sean capaces de enfrentarse a nuevos datos dando las respuestas esperadas3. Se definen como sistemas no lineales, flexibles y con gran capacidad de generalización. Estas propiedades han hecho que se difundieran en todos los campos científicos y que se demostrara su equivalencia o superioridad sobre algunas técnicas estadísticas4.
El interés en la aplicación de las RNA en medicina durante los últimos 10 años no ha hecho más que aumentar, como refleja el número progresivamente creciente de publicaciones que incluyen esta metodología5,6. Las áreas que han ido ocupando son el reconocimiento de imágenes7, análisis de ondas8, procedimientos de farmacología9, epidemiología10, predicción de resultados11 y procesos diagnósticos12.
La utilización de las RNA para la estratificación de riesgo ofrece como ventaja un posible aumento del poder predictivo (precisión), que se ha evaluado en un 5-10%, ya que no trabajan con las limitaciones rígidas de los modelos estadísticos13. Frente a las técnicas de RL, las RNA tienen en cuenta las relaciones no lineales, de manera automática, sin necesidad de seguir un modelo concreto, y la posible interdependencia de las variables de entrada.
En una unidad de cuidados intensivos (UCI) se utilizan de forma habitual sistemas de cálculo de probabilidad de muerte (como criterio de gravedad de los enfermos), y uno de los sistemas más habituales es el Acute Physiology and Chronic Health Evaluation II (APACHE II) construido con técnica de RL14.
Los objetivos de nuestro trabajo se centran en demostrar la utilidad de la metodología basada en redes neuronales para la estratificación de riesgos, aplicándola al cálculo de probabilidad de mortalidad hospitalaria, utilizando las variables del sistema APACHE II en una UCI concreta. Como referencia se utiliza un modelo de regresión logística.
Método
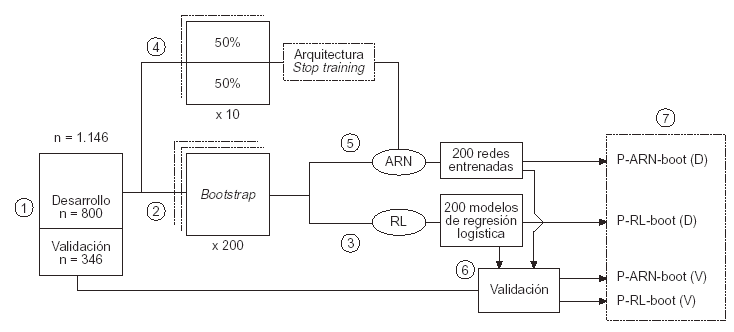
En la figura 1 se muestra el algoritmo del esquema utilizado para el desarrollo de la metodología aplicada.
Figura 1. Diagrama secuencial del desarrollo de la aplicación. RL: regresión logística; RNA: red neuronal artificial; P-RNA-D: probabilidad media de 200 redes entrenadas bootstrap y enfrentadas al grupo de desarrollo; P-RNA-V:
probabilidad media de 200 redes entrenadas bootstrap y enfrentadas al grupo de validación; P-RL-D: probabilidad
media de 200 modelos de regresión logística bootstrap y enfrentados al grupo de desarrollo; P-RL-V: probabilidad
media de 200 modelos de regresión logística bootstrap y enfrentados al grupo de validación (véase texto).
Sujetos
El estudio se ha realizado en la UCI polivalente del Hospital Universitario Arnau de Vilanova de Lleida. Se ha estudiado a los pacientes en un período de 5 años (1997-2001). No se incluye a los pacientes coronarios, los sujetos a cirugía cardíaca ni los quemados. Se ha excluido a los pacientes menores de 16 años, los que se han trasladado y los que han permanecido menos de 24 h ingresados. En los pacientes que reingresan sólo se ha tenido en cuenta el primer ingreso. Se han recogido, de forma prospectiva por un equipo entrenado, las variables demográficas, de evolución y de gravedad necesarias para el cálculo del sistema APACHE II14. La realización del estudio fue aprobada por el comité ético del hospital asegurando en todo momento el anonimato de los pacientes.
Paso 1
Se utilizan 14 variables fisiológicas (se amplían de 12 a 14, ya que para valorar la oxigenación utilizamos PaO2, más FiO2 y PaCO2), la edad y dos variables para determinar la puntuación según enfermedad crónica (enfermedad crónica y urgente/programado) que completan 17 variables de entrada (fig. 2). La variable de salida es la mortalidad hospitalaria. El cálculo de la probabilidad de muerte basada en APACHE II se hace de forma estándar, convirtiendo la puntuación APACHE II y aplicando la fórmula logística con los coeficientes publicados en el artículo original de Knaus et al14; no se incluye el ajuste por grupos diagnósticos ya que motivaría añadir más de 40 variables. Los 1.146 pacientes que cumplen los criterios de inclusión se asignaron aleatoriamente, un 70% al grupo de desarrollo (n = 800) y el 30% restante al grupo de validación (n = 346).
Figura 2. Arquitectura óptima de la red (perceptrón multicapa entrenado con algoritmo backpropagation). PAM: presión arterial media; FC: frecuencia cardíaca; FR: frecuencia respiratoria; FiO2: fracción inspirada de oxígeno; PaO2: presión arterial de oxígeno; PaCO2: presión arterial de anhídrido carbónico; Hto: hematocrito. Definición de variables según artículo original14.
Paso 2. Remuestreo bootstrap del grupo de desarrollo
Como el número de casos disponibles para el desarrollo del modelo es limitado, existe el riesgo de que tengan una pobre representatividad de la población; por tanto, debemos utilizar técnicas que optimizan los datos disponibles para conseguir una buena generalización. Como solución aplicamos técnicas de remuestreo (bootstrap) que han demostrado ser útiles para este fin15. En nuestro caso, para conseguir una precisión suficiente, este remuestreo debe repetirse, por lo menos, 200 veces16,17.
Paso 3. Modelo de regresión logística
Se utiliza un modelo de regresión logística múltiple con incorporación de todas las variables (full model). Se efectuará el cálculo en las 200 remuestras bootstrap del grupo de desarrollo. Los 200 modelos resultantes (sus coeficientes) se emplearán para calcular las probabilidades en el conjunto de desarrollo original. Con estas 200 probabilidades calculamos la probabilidad media, expresada como P-RL-D, y su error estándar.
Paso 4. Modelo de red neuronal artificial
Para un acercamiento a la metodología basada en RNA, remitimos a los lectores interesados a las revisiones publicadas y a los recursos de Internet que se actualizan de forma periódica5,18,19.
El tipo de red empleado es un perceptrón multicapa entrenado con algoritmo de backpropagation y función de activación sigmoidea. Utilizamos un modelo de 3 capas (de entrada, oculta y de salida) (fig. 2). La selección de la arquitectura y los parámetros óptimos se basan en un procedimiento empírico y en la validación cruzada. El conjunto de desarrollo se divide (al 50% para asegurar representatividad) en un conjunto de entrenamiento y otro de verificación. Esta división se hace de forma aleatoria y se repite 10 veces para confrontar los resultados en estas 10 ocasiones. El entrenamiento supervisado supone la presentación repetida del conjunto de entrenamiento a la red, en cada iteración se realiza un ajuste de los pesos para reducir al mínimo la función de error de la red. Los pesos son los valores internos de la red que se asemejan a las fuerzas sinápticas de los modelos biológicos. La función de coste o función de error evaluada (tanto en el conjunto de entrenamiento como en el de verificación) es la raíz del error cuadrático medio (ECM) entre las predicciones y los valores reales. Se añaden o retiran nodos de la capa oculta hasta conseguir reducir al mínimo el ECM (en el conjunto de verificación) lo que también determina el momento de parar el entrenamiento (stop training). Otros parámetros que se modifican durante el proceso de entrenamiento (coeficiente de aprendizaje, momento, etc.) se ajustan para conseguir esta optimización.
Paso 5. Entrenamiento de la red neuronal artificial
Las condiciones de entrenamiento fijadas en el punto anterior servirán para entrenar 200 redes con los datos de los 200 remuestras bootstrap del conjunto de desarrollo. Cuando estas redes se enfrentan a los datos del conjunto de desarrollo original, determinan 200 probabilidades y su media se denomina P-RNA-D.
Paso 6. Validación de los modelos
Tanto los 200 modelos de regresión logística como las 200 redes entrenadas deben enfrentarse a los datos del conjunto de validación. Las probabilidades medias calculadas se identificarán como P-RL-V y P-RNA-V, respectivamente.
Paso 7. Comparación de los modelos
Para comparar los distintos modelos se medirán sus propiedades de discriminación por medio del área bajo la curva ROC (ABC)20,21 y la calibración con el test de bondad de ajuste de Hosmer-Lemeshow C22, la construcción de las curvas de calibración y el cálculo de las razones de mortalidad estandarizada (RME, que es el cociente entre el número de muertos observados y el número de muertes esperadas según el modelo de predicción) con sus intervalos de confianza23.
Utilizamos el test de Bland-Altman para evaluar la concordancia entre las probabilidades obtenidas por cada modelo24.
Definimos caso extremo25 cuando el paciente alcanza una diferencia de probabilidad entre el modelo de RL y el de RNA con valor absoluto igual o superior a 0,2.
Los cálculos estadísticos se realizaron con el programa SPSS 10.0. El programa utilizado para la creación de las redes es Qnet 97 (Vesta Services Inc.)26.
Resultados
Características del grupo de estudio (nuestra UCI)
Nuestra UCI (tabla 1) queda definida por tener pocos pacientes programados, en comparación con unidades de su entorno, lo que determina una alta mortalidad y una estancia media prolongada. No se observan diferencias significativas entre los conjuntos de desarrollo y de validación.
Modelo de red neuronal artificial
El método de selección nos llevó a una arquitectura óptima con 9 nodos en la capa oculta y nodos plenamente interconectados (fig. 2). El punto de parada del entrenamiento quedó establecido en 1.500 iteraciones. Los parámetros propios del proceso de entrenamiento de la red fueron un coeficiente de aprendizaje de 0,01 y momento de 0,3.
Comparación de los modelos
En la tabla 2 se muestran los resultados de la comparación entre los diversos modelos. Se aprecian buenos resultados, tanto en discriminación como en calibración, del sistema APACHE II (en el conjunto de desarrollo y en el de validación). En el grupo de desarrollo se observan unos valores significativamente más altos en el ABC ROC de la RNA frente al modelo APACHE II. La RNA, comparada con el modelo de RL, muestra unos valores mejores (que se mantienen en el grupo de validación) tanto en discriminación como en calibración, aunque estos valores no alcanzan diferencia significativa.
En la figura 3 se muestran las curvas ROC y las curvas de calibración correspondientes al grupo de validación.
Figura 3. Curvas ROC y curvas de calibración en el grupo de validación. a) Curvas ROC. b) Curvas de calibración.
Línea sencilla: probabilidad APACHE II; línea discontinua: probabilidad media del modelo de regresión logística bootstrap; línea gruesa: probabilidad media del modelo de red neuronal artificial bootstrap.
Análisis por grupos diagnósticos
En los pacientes traumáticos (27%) ningún modelo alcanza una precisión suficiente (el sistema APACHE II se muestra como el mejor modelo, pero con ABC ROC menor de 0,75 y mala calibración tanto en el conjunto de desarrollo como en el de validación). En los pacientes respiratorios (25%) los mejores resultados se consiguen con las redes neuronales; y dentro de este grupo, los pacientes con enfermedad pulmonar obstructiva crónica (EPOC) (13%) son los que peor comportamiento tienen con el APACHE II y con RL manteniendo aceptables propiedades la RNA (ABC = 0,87 [0,78-0,91]).
Comparación de probabilidades entre los modelos
La técnica de Bland-Altman (fig. 4) nos demuestra la falta de concordancia en la asignación de probabilidad entre los modelos de RL y RNA (resultados mostrados en el conjunto de validación). La mayor concordancia tiende a producirse en los valores bajos del rango de probabilidad y se pierde al superar el 35% de probabilidad de muerte calculada.
Figura 4. Test de Bland-Altman (grupo de validación) entre las probabilidades calculadas por el método de regresión
logística frente a las probabilidades calculadas por modelo de red neuronal artificial. Las líneas de puntos diferencian
(por encima o por debajo) a 2 desviaciones estándares ± la media de la diferencia entre las probabilidades; P-RNA-V:
probabilidad media de 200 redes entrenadas bootstrap; P-RL-V: probabilidad media de 200 modelos de regresión
logística bootstrap.
Del total del grupo de estudio se identificó a 93 pacientes (8%) como casos extremos. Es difícil analizar la interrelación entre variables, pero encontramos que en el subgrupo de estos pacientes donde la probabilidad asignada por RL es claramente superior a la calculada por RNA (42 pacientes), la mayoría (36 pacientes) tiene alteraciones neurológicas, y apreciamos que la variable Glasgow adquiere más importancia en el modelo de RL manteniendo valores similares el resto de las variables.
Discusión
El primer análisis de nuestros resultados está dirigido a los buenos resultados encontrados con el modelo APACHE II, ya que este hecho no coincidía con otros resultados que habíamos ido obteniendo con series más pequeñas que analizaban menos años en nuestra base de datos. Nuestra UCI se caracteriza por tener menos pacientes quirúrgicos programados y más mortalidad que la serie que sirvió para confeccionar originalmente el sistema APACHE II. Este argumento lo utilizábamos para justificar, sobre todo, las desviaciones en la calibración, aspecto que casi es óptimo ahora que po demos analizar más pacientes. Esto apoya el concepto de la gran dependencia que tiene el tamaño muestral en cualquier análisis de estratificación de riesgo.
La alta mortalidad encontrada en nuestra serie está condicionada también por no incluir a los pacientes coronarios. No pudimos incluirlos por problemas de seguimiento asistencial, dadas las características de la atención de este grupo de pacientes en nuestro hospital.
Como resultado global, en nuestro trabajo encontramos mejores resultados con la metodología basada en RNA, aunque no alcanzan a ser significativos. Este resultado es similar al logrado con otras series27,28. En una revisión efectuada por Sargent, que analizaba 28 estudios en pacientes oncológicos, concluye que las redes son equivalentes o ligeramente superiores a la RL, al no tener que depender de exigencias rígidas de independencia de las variables o de modelos lineales29.
También vemos que a pesar de utilizar técnicas de remuestreo y de validación cruzada tenemos, en nuestra serie, cierto problema de «sobreaprendizaje»: la red aprende los patrones del conjunto de entrenamiento de forma muy precisa, pero pierde en capacidad de generalización al enfrentarse a nuevos datos del conjunto de validación.
Trabajando con redes, las condiciones necesarias para conseguir una buena generalización se centran en tres aspectos: a) que la información recogida en los datos sea suficiente (esto incide en el tamaño de la serie y en la calidad en la recogida de datos); b) que la «función» que aprenda la red sea suave (pequeños cambios en las variables de entrada produzcan pequeños cambios en la de salida), y c) que el tamaño del conjunto de entrenamiento sea suficiente y representativo de los datos totales19,30. El tamaño necesario viene determinado por el número de parámetros de la red, y se necesitan 5 registros por parámetro estimado28. En nuestro ejemplo con 17 variables de entrada, 9 nodos ocultos y un nodo de salida (que son 162 parámetros), 800 casos son suficientes.
El algoritmo que nosotros proponemos cumple con estas condiciones cuando se trabaja con series limitadas, y puede aplicarse en otro tipo de población o problema sanitario. Existen otros procedimientos basados en diferentes técnicas de remuestreo y de aprendizaje que han sido aplicados en otras poblaciones27,31.
El hecho de obtener resultados similares, en las propiedades de discriminación y calibración con los modelos de predicción estadísticos y neuronales, ha llevado a algunos autores a afirmar que la relación entre las variables es independiente y prácticamente lineal27. Nosotros aportamos la visión de que poder obtener probabilidades individuales diferentes implica que la relación entre las variables es diferente al aplicar RL o RNA. Es verdad que la interpretación de esta interrelación es difícil (concepto de caja negra de las RNA), pero podemos estudiar a los pacientes que se definen como casos extremos (pacientes con problemas neurológicos en nuestra serie), o comparar el distinto comportamiento según los grupos diagnósticos (p. ej., nuestros resultados diferentes en pacientes traumáticos y con EPOC).
El modelo APACHE II ha sido analizado en múltiples subgrupos de pacientes32, y en los pacientes traumáticos es donde se ha encontrado una peor precisión33-35. En nuestros pacientes traumáticos tampoco conseguimos una aceptable precisión con el modelo APACHE II y tenemos peores resultados tanto en RL como en RNA. Esto apoya la hipótesis de que la información aportada por las variables APACHE II en estos pacientes no es suficiente (analizada de forma estadística o neuronal) para poder asignarles una probabilidad de muerte precisa, lo que implica que son necesarios modelos específicos, con otras variables, para conseguir este objetivo en los pacientes traumáticos.
Las limitaciones de nuestro trabajo se centran primero en el tamaño de nuestra serie. Sólo los estudios multicéntricos pueden conseguir series grandes y comprobar la validez externa de los modelos, pero entonces incorporan el sesgo de analizar datos de diversas unidades. Hemos utilizado un modelo de red basado en el perceptrón multicapa entrenado con algoritmo de backpropagation, pero existen otros muchos tipos de red que podrían conseguir una mejor precisión en los resultados. El empirismo que envuelve al proceso de construcción y entrenamiento de una red sigue siendo una limitación importante. El tiempo de cálculo -no hay que olvidar que los programas utilizados habitualmente son de emulación- se convierte en un problema, y aumenta cuando el proceso debe realizarse múltiples veces.
Queríamos comparar modelos que utilizaran las mismas variables, por eso hemos trabajado con los que no incluyen la clasificación por grupos diagnósticos, ya que esto suponía aumentar su complejidad (se ampliaban más de 40 variables), lo que nos exigía un número de pacientes no disponible. La inclusión del ajuste por grupos diagnósticos en nuestro modelo APACHE II mejoraba la propiedad de discriminación, pero no alteraba su calibración.
En el modelo APACHE II las variables se categorizan según su desviación de la normalidad. En nuestros modelos basados en RL y RNA, los resultados se han calculado con las variables sin categorizar, aunque realizamos diversas pruebas con las variables categorizadas y encontramos resultados similares.
En resumen, el acercamiento a la metodología basada en redes neuronales artificiales puede hacerse con dos perspectivas. Por una parte, desde sus ventajas: las redes son capaces de trabajar sin las restricciones de los modelos estadísticos detectando las relaciones no lineales y las interacciones entre las variables predictoras. Y por otra, asumiendo sus desventajas: mayor complejidad de interpretación de sus parámetros de funcionamiento, mayor necesidad de recursos informáticos, alto componente empírico en su construcción y mayor dificultad de exportar el modelo para aplicarlo a otras poblaciones. Estas desventajas influyen en una menor difusión de la técnica para su uso habitual.
En general, una RNA es potencialmente más precisa que las técnicas estadísticas cuando la variable pronóstica se expresa como una función compleja de las variables predictoras o cuando existe interdependencia entre éstas; pero son estructuralmente complicadas y sus parámetros son más difíciles de interpretar. Por otra parte, la RL es una técnica más difundida y es más fácil interpretar sus coeficientes; aunque no será capaz de evaluar interacciones complejas entre las variables si no son especificadas en el modelo34.
Como principal conclusión podemos afirmar que la metodología basada en RNA es útil para la estratificación del riesgo de mortalidad hospitalaria. También debemos insistir en la distinta asignación de probabilidad entre las redes neuronales y la regresión logística y en la importancia del análisis por subgrupos que detecta los problemas de los sistemas de predicción. En un futuro cercano muchos de los sistemas de estratificación de riesgo acabarán enfrentados con distintos tipos de redes, pero este enfrentamiento no debe llevarnos a descartar una técnica por a otra, sino que deben complementarse y ayudarnos a comprender la relación entre las distintas variables predictoras de riesgo mejorando los modelos para hacerlos más precisos.
Agradecimientos
La financiación del proyecto se llevó a cabo mediante una beca FIS (00/0235).
Bibliografía
1. Librero J, Ordiñana R, Peiró S. Análisis automatizado de la calidad del conjunto mínimo de datos básicos. Implicaciones para los sistemas de ajuste de riesgos. Gac Sanit 1998;12:9-21. [ Links ]
2. Díaz C, Martínez D, Salcedo I, Masa J, De Irala J, Fernández-Crehuet R. Influencia de la infección nosocomial sobre la mortalidad en una unidad de cuidados intensivos. Gac Sanit 1998;12:23-8. [ Links ]
3. Armoni A. Use of neural networks in medical diagnosis. MD Computing 1998;15:100-4. [ Links ]
4. Sargent DJ. Comparison of artificial neural networks with other statistical approaches. Results from medical data sets. Cancer 2001;91:1636-42. [ Links ]
5. Dayhoff JE, DeLeo JM. Artificial neural networks. Opening the black box. Cancer 2001;91:1615-35. [ Links ]
6. Baxt WG. Application of artificial neural networks to clinical medicine. Lancet 1995;346:1135-8. [ Links ]
7. Axelson D, Bakken IJ, Susann I, Ehrnholm B, Nilsen G, Aasly J. Applications of neural network analyses to in vivo 1H magnetic resonance spectroscopy of Parkinson disease patients. J Magn Reson Imaging 2002;16:13-20. [ Links ]
8. Ohlsson M, Ohlin H, Wallerstedt SM, Edembrandt L. Usefulness of serial electrocardiograms for diagnosis of acute myocardial infarction. Am J Cardiol 2001;88:478-81. [ Links ]
9. Yamamura S, Nishizawa K, Hirano M, Momose Y, Kimura A. Prediction of plasma levels of aminoglycoside antibiotic in patients with severe illness by means of an artificial neural network simulator. J Pharm Sci 1998;1:95-101. [ Links ]
10. Coulter DM, Bate A, Meyboom RH, Lindquist M, Edwards IR. Antipsychotic drugs and heart muscle disorder in international pharmacovigilance: data mining study. BMJ 2001;322:1207-9. [ Links ]
11. Lippmann RP, Shahian DM. Coronary artery bypass risk prediction using neural networks. Ann Thorac Surg 1997;63:1635-43. [ Links ]
12. Baxt WG, Shofer FS, Sites FD, Hollander JE. A neural network aid for the early diagnosis of cardiac ischemia in patients presenting to the emergency department with chest pain. Ann Emerg Med 2002;40:575-83. [ Links ]
13. Levine RF. Conference concluding remarks. Cancer 2001;91:1696-7. [ Links ]
14. Knaus WA, Draper EA, Wagner DP, Zimmerman JE. APACHE II: A severity of disease classification system. Crit Care Med 1985;13:818-29. [ Links ]
15. Tu JV. Advantages and disadvantages of using artificial neural networks versus logistic regression for predicting medical outcomes. J Clin Epidemiol 1996;49;11:1225-31. [ Links ]
16. Tourassi GD, Floyd CE. The effect of data sampling on the performance evaluation of artificial neural networks in medical diagnosis. Medical Decision Making 1997;17:186-192. [ Links ]
17. Llorca J, Dierssen T. Comparación de dos métodos para el cálculo de la incertidumbre en los análisis de laboratorio. Gac Sanit 2000;14:458-63. [ Links ]
18. Cross BS, Harrison RF, Kennedy RL. Introduction to neural networks. Lancet 1995;346:1075-9. [ Links ]
19. Neural Network FAQ (Sarle WS) [consultado 5/05/2002]. Disponible en: ftp://ftp.sas.com/pub/neural/FAQ.html
20. Hanley JA, McNeil BJ. The meaning and use of the area under a receiver operating characteristic (ROC) curve. Radiology 1982;143:29-36. [ Links ]
21. Hanley JA, McNeil BJ. A method of comparing the areas under receiver operating characteristic curves derived from the same cases. Radiology 1983;148:839-43. [ Links ]
22. Lemeshow S, Hosmer DW. A review of goodness of fit statistics for use in the development of logistic regression models. Am J Epidemiol 1982;115:92-106. [ Links ]
23. Rapoport J, Teres D, Lemeshow S, Gehlbach S. A method for assessing the clinical performance and cost-effectiveness of intensive care units: a multicenter inception cohort study. Crit Care Med 1994;22:1385-91. [ Links ]
24. Bland JM, Altman DG. Statistical methods for assessing agreement between two methods of clinical measurement. Lancet 1986;1:307-10. [ Links ]
25. Abizanda R, Balerdi B, Lopez J, Valle FX, Jorda R, Ayestaran I, Rubert C. Fallos de predicción de resultados mediante APACHE II. Análisis de los errores de predicción demortalidad en pacientes críticos. Med Clin (Barc) 1994;102: 527-31. [ Links ]
26. Qnet (Vesta Services Inc.) ) [consultado 5/05/2002]. Disponible en: /www.qnetv2k.com/qnet2000information.htm
27. Wong LSS, Young JD. A comparison of ICU mortality prediction using the APACHE II scoring system and artificial neural network. Anaesthesia 1999;54:1048-54. [ Links ]
28. Clermont G, Angus DC, DiRusso SM, Griffin M, Linde-Zwirble WT. Predicting hospital mortality for patients in the intensive care unit: A comparison of artificial neural networks with logistic regression models. Crit Care Med 2001;29:291-6. [ Links ]
29. Sargent DJ. Comparison of artificial neural networks with other statistical approaches. Results from medical data sets. Cancer 2001;91:1636-42. [ Links ]
30. Martin B, Sanz A. Redes neuronales y sistemas borrosos. Zaragoza: Editorial Ra-Ma; 1997. [ Links ]
31. Ciampi A, Zhang F. A new approach to training back-propagation artificial neural networks: empirical evaluation on ten data sets from clinical studies. Statist Med 2002;21:1309-30. [ Links ]
32. Marik PE, Varon J. Severity scoring and outcome assessment. Computerized predictive models and scoring systems. Crit Care Clin 1999;15:633-46. [ Links ]
33. Cho DY, Wang YC. Comparison of the APACHE III, APACHE II and Glasgow coma scale in acute head injury for prediction of mortality and functional outcome. Intensive Care Med 1997;23:77-84. [ Links ]
34. Muckart DJJ, Bhagwanjee S, Gouws E. Validation of an outcome prediction model for critically ill trauma patients without head injury. J Trauma 1997;43:934-9. [ Links ]
35. Álvarez M, Nava JM, Rue M, Quintana S. Mortality prediction in head trauma patients: Performance of Glasgow Coma Score and general severity systems. Crit Care Med 1998;26:142-8. [ Links ]
36. Liestol K, Anderesen PK, Andersen U. Survival analysis and neural nets. Statist Med 1994;13:1189-200. [ Links ]