My SciELO
Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
 Cited by Google
Cited by Google -
 Similars in
SciELO
Similars in
SciELO  Similars in Google
Similars in Google
Share
Revista Española de Enfermedades Digestivas
Print version ISSN 1130-0108
Rev. esp. enferm. dig. vol.97 n.3 Madrid Mar. 2005
| TRABAJOS ORIGINALES |
Sobreexpresión de c-myc y pérdida de heterozigosidad en 2p, 3p, 5q, 17p y 18q en
carcinoma colorrectal esporádico
A. Sánchez-Pernaute, E. Pérez-Aguirre, F.J. Cerdán, P. Iniesta1, L. Díez Valladares, C. de Juan1,
A. Morán1, A. García-Botella,
C. García Aranda1, M. Benito1, A.J. Torres y J.L. Balibrea
Departamento de Cirugía. Hospital Universitario San Carlos. Universidad Complutense de Madrid. 1Departamento de Biología Molecular
y Bioquímica. Facultad de Farmacia. Universidad Complutense de Madrid
RESUMEN
Objetivo: el objetivo del presente trabajo es evaluar la importancia pronóstica de la pérdida de heterozigosidad en las regiones 2p, 3p, 5q, 17p y 18q y de la sobreexpresión del gen c-myc en el carcinoma colorrectal esporádico, mediante el estudio de la supervivencia libre de enfermedad tras cirugía potencialmente curativa.
Métodos: se han analizado muestras tumorales y no tumorales de mucosa colónica de 153 pacientes. El 51% de los pacientes eran varones y la edad media de la serie fue 67 años. Los tumores fueron proximales en 37 casos, distales en 37 y localizados en recto en 79. Se analizó la sobreexpresión del RNA de c-myc por Northern blot, y la presencia de pérdida de heterozigosidad en las diferentes regiones consideradas por análisis de microsatélites.
Resultados: se detectó sobreexpresión de c-myc en el 25% de los casos, y pérdida de heterozigosidad en alguna de las regiones estudiadas en el 48%. No hubo asociación entre las variables clínicas o patológicas y las alteraciones génicas. Se encontró un intervalo libre de enfermedad más corto para los pacientes con pérdida de heterozigosidad y sobreexpresión de c-myc, y este factor tuvo valor pronóstico independiente en el análisis multivariante (RR: 4.34, p < 0,0001).
Conclusiones: la coexistencia de pérdida de heterozigosidad y sobreexpresión del oncogén c-myc distingue un grupo de pacientes con recurrencia más precoz tras cirugía curativa por carcinoma
colorrectal.
Palabras clave: Pérdida de heterozigosidad. c-myc. Oncogenes. Pronóstico. Carcinoma colorrectal.
INTRODUCCIÓN
El carcinoma colorrectal (CCR) es uno de los tumores sólidos más frecuentemente diagnosticados. Es, en el mundo occidental, la segunda causa de muerte por cáncer (1,2), causando en España el 11% de las defunciones por cáncer en hombres y el 15% en mujeres (3).
La elevada incidencia del CCR y la facilidad para obtener muestras en distintas etapas evolutivas han permitido describir un modelo de progresión tumoral, desde los pólipos benignos hasta los cánceres invasivos (4), y los acontecimientos genéticos que acompañan a esta transformación neoplásica, como son la deleción de APC, la hipometilación del DNA, las mutaciones en k-ras, la pérdida alélica en DCC o las alteraciones de p53 (4,5).
La expresión de unas u otras alteraciones génicas parece acompañar a fenotipos tumorales más o menos agresivos, y su reconocimiento puede sentar las bases para decidir la aplicación de tratamientos adyuvantes, que hoy solamente se administran en función del único factor pronóstico aceptado universalmente, el estadio tumoral.
En el presente trabajo se ha estudiado la sobreexpresión del gen c-myc, un oncogén dominante con capacidad de comportarse también como gen supresor, y la pérdida de heterozigosidad en distintas regiones cromosómicas, en una serie de pacientes tratados por CCR esporádico, y la influencia de estas alteraciones sobre la recurrencia de la enfermedad.
MATERIAL Y MÉTODOS
Pacientes
Se incluyeron 153 pacientes, 78 varones y 75 mujeres, intervenidos por CCR esporádico. Fueron incluidos prospectivamente todos aquellos pacientes intervenidos de forma programada de quienes se pudo obtener tejido tumoral y no tumoral en fresco para el estudio genético. La edad media fue 67 años (31-89). Dieciocho enfermos se encontraban en estadio A de Dukes, 61 en estadio B, 47 en estadio C y 27 en estadio D. Treinta y siete tumores se localizaron en colon proximal, 37 en colon distal y 79 en recto.
Tres pacientes (2%) fallecieron en el postoperatorio y fueron excluidos para el análisis de supervivencia.
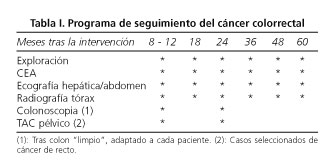
Los pacientes en estadio B o superior fueron tratados con quimioterapia siempre que no existiera contraindicación médica. Posteriormente iniciaron un programa de seguimiento según un protocolo publicado previamente (6) (Tabla I).
La sospecha de recidiva motivó el intento de confirmación citológica o histológica, pero la imagen radiológica con o sin elevación de los marcadores tumorales fue considerada suficiente para el diagnóstico de recurrencia.
Toma de muestras. Inmediatamente tras la resección de la pieza se procedió a su apertura y lavado. Se tomaron muestras de tejido tumoral y de mucosa colónica macroscópicamente normal. Las muestras tisulares se congelaron en nitrógeno líquido y se etiquetaron y archivaron en un congelador a -80 ºC hasta su posterior análisis. Todos los pacientes dieron su consentimiento para la utilización de sus tejidos y el estudio fue aprobado por el Comité Ético de Investigación Clínica del Hospital.
Sobreexpresión de c-myc. Se determinó la sobreexpresión de c-myc por medio de análisis Northern blot. Los tejidos se homogeneizaron en una solución congelada de tiocianato de guanidinio. El método empleado para la extracción de RNA fue el descrito por Chomczynski y Sacchi (7), y posteriormete se cuantificó por espectrofotometría ultravioleta. Se desnaturalizaron 30 mg de RNA total en formamida al 50%, formaldehído 2,2 M, 20 mM de MOPS (ácido 3- [N-morfolino] propanosulfónico) (pH 7,0) y glicerol al 6% a 65 ºC durante 15'; posteriormente las muestras de RNA total se resolvieron en geles de agarosa al 0,9% que contenían formaldehído 0,6 M, y finalmente se transfirieron a membranas GeneScreen (NEN Research Products, Dupont, Boston, MA). La adhesión del RNA a la membrana se demostró mediante luz ultravioleta. La membrana se prehibridó durante 3 horas y posteriormente se hibridó durante 24 horas a 42 ºC en un tampón conteniendo Na2PO4 0,25 M (pH 7.2), NaCl 0,25 M, 1 mM de EDTA, 100 μg/ml de DNA desnaturalizado de esperma de salmón, SDS (dodecil-sulfato sódico) al 7%, formamida desionizada al 50% y la sonda de c-myc marcada con32P) dCTP. Posteriormente las membranas se lavaron y expusieron a películas Kodak X-OMAT AR. Se realizó cuantificación densitométrica de las autorradiografías en un densitómetro láser (Molecular Dinamics, Sunnyvale, CA). Para la normalización del RNA se utilizó la sonda ribosomal 18S. Los resultados se obtuvieron comparando la señal de c-myc en el tejido tumoral con la del tejido no tumoral.
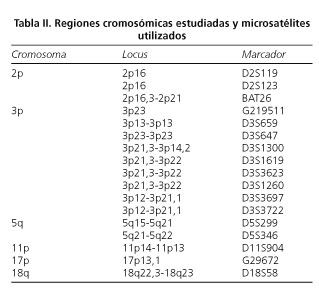
Pérdida de heterozigosidad. Se procedió a la extracción de DNA por el método descrito por Blin y Stafford (8). Se estudió la pérdida de heterozigosidad en los loci expuestos en la tabla II. Las reacciones en cadena de la polimerasa (PCRs) se llevaron a cabo en un Thermocycler Perkin Elmer (Gene Amp PCR System 2400) en un volumen de 20 μl conteniendo: 50 ng de DNA genómico, 1 μM de cada primer, 0,2 mM de cada deoxinucleósido trifosfato, 10 mM Tris HCI (pH 8.3), 50 mM de KCl, 2 mM de MgCl2, 0,5 μl de (α32p) dCTP (3000 Ci/mmol) (Amersham, Buckinghamshire, UK) y 1 U de DNA polimerasa (Dynazyme Thermostable, Finnzymes OY, Helsinki, Finlandia). Las condiciones de PCR fueron: desnaturalización inicial a 94 ºC durante 5 minutos, 30 ciclos de 94 ºC durante 0,5 minutos, 55 ºC durante 0,5 minutos y 72 ºC durante 1 minuto. La extensión final fue 72 ºC durante 7 minutos. Después los productos PCR se desnaturalizaron por medio de formamida al 95% y se realizó una electroforesis en gel de poliacrilamida con urea 7 M durante 3 horas a 43 W, seguida de autorradiografiado. Se consideró pérdida de heterozigosidad cuando apareció pérdida de al menos un 50% de uno de los dos alelos del locus.
Análisis estadístico. Se compararon los resultados clínicos y patológicos con los datos moleculares por medio de las pruebas de
χ2 o de Fisher para las variables cualitativas, y t de Student para las cuantitativas. Se estudió la supervivencia libre de enfermedad mediante curvas de Kaplan Meier y la prueba de log rank. Las variables con significación pronóstica se incluyeron en un modelo de riesgos proporcionales de Cox. Todos los cálculos se realizaron con el programa SPSS versión 10.0 para Windows (SPSS Inc, Chicago, IL, licencia nº: 7269515).
RESULTADOS
En 39 casos se encontró sobreexpresión de c-myc (25,5%), sin relación con el sexo, la localización tumoral, el grado de diferenciación o el estadio. Los pacientes con tumores con sobreexpresión de c-myc fueron significativamente más jóvenes, 64,2 versus 68,6 años, p = 0,03.
Se pudo realizar el estudio de pérdidas de heterozigosidad (loh) en 143 muestras; en 69 (48,3%) se detectó loh en al menos una de las regiones estudiadas. No se encontró relación entre esta y ninguna de las variables estudiadas, aunque se observó una asociación no estadística entre loh y grado de diferenciación: entre los tumores bien diferenciados el porcentaje de loh fue 32%, entre los medianamente diferenciados 51% y entre los pobremente diferenciados 57% (p = 0,09).
De los 143 pacientes en los que se estudiaron las dos alteraciones genéticas, 57 (39,9%) fueron negativos para ambas, y 20 (14%) positivos para las dos.
Estudios de supervivencia
El seguimiento medio fue de 28 meses, con un rango de 0 a 115. Se detectaron 35 recidivas tumorales (22,9%). La supervivencia libre de enfermedad para los estadios A-C fue 69% a 5 años. La sobreexpresión de c-myc se asoció con una menor supervivencia libre de enfermedad a 5 años, principalmente en el estadio B (c-myc +: 58% vs. c-myc -: 86%), aunque no se alcanzó significación estadística.
La loh fue un parámetro de mal pronóstico; la supervivencia estimada libre de enfermedad fue del 56% a 5 años para los pacientes con pérdida alélica, y del 75% para los enfermos sin ella (p = 0,07); las diferencias fueron más acusadas en el estadio B (68 vs. 92%, p < 0,05), donde sí hubo significación estadística.
Con el fin de determinar la influencia pronóstica de la conjunción de ambas alteraciones genéticas, se subdividió la serie en cuatro grupos: pacientes sin alteraciones detectables, pacientes con sobreexpresión de c-myc sin loh, pacientes con loh sin alteración en c-myc y pacientes portadores de ambas alteraciones. El análisis de la supervivencia libre de enfermedad demostró un significativo peor pronóstico para este último grupo, los enfermos con sobreexpresión de c-myc y loh, entre los que solamente el 33% se encontraban sin enfermedad a los 5 años (p = 0,002) (Fig. 1).
Para estudiar la independencia pronóstica se comparó la asociación de alteraciones genéticas con parámetros pronóstico "clásicos", como el estadio de Dukes y la localización tumoral. En el análisis multivariante los tres demostraron ser factores pronóstico independientes, siendo el riesgo relativo de recurrencia 3,11 para los tumores en estadio C, 3,35 para los tumores localizados en el recto y 4,34 para los tumores con loh y sobreexpresión de c-myc (Tabla III).
DISCUSIÓN
El oncogén c-myc aparece sobreexpresado o amplificado en la tercera parte de los CCR (9,10), y hasta en un 65% cuando se estudia su proteína por inmunohistoquímica (11). Las diferencias entre el análisis del DNA o RNA y el estudio de la proteína las explica el que las dos primeras técnicas solamente evalúan el incremento de la síntesis, mientras que la tercera incluye la disminución de la degradación. No está claro si la acumulación de c-myc se produce más en etapas tumorales más avanzadas, como comunican Masramon (9) y Heerdt (12), o si como encontramos nosotros la sobreexpresión del gen se produce desde etapas tempranas, siendo independiente del estadio tumoral (11,13). Lo que sin embargo sí parece claro, aunque nosotros no lo confirmamos, es que acompaña principalmente a los tumores de localización más distal, mientras que los proximales se caracterizarían por la presencia de otras alteraciones genéticas, como es la inestabilidad de microsatélites (14,15).
La loh se ha comunicado hasta en un 80% de los CCR. Con este término nos referimos a la alteración detectada por la pérdida parcial o total de uno de los dos alelos de los loci estudiados, y distinguimos así el genotipo loh + (loss of heterozigosity, pérdida de heterozigosidad) que parece caracterizar a un fenotipo distinto de cáncer de colon (16). En nuestro estudio se han investigado todas las regiones cromosómicas que se suelen involucrar en el CCR, es decir 2p, 3p, 5q, 17p y 18q, aquellas donde se encuentran genes supresores de tumores (APC, p53, DCC) o se supone su existencia (2p, 3p). En la literatura se comunica la loh en tumores más avanzados, como si existiera una pérdida progresiva de material genético durante la progresión tumoral (17); en nuestra serie la prevalencia es ligeramente inferior al 50%, y no la encontramos asociada con el estadio tumoral ni con la localización de la lesión (18).
El valor pronóstico de la sobreexpresión de c-myc en el CCR se ha estudiado de múltiples maneras, pero ni la amplificación de DNA (19), ni la sobreexpresión de RNA (20,21), ni el análisis inmunohistoquímico de la proteína (11,13) han podido esclarecer el papel que desempeña el gen en la evolución de la enfermedad. Es de destacar el trabajo publicado por Augenlicht (19), en el que se demuestra cómo los pacientes con amplificación génica responden mejor al tratamiento con cirugía y un régimen quimioterápico de levamisol y 5-fluorouracilo; aunque el número de enfermos es pequeño y no existe independencia pronóstica, el estudio constituye una buena base para profundizar la investigación sobre la relación entre la expresión de c-myc y la respuesta a la quimioterapia. Bhatavdekar (11) sí encuentra diferencias pronósticas en función de la expresión de la proteína c-myc en una corta serie de enfermos intervenidos en estadio B y C, con una mayor significación cuando se consideran conjuntamente estas alteraciones con las de las proteínas Bcl-2 y p53, y ya estos autores postulan que la importancia pronóstica de estos genes o proteínas radica en la cooperación entre ellos. Lo mismo se deduce del trabajo de Smith y Goh (21), que encuentran un distinto pronóstico, en este caso mejor, para el grupo de pacientes con sobreexpresión de c-myc siempre que coexista la expresión de "wild type" p53.
Las deleciones cromosómicas suelen afectar, de forma individual, negativamente al pronóstico del CCR. Nuestro grupo comunicó recientemente la correlación entre la loh en 3p23 y una menor supervivencia (22). En cuanto a 17p, la influencia pronóstica es variable (23), encontrándose incluso publicaciones que confieren valor protector a las alteraciones de p53 (24,25). Las pérdidas alélicas en 18q se relacionan normalmente con una peor evolución, como encontraron el grupo de Vogelstein (26) o en nuestro país el de Rosell (27); en sendos trabajos, la loh en 18q se comportó como variable pronóstica independiente. De igual manera la loh en 5q (APC) se comporta generalmente como variable pronóstica negativa (28).
Tomadas en conjunto las loh, es decir, considerando como genotipo loh + aquellos casos en que se presente al menos una región cromosómica con pérdida alélica, encontramos un peor pronóstico no significativo para los pacientes portadores de la anomalía, igual que ha sido publicado por nuestro grupo con anterioridad (29) y por otros autores (17). Si bien no se alcanza la significación estadística, lo mismo que ocurre con las alteraciones de c-myc, al analizar el estadio B las diferencias son casi significativas en el caso de c-myc y claramente significativas en el del genotipo loh. Este hecho es muy importante, ya que los pacientes en estadio B constituyen el grupo en que de forma global no se obtiene claramente un beneficio con el tratamiento adyuvante. Mediante el estudio genético se podrían distinguir los pacientes que van a tener una peor evolución tras cirugía potencialmente curativa, y serían ellos los que recibirían quimioterapia postoperatoria.
La asociación de ambas alteraciones es un acontecimiento genético de claro valor pronóstico negativo, de manera independiente frente a otros parámetros tradicionales, y que concede un riesgo relativo de recidiva elevado. ¿Cómo se relacionan ambos acontecimientos génicos? El gen c-myc fue descrito originalmente como un gen activable tras estímulos mutagénicos, dependiente de la expresión de factores de crecimiento y con una actividad reguladora positiva del crecimiento celular. Su presencia parece ser esencial para la progresión de Gl a S y también regula la diferenciación celular y promueve la transformación.
Recientemente se ha descubierto su capacidad de funcionamiento como gen supresor tumoral, al ser capaz de inducir la muerte celular por apoptosis. La dualidad de función dependerá del medio ambiente celular y del estado de los genes diana sobre los que actúa (30). En lo que se refiere a los mecanismos de cooperación, o a la relación entre c-myc y los genes supresores delecionados en el genotipo loh, alguno de ellos está, al menos parcialmente, dilucidado. El gen APC en 5q se relaciona con c-myc por la vía wnt, proteína de membrana implicada en la comunicación intercelular y en el crecimiento y diferenciación. La señalización a partir de wnt induce la liberación de complejo formado por APC y β-catenina, pudiendo entonces la β-catenina viajar al núcleo e inhibir la represión mantenida sobre c-myc por proteínas como Groucho o CBP. Así el estímulo del receptor wnt favorece la proliferación y la transformación celular mediada por c-myc y este mecanismo se reproduce de la misma manera al existir una mutación en APC o una deleción en 5q, y que entonces la b-catenina puede ejercer su acción (31-33). Por otra parte, la cooperación con p53 parece desarrollarse a través de la vía opuesta, el control negativo del crecimiento o el comportamiento como inductor de la apoptosis y por tanto como gen supresor tumoral. Parece que la proteína p53 "wild type" participa en la apoptosis mediada por Fas e inducida por c-myc (34). Según esto la presencia de mutaciones génicas o deleciones en 17p causaría una inhibición de la apoptosis mediada por c-myc, favoreciéndose así la transformación celular y el desarrollo neoplásico. Probablemente mecanismos similares, sean de sentido negativo o positivo, imbrican a los distintos genes que caracterizan el genotipo loh, responsabilizándose así de la peor evolución de los enfermos portadores de estas alteraciones.
BIBLIOGRAFÍA
1. Greenle RT, Murray T, Bolden S, Wingo PA. Cancer statistics, 2000. CA Cancer J Clin 2000; 50: 7-33. [ Links ]
2. Cohen AM, Minsky BD, Schilsky RL. Cancer of the colon. In: DeVita Jr VT, Hellman S, Rosenberg SA. Cancer. Principles and practice of oncology. 5th ed. Philadelphia: Lippincott - Raven Publishers, 1997. p. 1144-97. [ Links ]
3. Centro Nacional de Epidemiología. Área de Epidemiología Ambiental y Cáncer. Mortalidad por Cáncer en España, 2000. http://cne.isciii.es/cancer/mort2000.txt. [ Links ]
4. Fearon ER, Vogelstein B. A genetic model for colorectal carcinogenesis. Cell 1990; 61: 759-67. [ Links ]
5. Toribara NW, Slisenger MH. Screening for colorectal cancer. N Engl J Med 1995; 332: 861-7. [ Links ]
6. Cerdán FJ. Seguimiento de los pacientes intervenidos de cáncer colorrectal. Rev Cancer 1997; 11: 32-41. [ Links ]
7. Chomczynski P, Sacchi N. Single-step method of RNA isolation by acid guanidinium thiocyanate-phenol-chloroform extraction. Anal Biochem 1987; 162: 156-9. [ Links ]
8. Blin N, Stafford DW. A general method for isolation of high molecular weight DNA from eukaryotes. Nucleic Acid Res 1976; 3: 2303-8. [ Links ]
9. Masramon L, Arribas R, Tórtola S, Perucho M, Peinado MA. Moderate amplifications of the c-myc gene correlate with molecular and clinicopathological parameters in colorectal cancer. Br J Cancer 1998; 99: 2349-56. [ Links ]
10. Arango D, Corner GA, Wadler S, Catalano PJ, Augenlicht LH. C-myc/p53 interaction determines sensitivity of human colon carcinoma cells to 5-fluorouracil in vitro and in vivo. Cancer Res 2001; 61: 4910-5. [ Links ]
11. Bhatavdekar JM, Patel DD, Ghosh N, Chikhlikar PR, Trivedi TI, Suthar TP, et al. Coexpression of Bcl-2, c-Myc and p53 oncoproteins as prognostic discriminants in patients with colorectal carcinoma. Dis Colon Rectum 1997; 40: 785-90. [ Links ]
12. Heerdt BG, Molinas S, Deitch D, Augenlicht LH. Aggressive subtypes of human colorectal tumors frequently exhibit amplification of the c-myc gene. Oncogene 1991; 6: 125-9. [ Links ]
13. Miller F, Heimann TM, Quish A, Pyo DJ, Szporn A, Martinelli G, et al. Ras and c-myc protein expression in colorectal carcinoma. Study of cancer prone patients. Dis Colon Rectum 1992; 35: 430-5. [ Links ]
14. Rothberg G, Spandorfer JM, Erisman MD, Staroscik RN, Sears HF, Petersen RO, et al. Evidence that c-myc expression defines two genetically distinct forms of colorectal adenocarcinoma. Br J Cancer 1985; 52: 629-32. [ Links ]
15. Bufill JA. Colorectal cancer: Evidence for distinct genetic categories based on proximal or distant tumor location. Ann lntern Med 1990; 113: 779-88. [ Links ]
16. Goel A, Arnold CN, Niedzwiecki D, Chang DK, Ricciardiello L, Carethers JM, et al. Characterization of sporadic colon cancer by patterns of genomic instability. Cancer Res 2003; 63: 1608-14. [ Links ]
17. Weber JC, Schneider A, Rohr S, Nakano H, Bachellier P, Méchine A, et al. Analysis of allelic imbalance in patients with colorectal cancer according to stage and presence of synchronous liver metastases. Ann Surg 2001; 234: 795-803. [ Links ]
18. García-Hisrschfeld García J, Blanes Berenguel A, Vicioso Recio L, Márquez Moreno A, Rubio Garrido J, Matilla Vicente A. Colon cancer: p53 expression and DNA ploidy. Their relation to proximal or distal tumor site. Rev Esp Enferm Dig 1999; 91: 481-8. [ Links ]
19. Augenlicht LH, Wadler S, Corner G, Richards C, Ryan L, Multani AS, et al. Low-level c-myc amplifications in human colonic carcinoma cell lines and tumours: a frequent, p53-independent mutation associated with improved outcome in a randomized multi-institutional trial. Cancer Res 1997; 57: 1769-75. [ Links ]
20. Nagai MA, Habr-Gama A, Oshima CT, Brentani MM. Association of genetic alterations of c-myc , c-fos, and c-Ha-ras proto-oncogenes in colorectal tumors. Frequency and clinical significance. Dis Colon Rectum 1992; 35: 444-51. [ Links ]
21. Smith DR, Goh HS. Overexpression of the c-myc proto-oncogene in colorectal carcinoma is associated with a reduced mortality that is abrogated by point mutation of the p53 tumor suppressor gene. Clin Cancer Res 1996; 2: 1049-53. [ Links ]
22. lniesta P, Massa MJ, González-Quevedo R, de Juan C, Morán A, Sánchez-Pernaute A, et al. Loss of heterozygosity at 3p23 is correlated with poor survival in patients with colorectal carcinoma. Cancer 2000; 89: 1220-7. [ Links ]
23. Petersen S, Thames HD, Nieder C, Petersen C, Baumann M. The results of colorectal cancer treatment by p53 status: treatment-specific overview. Dis Colon Rectum 2001; 44: 322-34. [ Links ]
24. Soong R, Grieu F, Robbins P, Dix B, Chen D, Parsons R, et al. p53 alterations are associated with improved prognosis in distal colonic carcinomas. Clin Cancer Res 1997; 3: 1405-11. [ Links ]
25. Ahnen DJ, Feigi P, Quan G, Fenoglio-Preiser C, Lovato LC, Bunn Jr PA, et al. Ki-ras mutations and p53 overexpression predict the clinical behavior of colorectal cancer: a Southwest Oncology Group Study. Cancer Res 1998; 58: 1149-58. [ Links ]
26. Jen J, Kim H, Piantadosi S, Liu Z-F, Levitt RC, Sistonen P, et al. Allelic loss of chromosome 18q and prognosis in colorectal cancer. N Engl J Med 1994; 331: 213-21. [ Links ]
27. Martínez-López E, Abad A, Font A, Monzó M, Ojanguren I, Pifarré A, et al. Allelic loss on chromosome 18q as a prognostic marker in stage II colorectal cancer. Gastroenterology 1998; 114: 1180-7. [ Links ]
28. McLeod HL, Murray GI. Tumour markers of prognosis in colorectal cancer. Br J Cancer 1999; 79: 191-203. [ Links ]
29. Massa MJ, lniesta P, González Quevedo R, de Juan C, Caldés T, Sánchez- Pernaute A, et al. Differential prognosis of replication error phenotype and loss of heterozigosity in sporadic colorectal cancer. Eur J Cancer 1999; 35: 1676-82. [ Links ]
30. Packham G, Cleveland JL. c-Myc and apoptosis. Biochem Biophys Acta 1995; 1242: 11-28. [ Links ]
31. Pennisi E. How a growth control path takes a wrong turn to cancer. Science 1998; 281: 1438-41. [ Links ]
32. He T-C, Sparks AB, Rago C, Hermeking H, Zawel L, da Costa LT, et al. Identification of C-MYC as a target of the APC pathway. Science 1998; 281: 1509-12. [ Links ]
33. Seidler HBK, Utsuyama M, Nagaoka S, Takemura T, Kitagawa M, Hirokawa K. Expression level of Wnt signaling components possibly influences the biological behavior of colorectal cancer in different age groups. Exp Mol Pathol 2004; 76: 224-33. [ Links ]
34. Green DR. A Myc-induced apoptosis pathway surfaces. Science 1997; 278: 1246-7. [ Links ]











 text in
text in