Mi SciELO
Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO -
 Accesos
Accesos
Links relacionados
-
 Citado por Google
Citado por Google -
 Similares en
SciELO
Similares en
SciELO -
 Similares en Google
Similares en Google
Compartir
Revista Española de Salud Pública
versión On-line ISSN 2173-9110versión impresa ISSN 1135-5727
Rev. Esp. Salud Publica vol.84 no.5 Madrid sep./oct. 2010
Gripe pandémica H1N1 (2009). Experiencia de la Red de Laboratorios de Gripe del Sistema de Vigilancia de la Gripe en España (SVGE)
Pandemic Influenza A(H1N1). The experience of the Spanish Laboratories of Influenza Network (ReLEG)
María Teresa Cuevas González-Nicolás, Juan Ledesma Moreno, Francisco Pozo Sánchez, Inmaculada Casas Flecha y Pilar Pérez-Breña.
Laboratorio de Gripe y Virus Respiratorios. Centro Nacional de Microbiología. Instituto de Salud Carlos III. Madrid.
Dirección para correspondencia
RESUMEN
Existen tres tipos de virus de la gripe: A, B y C. Estos virus evolucionan constantemente debido a que presentan dos características principales, la primera es la falta de capacidad correctora de la polimerasa viral que hace que se acumulen mutaciones puntuales en sus genes (deriva antigénica), y la segunda la naturaleza de su genoma formando por ocho segmentos lo que le permite el intercambio de genes entre distintos virus (salto antigénico). Esta plasticidad viral ha permitido que los virus de la gripe A sean capaces de adaptarse a diferentes hospedadores y adquirir capacidades pandémicas.
El sistema de vigilancia de la gripe en España (SVGE) surgió como respuesta a la preocupación de que se produjera una pandemia, máxime después de los casos de gripe aviar detectados en el ser humano. Este sistema de vigilancia esta formado por dieciséis redes de médicos generales y pediatras centinela y diecinueve servicios de epidemiología, coordinados por el Centro Nacional de Epidemiología (CNE) y una red de dieciocho laboratorios, la red de laboratorios de Españoles de Gripe (ReLEG), coordinados por el Centro Nacional de Microbiología (CNM).
El objetivo de este artículo es presentar la actuación de la ReLEG durante la pandemia producida por el virus de la gripe (H1N1)2009, durante la temporada 2009-2010. La función principal de la red es la vigilancia de los virus circulantes mediante su detección y posterior caracterización genética y antigénica, incluyendo la detección de las mutaciones de resistencia que afectan a los fármacos en uso, principalmente el Oseltamivir.
Palabras clave: Virología. Pandemia. Subtipo H1N1 del Virus de la Influenza A. Gripe humana. España.
ABSTRACT
There are three types of influenza viruses: A, B, C. These viruses evolves constantly due to two main characteristics: the first one is the lack of the correction ability of the viral polymerase which causes the accumulation of single nucleotide mutations in the viral genes introduced by an error-prone viral RNA polymerase, (antigenic shift). The second one is the nature of their genome, formed by eight segments, which allows the interchange of genes between two different viral strains (antigenic drift). This viral plasticity, has allowed to the influenza A viruses to infect new host species and to cause infections with a pandemic characteristics.
The Spanish influenza surveillance system, SVGE (its Spanish acronym), arises as a response to the possibility of facing a pandemic situation, especially after the transmission of avian influenza viruses to humans. This surveillance system is formed by sixteen physician and paediatrics network, nineteen epidemiological services coordinated by the National Epidemiological Centre (CNE) and eighteen laboratories , the Spanish Laboratories of Influenza network (ReLEG), coordinated by the National Centre of Microbiology.
The aim of this article is to show the action of the ReLEG, in the pandemic caused by the influenza virus A(H1N1) during the season 2009-2010. The main objective of this network is the surveillance of the circulating viruses by means of their detection and their subsequent antigenic and genetic characterization, including the detection of resistance mutations against the main drugs, such as Oseltamivir.
Key words: Virology. Sentinel surveillance. Disease Outbreaks. Influenza A virus, H1N1 subtype. Grippe. Influenza, human. Spain.
El virus pandémico (H1N1) 2009
Los virus de la gripe, o virus influenza, se clasifican en tres tipos diferentes, A, B y C. Los del tipo A afectan a gran variedad de aves y mamíferos, entre ellos el ser humano. Estructuralmente (figura 1) el virus presenta una envuelta lipídica de donde sobresalen las glicoproteínas hemaglutinina (HA) y neuraminidasa (NA) que contienen los principales determinantes antigénicos virales. Una tercera proteína de membrana, la proteína matriz M2, forma el canal iónico transmembrana. Este conjunto o envuelta viral está recubierto por dentro por una capa formada por la proteína matriz M1, que encierra las «ribonucleoproteínas» que contienen el genoma viral y el complejo de la polimerasa (PB1, PB2 y PA) y que están estructuradas por la nucleoproteína (NP). El genoma está formado por ocho segmentos de ARN de cadena sencilla y polaridad negativa, con un tamaño de 13.600 nucleótidos y codifica 11 proteínas.
Los virus de la gripe evolucionan constantemente utilizando diferentes mecanismos. La falta de capacidad correctora de la polimerasa viral ocasiona gran número de mutaciones puntuales durante la replicación del genoma, lo que da lugar al fenómeno de deriva antigénica (antigenic drift). La alta tasa de mutación a la que están sometidas las proteínas HA y NA obliga cada año a revisar los virus que forman parte de las vacunas1. Otros mecanismos de evolución de los virus influenza se deben al hecho de poseer un genoma segmentado que facilita el intercambio genético o la recombinación genética, originando el llamado salto antigénico (antigenic shift). Hasta ahora se han descrito 16 subtipos de HA (H1-H16) y 9 subtipos de NA (N1-N9). La plasticidad de estos virus, que les permite incorporar pequeños y grandes cambios, es la base de su evolución constante y de su naturaleza de agente zoonótico con capacidad para adaptarse a nuevos hospedadores y llegar a adquirir capacidades pandémicas para el ser humano. Este tema siempre ha preocupado a los expertos y a las autoridades sanitarias, máxime a raíz de los casos humanos por gripe aviar detectados en los últimos años.
La estructura del nuevo virus pandémico (H1N1) 2009 se describe en2 según el esquema reproducido en la figura 2. Todos los segmentos del genoma tienen su origen en las aves desde las que durante diferentes años pasaron a los cerdos, excepto en el caso del segmento PB1 que tuvo un paso intermedio por el ser humano. Hacia 1918 se produjeron infecciones de cerdos por virus aviares que poseían los genes HA, NP y NS, que desde entonces se han mantenido en los virus del linaje porcino clásico o americano. Hacia finales de la década de los 90 se hizo enzoótico en la cabaña americana un virus en el que se identificó un triple reagrupamiento génico (triple reassortant) y que produjo algunos casos humanos3. Los nuevos virus detectados en California incorporan seis genes del virus con triple agrupamiento, mientras los genes NA y M corresponden al linaje porcino Euroasiático, que fue originado también por virus aviares que hacia 1979 infectaron las piaras de Europa.
Un estudio de evolución del nuevo virus (H1N1) 2009 basado en el análisis del genoma viral completo de 290 aislados obtenidos por secuenciación masiva4, indica que el virus se ha diversificado en al menos 7 clados o grupos en los 4 primeros meses de circulación. Se corresponden con modelos definidos que, excepto en el clado 4, estarían dispersos en distintos países y continentes, pudiendo co-circular en tiempo y espacio con aparente buena transmisibilidad en el hombre. En los distintos genes, en cada clado se identifica una serie de cambios de amino-ácidos, aunque ninguno de estos cambios se localiza en los sitios antigénicos de la subunidad HA1 de la HA, ni en posiciones asociadas con funciones importantes, como podrían ser el sitio de unión al receptor celular (que define el tropismo del virus por diferentes tejidos u hospedadores), o también las regiones dianas de los distintos antivirales.
La secuenciación total o parcial de HA y de NA de numerosos virus (H1N1) 2009 en todo el mundo está permitiendo seguir la aparición de mutaciones que pueden tener importancia biológica por su situación en los tipos de zonas antes mencionadas Se especula con que la mutación D222G, detectada en primer lugar en Noruega5, podría contribuir a un mayor neumotropismo del virus, aumentando por tanto la gravedad de la infección. Hacia finales de 2009 una evaluación de la OMS de los datos obtenidos en gran parte del mundo6 no pudo llegar a demostrar esta hipótesis.
Por otra parte, el estudio de la aparición y de la evolución de las resistencias a agentes antivirales presenta el máximo interés en los inicios de una pandemia ya que, en ausencia de nuevas vacunas, constituyen el único instrumento de control específico. En esta ocasión, se determinó desde el principio que el virus (H1N1) 2009 era resistente a los adamantanos, por lo que solo podrían emplearse drogas inhibidoras de la NA, como el Oseltamivir o el Zanamivir. La OMS pudo valorar hacia el final de 20097 los primeros 109 casos encontrados, lo que influyó en las recomendaciones para la utilización de antigripales.
Preparación pandémica
Las relaciones de los virus gripales con el ser humano han sido esquematizadas por la OMS en una serie de Fases que abarcan desde los brotes estacionales hasta las pandemias8. Las Fases 1-3 recogen los estadios en los que no se han detectado en el hombre infecciones por virus animales o solo se han detectado esporádicamente, o bien los virus no han alcanzado la transmisión eficaz al ser humano. En la Fase 4 se detecta transmisión persona-persona dando lugar a brotes a nivel comunitario. En la Fase 5 los virus ya se han propagado al menos en dos países de una misma región de la OMS. En la Fase 6 se considera que existe una situación de pandemia cuando se detectan brotes en un tercer país perteneciente a una región diferente de la OMS. La fase 6 se prolonga en 2 períodos, el posterior al pico máximo, reconoce la actividad decreciente, pero todavía no puede descartar que suceda otra onda pandémica. En el periodo post-pandémico la actividad gripal ha vuelto ya a los niveles estacionales habituales. Durante las fases 1 a 3 es necesario desarrollar la capacidad y planificación para la prepararse ante las fases siguientes. En las fases 4 a 6 habrá que llevar a cabo las medidas de respuesta y mitigación. En los períodos posteriores a la fase 6 es importante mantener la vigilancia y realizar una fase de recuperación y de evaluación de la pandemia.
En España El Plan Nacional de Preparación y Respuesta ante una Pandemia de Gripe se fue desarrollando a lo largo de varias versiones y años9. El 18/09/2003 se celebró la primera reunión del Comité Ejecutivo Nacional encargado de hacer una evaluación del riesgo para la población española y de aprobar protocolos de actuación. Así mismo, se designaron 4 grupos de trabajo específicos llamados Subcomité de Vigilancia de Gripe, Subcomité de Vacunas y Fármacos antivirales, Subcomité de Respuesta a la Emergencia y Subcomité de Comunicaciones.
Sistema de Vigilancia de Gripe en España (SVGE)
Según se muestra en la figura 3 está formado por:
- 19 Servicios de Epidemiología de las 17 CCAAs y las ciudades de Ceuta y Melilla, coordinados por el Centro Nacional de Epidemiología (CNE).
- 16 Redes de médicos y pediatras centinela, en la mayoría de las Comunidades Autonomas (CCAA).
- Una red nacional, ReLEG (Red de Laboratorios Españoles de Gripe) formada por 18 laboratorios con capacidad de detección de virus gripales, coordinados por el Centro Nacional de Microbiología (CNM), que es el centro de referencia a nivel nacional. Tres de los laboratorios de ReLEG son a la vez internacionales National Influenza Centres de la OMS [NIC]: CNM del ISCIII, Facultad de Medicina de Valladolid y Hospital Clinic de Barcelona.

La misión de la RELEG es vigilar los virus circulantes mediante su detección y caracterización genética y antigénica, lo que se lleva a cabo en colaboración con las Redes de Médicos Centinelas, que cubren a un 2% de la población española. La vigilancia de la gripe se basa en el análisis semanal conjunto de los datos virológicos y clínico-epidemiológicos, lo que permite al SVGE tener la visión detallada de la evolución de la actividad gripal en España y en cada una de sus CCAA. La explotación y análisis de los datos a nivel nacional la realiza el CNE. Semanalmente se publica un boletín de vigilancia de la gripe disponible en http://vgripe.isciii.es/gripe/inicio.do. La difusión de la información generada se integra en la vigilancia internacional de la enfermedad mediante el envío de la información semanal al ECDC.
El otro objetivo del diagnóstico virológico de la gripe es el de contribuir a la toma de decisiones clínicas en determinados pacientes. Se realiza fundamentalmente en los hospitales, como soporte a la asistencia que lleva a cabo el sistema sanitario. A través de ReLEG se incorpora al SVGE esta información virológica procedente de fuentes no centinela. Durante la pandemia estos datos han resultado de gran utilidad, para caracterizar el patrón de los virus responsables de las infecciones respiratorias de los pacientes hospitalizados.
Descripción de la vigilancia virológica realizada en España en las distintas etapas de la pandemia por el nuevo virus A(H1N1)
El 17 de Abril de 2009 los Center for Disease Control and Prevention (CDC)10 informaron de los primeros casos de infección por gripe A de origen porcino (H1N1) en dos niños que vivían en el sur de California. En ambos casos los virus estaban muy relacionados genéticamente, eran resistentes a los adamantanos y contenían una combinación única de segmentos genéticos que no se había descrito hasta ese momento ni en seres humanos ni en cerdos. Tras identificarse también otros casos humanos en México y sur de EEUU, los datos epidemiológicos disponibles hicieron sospechar que la enfermedad pudiera transmitirse de persona a persona11.
El 27 de abril la OMS pasó de la fase 3 de alerta pandémica a la 4 y dos días después la elevó a la fase 5, al comprobarse la existencia de casos en más de dos países de una misma Región de la OMS.
En España el 26 de abril se inició el estudio de tres pacientes que habían vuelto de México, confirmándose dos de ellos el día 27 en el CNM, como infección debida al nuevo virus de la gripe A12. De este modo, España, fue el primer país europeo que llevó a cabo el diagnóstico específico del virus A(H1N1)pdm.
A partir de ese momento la función del CNM fue diagnosticar los casos sospechosos importados y a sus contactos, mediante la utilización de técnicas de desarrollo propio, que por el momento eran las únicas disponibles. Inmediatamente se llevó a cabo la transferencia y puesta a punto de estas técnicas en el resto de laboratorios de la ReLEG. Por tanto, en los momentos iniciales el CNM se hizo cargo del diagnóstico de todos los casos, aunque después se fue asumiendo por la ReLEG, excepto en aquellas CCAA donde no se contaba todavía con laboratorio de referencia, en las que el CNM continuó asumiendo esta función. De esta forma el CNM se centró entonces en la confirmación de los casos positivos, en el apoyo a otros laboratorios en casos de diagnóstico problemático, y en la caracterización de los diferentes virus identificados. La ReLEG misma sirvió de apoyo a nuevos laboratorios hospitalarios que se iban sumando al diagnóstico virológico de los casos graves.
A continuación se describen sucintamente varias etapas diferenciadas en el transcurso de la pandemia, según los cometidos de la ReLEG y en las que sus laboratorios resultaron claves. Las características de estas etapas pueden identificarse en la figura 4.
Etapa 1. 25/04/2009-8/05/2009: (Semanas 17 y18, incluidas)
Esta etapa corresponde a la fase de contención del virus y el objetivo virológico es caracterizar la transmisión existente en el país (identificación de casos importados y posible transmisión autóctona). Los datos virológicos deben servir de soporte a cualquier cambio observado en la epidemiología o en la presentación clínica de la infección, para instaurar las medidas oportunas de control a nivel comunitario. En los primeros días en que no existía un método de diagnóstico molecular específico de la gripe A(H1N1)pdm, el CNM puso en marcha técnicas que había desarrollado antes. Resultó muy útil una PCR genérica y múltiple capaz de identificar cualquier virus gripal de los tipos A, B y C, la cual se basaba en la amplificación del gen de la NP, una de las proteínas virales más conservadas. También se habían desarrollado PCRs en otras proteínas virales, en las que se utilizaban primers con bases degeneradas que son más versátiles. A aquellos pacientes sospechosos de padecer la nueva gripe (por cumplir la definición de caso) se les tomaba preferentemente exudado nasal o faríngeo (siguiendo las directrices recogidas en el Plan Pandémico para AH5N1) y se enviaban al CNM para su diagnóstico. Este se basaba en dos tipos de ensayos independientes que se realizaban en paralelo. El primero consistía en la RT-PCR anidada en el gen de la NP, explicada antes, seguida por la caracterización mediante secuenciación. El segundo ensayo era semejante a este pero dirigido a amplificar la HA. En caso de obtenerse resultados contradictorios se realizaban otras PCR alternativas a fin de poder resolver el diagnóstico. La secuenciación y análisis de los productos amplificados mostró que en todos los casos se trataba de virus similares a los aislados en California (A/California/04/2009).
Previamente, en noviembre de 2008, la ReLEG había conseguido detectar un caso de gripe porcina en una mujer de Teruel con síntomas gripales, que trabajaba en una granja de cerdos. La muestra se identificó como virus gripe A en el Hospital Miguel Servet y la caracterización se llevó a cabo en el CNM demostrándose que los genes de la HA, NA, M y NP correspondían al linaje porcino euroasiático. No se pudieron demostrar infecciones secundarias13. Una vez iniciado el brote de México y EEUU, por indicación de la OMS se realizó otro análisis filogenético incluyendo los nuevos virus, demostrándose que el virus de Teruel se separaba claramente de ellos (figura 5).
A lo largo de esta etapa se confirmaron 88 casos de 640 sospechosos analizados. La mayoría de ellos (78%) correspondían a personas que habían viajado a México y el resto a contactos con los casos confirmados. La distribución de estas infecciones afectaba a toda España a excepción de Asturias, Canarias, Cantabria, Castilla y León, Navarra y La Rioja.
Etapa 2. 8/05/2009 - 10/06/2009: (Semanas 19-21, incluidas)
Continúa la fase de contención, centrándose en el diagnóstico como apoyo al análisis y caracterización de los brotes que van apareciendo.
El CDC puso a disposición de los laboratorios de la OMS una PCR a tiempo real, específica14 para la detección de los nuevos virus A(H1N1)pdm, que se recibió inmediatamente en el CNM, dado el elevado número de casos que se estaban diagnosticando en España. Se trataba de un método que permitía realizar cuatro PCRs de forma simultánea, conteniendo los primers para detectar: a) Gripe A genérica, mediante la amplificación del gen M; b) Gripe A porcina basada en el gen NP;. c) Nueva gripe A(H1N1)pdm por amplificación específica de HA; d) Control interno endógeno que amplificaba los genes de Ribonucleoproteasa P humana, a fin de controlar la presencia de inhibidores de la reacción en la muestra, sirviendo a la vez de control del proceso de extracción de ácidos nucleicos. La primera función del CNM fue probar el kit del CDC y realizar una evaluación comparativa de las técnicas del laboratorio frente al mismo. Se comprobó también que la PCR de gripe A era más sensible que la específica del virus pandémico, pudiendo ser origen de confusión con infecciones por virus de gripe estacional. Estas y otras observaciones se avisaron oportunamente a los laboratorios ReLEG y a la OMS. De hecho el CDC tiene ya disponible una segunda versión mejorada de este kit15.
Durante esta etapa, España seguía siendo el país europeo con más casos, excepto Gran Bretaña, donde aumentaron de forma vertiginosa. Las necesidad de disponer de un diagnóstico reproducible en todos los laboratorios ReLEG se fue resolviendo a través de las frecuentes teleconferencias convocadas por el laboratorio coordinador, que permitían la discusión y toma de decisiones comunes. A la vez, representantes de la ReLEG asistían también a las teleconferencias del Subcomité de Vigilancia, lo que ayudaba a alcanzar criterios y pautas comunes entre epidemiólogos y virólogos, traduciéndose en una mayor eficacia a la hora de tomar decisiones. A lo largo de esta etapa se acordaron los siguientes puntos en el seno de la ReLEG:
- La responsabilidad de la confirmación de los casos pasaba gradualmente desde el CNM a los laboratorios autonómicos integrantes de ReLEG.
- Se llevó a cabo una puesta en común de las técnicas para el tipado y subtipado de las muestras. Se alcanzó la unificación de criterios para realizar un diagnóstico homogéneo en todo el país.
- Se acordó la restricción del uso de test rápidos para la detección de gripe para casos muy puntuales, ya que presentaban menos sensibilidad y un alto coste.
- Se impulsó la introducción de la PCR a tiempo real (CDC) y su uso como técnica base para el diagnóstico.
- El CNM analizó una selección de los casos confirmados para evaluar comparativamente varios kits disponibles en el mercado. Varios laboratorios ReLEG aportaron también sus datos
- Se adoptaron medidas de bioseguridad y normas comunes en los laboratorios, especialmente en relación al aislamiento del virus.
- Se acordó llevar a cabo en el seno de la ReLEG la búsqueda activa de virus con mutaciones de resistencia a antivirales.
- El CNM continuó encargado de recoger datos de otras técnicas comerciales utilizadas en otros laboratorios o de la bibliografía para el caso de que se considerase necesaria su utilización.
- Se comenzó la preparación de una guía que recogiera las pautas y criterios que sobre el diagnóstico virológico se siguieron en los laboratorios de la ReLEG desde el inicio del brote de (H1N1) 200916.
El SVGE propuso, y así se aceptó por las autoridades sanitarias, que siguiendo las directrices de la OMS, la vigilancia centinela de gripe se prolongara entre las semanas 20 y 39 (última de septiembre) para el seguimiento del desarrollo de la pandemia durante el verano.
Etapa 3. 11/06/2009 - 26/07/2009. (Semanas 22-29, incluidas)
El 11 de junio la OMS declaró la Fase 6 de la pandemia y, por tanto, se pasó de un periodo de contención de la transmisión del virus a una fase de mitigación del impacto sanitario y social de la enfermedad.
Dado el gran número de diagnósticos que se requerían en aquel momento y el buen nivel tecnológico de los laboratorios ReLEG, se decidió que no era necesario que el CNM confirmara los resultados obtenidos en estos laboratorios, aunque debía mantener su labor de apoyo a los mismos. Durante esta etapa se analizaron 939 casos, resultando positivos 660 (70%) y en su mayoría (631) correspondientes a gripe A (H1N1) pdm (tabla 1). En la Comunidad de Madrid se observó un elevado número de casos debido a los brotes en el cuartel militar de Hoyo de Manzanares (iniciado en mayo), así como a los detectados en centros educativos y guarderías.
El objetivo principal de la ReLEG en la fase 6 fue identificar el número máximo posible de brotes mediante el diagnóstico de un pequeño porcentaje de casos de cada uno de ellos, para ayudar a interpretar los cambios epidemiológicos o clínicos que se fueran produciendo. En los casos individuales se decidió también una limitación del diagnóstico a casos seleccionados. Durante esta fase se potenció la identificación de mutaciones que pudieran indicar la evolución del virus o posibles cambios de comportamiento del mismo. De momento el virus se mantuvo aparentemente estable, en base a los análisis filogenéticos realizados en los fragmentos de genes HA y NA.
Etapa 4. 26/07/2009 -3/10/2009. (Semanas 30-39, incluidas)
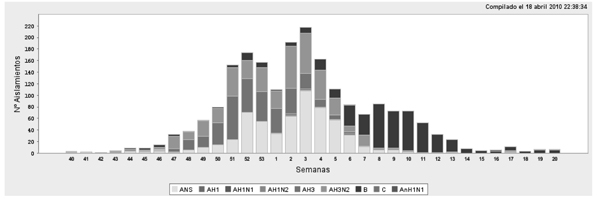
A partir de esta semana el Ministerio de Sanidad y Política Social determinó que no se contabilizaran los casos de gripe individualmente, sino que se realizaran estimaciones del número de casos respecto a la población general. La estimación se llevaba a cabo por el SVGE. Se observó que la contribución porcentual del virus A(H1N1)pdm al total de detecciones virales semanales, centinelas y no centinelas, fue aumentando desde la semana 21/2009, alcanzándose en la semana 37/2009 el máximo del 100% (figura 4).
Los laboratorios ReLEG realizaron además un gran número de diagnósticos de casos graves y a la vez sirvieron de apoyo a otros laboratorios de sus CCAA. El CNM ayudó a en la consolidación del estudio de mutaciones en HA y NA en los laboratorios autonómicos que podían hacerlo y asumió la caracterización del resto, ampliándolo también a los genes M, NP y NS. Se consideró prioritaria la búsqueda de mutaciones de resistencia a antigripales, especialmente en casos de personas inmunodeprimidas y en sus contactos más cercanos. Los laboratorios ReLEG declararon 1.340 casos positivos con más del 90% virus A(H1N1)pdm. Hasta el 3/10/2009 el CNM analizó 254 secuencias de HA y 210 de NA de cepas procedentes de las diferentes CCAA (tabla 2), determinando mutaciones en la secuencia respecto a la cepa de referencia A/California/07/2009.
Etapa 5. 4/10/2009 -15/05/2010 (Temporada 2009-2010)
Esta etapa abarcó toda la temporada 2009-2010 que, como se aprecia en la figura 4, correspondió la mayor parte de la onda pandémica, ya que la semana 40/2010 coincidió con el brusco aumento en la tasa de incidencia semanal que llegó al máximo en la semana 46. La ReLEG diagnosticó 1.0761 casos positivos, de los cuales el 99% correspondieron a gripe A y el 1% a gripe B. De la gripe A el 98% fue A(H1N1)pdm. La figura 6 muestra la gran homogeneidad de los virus durante aquella temporada en comparación con la mayor diversidad de la temporada 2008-2009. Durante el comienzo de esta temporada el CNM realizó la caracterización molecular de 307 HA y 227 NA de virus A(H1N1)pdm de diferentes CCAA, tal como se recoge en la tabla 2, juntamente con los datos de la etapa anterior. Diversos laboratorios de la ReLEG realizaron también un elevado número de caracterizaciones (alrededor de 300 HA y 800 NA). El análisis filogenético de las cepas estacionales AH3N2 y B que se identificaron durante la pandemia las clasificó como cercanas a las cepas vacunales de la temporada 2009-2010: A/Brisbane/10/2007(H3N2) y B/Brisbane/60/2008.
Reuniones del SVGE y de ReLEG: Además de las teleconferencias mencionadas a lo largo de la pandemia se realizaron reuniones presenciales tanto del SVGE, entre virólogos y epidemiólogos, como específicas de la ReLEG. La última se dedicó a hacer un balance de las actuaciones llevadas a cabo durante la pandemia y se llegó a acuerdos de organización para la siguiente temporada. Tanto los laboratorios de la ReLEG como el CNM continuarán las actividades de vigilancia gripal habituales para la circulación estacional, que se supone será la situación más probable durante la temporada 2010-2011. Manteniendo su la línea de coordinación y desarrollo para el futuro de ReLEG, el CNM hace especial hincapié en los siguientes puntos, que revierten en la especialización de ReLEG:
- Desarrollo de nuevas PCR a tiempo real propias, tanto para el tipado como para el subtipado de virus de la gripe, así como de otros virus respiratorios.
- Refuerzo de los métodos para estudios seroepidemiológicos, incorporando la Microneutralización a la Inhibición de Hemaglutinación, ya disponible.
- Estudio fenotípico de susceptibilidad a los agentes antivirales de forma sistemática. El estudio de resistencias ha sido prioritario para el CNM que venía participando desde hace años en el grupo de trabajo de antivirales europeo (ECDC)
- Puesta a punto de métodos adicionales de secuenciación, tales como la pirosecuenciación para el estudio de mutaciones puntuales. El CNM está participando en estudios de secuenciación masiva de virus pandémicos, que en breve se podrán realizar en un secuenciador propiedad .
Por su parte los laboratorios de la red continuarán profundizando o pondrán en marcha los estudios de caracterización de los genes HA y NA. En este aspecto, a causa del interés suscitado por la secuenciación y el análisis filogenético durante la pandemia, entre los días 16 y 17 de Marzo de 2010 se realizó en el CNM el «Curso práctico de análisis molecular de los virus de la gripe título», para la enseñanza práctica de las técnicas de análisis de las secuencias de la gripe pandémica. En él participaron 15 laboratorios de la ReLEG.
Ampliando las fronteras al espacio iberoamericano y dentro del proyecto VIRORED de CYTED, en mayo de 2010 se realizó otro curso en el CNM en el que participaron 10 países de América Latina y Centro América. En él, además de las clases prácticas de filogenia de gripe, se hizo un análisis y puesta en común de cómo se había afrontado la pandemia en cada país, comenzando por la experiencia en España. Con este mismo objetivo de mejorar el diagnóstico y caracterización viral, distintos investigadores han realizado estancias de mayor o menor duración en el laboratorio del CNM. Dentro de este ámbito internacional, dado que España fue el primer país europeo en diagnosticar casos de gripe A, el laboratorio coordinador fue invitado a participar en las reuniones especificas sobre gripe A realizadas por el ECDC, a fin de explicar su forma de actuar en las primeras semanas de la pandemia.
Conclusiones
1. A pesar del comienzo brusco de la pandemia todos los elementos de la red comenzaron a funcionar de forma eficiente y coordinada y no se perdió tiempo en las actuaciones primarias. Esto fue posible gracias a que uno de los objetivos prioritarios de esta red ha sido irse capacitando técnicamente y con equipamiento, protocolos de trabajo y organización de laboratorios, para una eventualidad de emergencia de un nuevo virus respiratorio, como fue el caso del SARS o la pandemia actual, o podría ser una pandemia por el virus aviar AH5N1.
2. La ReLEG ha servido también de apoyo a otros laboratorios del Sistema Sanitario con su experiencia, metodología, cesión de reactivos y de protocolos. Las discusiones en el seno de la red sobre criterios y pautas de actuación, dieron lugar a recomendaciones y documentos guía para distintos momentos de la pandemia. Esto mismo ha sido útil también para los laboratorios iberoamericanos.
3. Lo más importante es que el objetivo principal de la red de laboratorios del SVGE se alcanzó, adaptándose para cubrir en cada momento las necesidades de la vigilancia, tanto en las ultimas fases prepandémicas como durante la pandemia y en el período postpandémico. Esta experiencia le fue muy útil a la red, que ahora se encuentra mejor preparada para cualquier otra eventualidad o emergencia.
4. Como experiencia negativa de la que se deben sacar consecuencias hay que poner de relieve que al ir avanzando en la fase 6 hacia una organización sostenible, la gran carga de trabajo existente durante un tiempo prolongado llevó en muchos momentos en distintos laboratorios a quebrar sus esquemas de trabajo. Por tanto habrá que revisar las estructuras que se han utilizado en las fases avanzadas de la pandemia. Estos aspectos sobre situación de larga duración no estaban suficientemente previstos y desarrollados en el Plan Pandémico.
5. A partir de la experiencia adquirida deben preparase mejor las fases no iniciales de la pandemia, haciendo disponibles los recursos necesarios, especialmente de personal, para evitar que se produzca fatiga y la vigilancia pueda hacerse sostenible.
6. Afianzar y definir mejor la relación entre la vigilancia humana y la animal para que en la gripe y en todas las zoonosis se compartan fácilmente los datos y se genere un verdadero conocimiento de la infección que permita estar preparados ante cualquier emergencia en cualquiera de los dos ámbitos, animal y humano
Bibliografía
1. Arias CF, Escalera-Zamudio M, Soto-del Rio MD et al. Molecular Anatomy of 2009 Influenza Virus A (H1N1). Arch Med Res. 2009;40(8):643-54. [ Links ]
2. Garten RJet al. Antigenic and genetic characterizations of swine origin 2009 A(H1N1) influenza virus circulating in humans. Science. 2009; 325: 197-201. [ Links ]
3. Shinde et al. Triple-reassortant swine influenza A (H1) in humans in the United States, 2005-2009. N Engl J Med. 2009; 360(25): 2616-25. [ Links ]
4. Nelson M, Sprio D, Wentworth D et al. The early diversification of influenza A/H1N1 pdm. PLoS Currents Influenza. 2009. RRN1126. [ Links ]
5. Kilander A, Rykkvin R, Dudman SG, Hungnes O. Observed association between the HA1 mutation D222G in the 2009 pandemic influenza A(H1N1) virus and severe clinical outcome, Norway 2009-20010. Eur Surveill. 2010;15(9)pii.19498). [ Links ]
6. World Health Organization. Preliminary review of D222G amino acid substitution in the haemagglutinin of pandemic influenza A(H1N1) 2009 viruses. Wkly Epidemiol Rec. 2010;85(4):21-2. [ Links ]
7. World Health Organization. Pandemic (H1N1) 2009: antiviral drug resistance. Disponible en: http://www.who.int/csr/disease/swineflu/frequently_asked_questions/antivirals/resistance/en/ [ Links ]
8. World Health Organization Current WHO phase of pandemic alert for avian influenza H5N1. Disponible en: http://www.who.int/csr/disease/avian_influenza/phase/en/ [ Links ]
9. Ministerio de Sanidad y Política Social. Plan Nacional de preparación y respuesta ante una pandemia de gripe. Actualización. Disponible en: http://www.msps.es/ciudadanos/enfLesiones/enfTransmisibles/docs/ActualizacionPlan_diciembre2006.pdf [ Links ]
10. Centers for Disease Control and Prevention. Swine Influenza A (H1N1) infection in two children-Southern California, March-April 2009. MMWR. 2009;58(15):400-402. [ Links ]
11. Centers for Disease Control and Prevention (CDC). Outbreak of swine-origin influenza A(H1N1) virus infection-Mexico, March-April 20099. MMWR. 2009;58(dispatch):1-3. [ Links ]
12. Surveillance Group for New Influenza A(H1N1) Virus investigation an control in Spain. New influenza a (H1N1) virus infection in Spain, April-May 2009. Euro Surveill. 2009;14(19):1-4. [ Links ]
13. Adiego Sancho B, Omeñaca Terés M, et al. Human case of swine influenza A (h1N1), Aragon, Spain, November 2008. Euro Surveill. 2009;14(7)pii: 19120. [ Links ]
14. Who Health Organization. CDC protocol of realtime RTPCR for influenza A (H1N1). Disponible en: http://www.who.int/csr/resource/publications/swineflu/realtimepcr/en/index.html [ Links ]
15. New CDC Test to Detect Human Infections with the 2009 H1N1 Influenza Virus Authorized for Use by FDA. Disponible en: http://www.cdc.gov/media/pressrel/2010/r100622.htm [ Links ]
16. ReLEG. Infección por el virus pandémico (H1N1) 2009. Protocolo de diagnóstico virológico.Disponible en: http://www.msc.es/profesionales/saludPublica/gripeA/docs/ProtocoloVirologico.pdf [ Links ]
 Dirección para correspondencia:
Dirección para correspondencia:
Pilar Pérez-Breña
Carretera Majadahonda-Pozuelo km 2
Majadahonda 28220 (Madrid)
pperez@isciii.es


















