Mi SciELO
Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO -
 Accesos
Accesos
Links relacionados
-
 Citado por Google
Citado por Google -
 Similares en
SciELO
Similares en
SciELO -
 Similares en Google
Similares en Google
Compartir
Cuadernos de Medicina Forense
versión On-line ISSN 1988-611Xversión impresa ISSN 1135-7606
Cuad. med. forense vol.16 no.1-2 Málaga ene./jun. 2010
Nuevas aplicaciones en identificación genética
New applications in genetic identification
M.J. Álvarez-Cubero1; L.J. Mtnez.-Gonzalez1; M. Saiz1; J.C. Álvarez1,2; J.A. Lorente1,2
1Laboratorio de Identificación Genética, Departamento de Medicina Legal. Universidad de Granada.
2GENyO - Pfizer. Universidad de Granada. Junta de Andalucía. Centro de Genética y Oncología. Armilla (Granada).
Dirección para correspondencia
RESUMEN
Cada vez más, los avances en identificación genética permiten la aplicación de estas técnicas a áreas muy diferentes. Los progresos científicos y tecnológicos en las herramientas utilizadas en genética forense han permitido nuevas aplicaciones de este campo en otras disciplinas. En este trabajo se pretenden abordar algunas de esas nuevas alternativas. La primera de ellas es la creación de bases de datos genéticas civiles que ha permitido el desarrollo de dos programas de identificación de personas: programa FÉNIX de identificación de cadáveres y restos humanos desaparecidos y la iniciativa DNA-PROKIDS que lucha contra el tráfico de seres humanos y pretende reunir a las víctimas con sus familias. En la última década, cada vez con más frecuencia aparece el análisis del ADN en proyectos de historia, paleontología, Es en estos casos donde nos encontramos con muestras antiguas. Por tanto, una segunda aplicación es el trabajo con aDNA, del que mostramos varios ejemplos. Gracias a estas nuevas técnicas se han podido identificar los restos de varios personajes históricos, el Zar Nicolás II de Rusia, Cristóbal Colón y sus familiares, Blanca de Navarra y el Príncipe de Viana y el general Miranda; así como la recuperación de especies en peligro de extinción y la caracterización de elementos históricos. Por último, tienen gran importancia sus aplicaciones en biomedicina, ya que cada día es más frecuente la colaboración entre grupos forenses y otras especialidades médicas. Dos ejemplos de ello serían, la caracterización de muestras provenientes de tumores y en la asociación de enfermedades a haplotipos genéticos.
Palabras clave: aDNA. Cáncer. Base de datos genéticas.
ABSTRACT
More now than ever, advances in genetic identification allow the application of these new techniques to different fields. Scientific and technological progress in the tools used in forensic genetics has permitted new implementations of this field to others. In this paper, some of the newest alternatives will be tackled. Firstly, the creation of a civil genetic database has allowed the development of programs for human identification. FENIX, program, to identify corpses and missing human remains and DNA-PROKIDS that fights against human trafficking and hopes to reunite victims with their families. In the last decade, it is becoming more common to encounter DNA analysis in history projects, paleontology and so on. It is in such cases that we find ancient samples. So, another application is the work with aDNA of which we will show some examples. Thanks to these new techniques some historical remains of important personages have been identified, Zar Nicolás II of Rusia, Cristobal Colombus and his relatives, Blanca of Navarra, the Prince of Viana and the general Miranda; as well as the recovery of some species in danger of extinction and the characterization of some historical elements. Lastly, it has also a great relevance the applications of these techniques to biomedicine, given than the collaboration between forensic groups and other medical specialities is more and more frequent theses days. Two examples of this situation will be the characterization of tumor samples and the association of some diseases with genetic haplotypes.
Key words: aDNA. Cancer. Genetic database.
Introducción
Este año se cumplen 25 años desde que Alec J. Jeffreys, profesor de Genética de la Universidad de Leicester, descubrió el primer locus polimórfico. Para hallar este marcador se empleó una sonda de ADN arbitraria. De este modo se observaron fragmentos de más de 15 longitudes diferentes. Posteriormente, se encontraron otros loci hipervariables que constaban de repeticiones en tándem de una secuencia de oligonucleótidos (11 a 60 pb), de manera que las diferentes longitudes de los fragmentos originados dependían del número de dichas repeticiones y se les denominó VNTR ("Variable Number of Tandem Repeat"). Tras el descubrimiento de los primeros VNTRs se vio que éstos podían ser aplicados a la medicina forense y sustituir a los marcadores clásicos. En un principio la cantidad de ADN necesaria, así como, el estado del mismo era un requisito indispensable para poder utilizar estos marcadores. Todas estas limitaciones fueron superadas gracias a la aplicación en Genética Forense de una técnica, la Reacción en Cadena de la Polimerasa ("PCR"), que supuso una revolución en muchos campos de la Biología y de la Medicina. Esta técnica, ideada en 1989 por Kary B. Mullis, permitió que se pudieran obtener resultados de muestras biológicas con mínimas cantidades de ADN.
A finales de los 90 aparecen en nuestro país nuevas tecnologías que permiten acelerar el proceso de análisis de las muestras. Así el procesamiento de un centenar de perfiles genéticos se podía realizar en un par de días. La aparición de los analizadores genéticos rápidamente desplazó a los laboriosos geles de acrilamida, que limitaban en gran medida los resultados de un laboratorio. La electroforesis capilar ha sido, durante estos últimos quince años, la herramienta más utilizada por los laboratorios de genética forense.
Durante estos años, los grupos de investigación en este campo se han esforzado en descubrir nuevos marcadores, en nuestro genoma, que faciliten la resolución de los casos forenses: marcadores autosómicos, ADN mitocondrial y cromosoma Y. En esta línea muchos grupos siguen buscando marcadores del tipo SNPs (polimorfismos de un único nucleótido) que nos den información sobre el fenotipo y la procedencia geográfica de una muestra biológica. También se ha intentado unificar en todas las poblaciones el tipo de marcador utilizado, siendo el sistema CODIS (Combined DNA Index System) desarrollado en Estados Unidos por el FBI, el que ha conseguido una mayor difusión.
Su aplicación ha hecho que poco a poco fuera necesario el uso de bases de datos genéticos que permitieran la búsqueda y el rastreo de perfiles comparados, lo que ha permitido la incorporación de bioinformáticos y bioestadísticos en los grupos de investigación forense.
Las nuevas plataformas de secuenciación, así como, los nuevos kits de extracción y purificación de las muestras, asociados a robot de extracción, son en este momento las nuevas tecnologías de las que los grupos comenzarán a disponer en breve. Estas nuevas herramientas permitirán un aumento exponencial del número de datos generados. Con lo que los grupos podrán analizar miles de muestras a diario, y tendrán que ser los nuevos software, expert system, los que valoren los resultados.
Toda esta experiencia, en el campo de la genética, no sólo ha mejorado la resolución de los casos de filiación y criminalística, sino que ha permitido que los grupos de genética forense aborden nuevos campos de investigación, y que colaboren con otros grupos ampliando así las fronteras de nuestra comunidad científica. En este trabajo se presentan algunas estas nuevas aplicaciones, que abordan el ámbito social, histórico y sobre todo la ayuda a todas las especialidades biomédicas.
Aplicaciones en bases de datos genéticas
Los avances científicos y técnicos en las herramientas moleculares empleadas en genética han hecho posible su aplicación en el campo de la genética forense teniendo un gran impacto social. Gracias a ellos se han generado una cantidad de información extraordinaria que ha de almacenarse de modo racional y ordenado para su posterior uso. Surge así la necesidad de las denominadas "bases de datos", lugares donde se almacena de modo ordenado y coherente cualquier tipo de información, que luego es rescatada de modo automático de acuerdo a parámetros previamente establecidos. El acceso a las mismas es más o menos restringido, dependiendo de la trascendencia de los datos almacenados, siendo los más protegidos aquéllos que contienen información sobre la "identidad" de las personas1-3.
Las bases de datos de identificación genética contienen números y letras asociados al código de identificación de una persona. Su acceso a los datos debe de estar perfectamente controlado, y las conclusiones que se pueden obtener de los mismos van a depender de los programas informáticos que se autoricen por las diferentes legislaciones. Dependiendo del contenido de estas bases de datos se pueden diferenciar:
- Bases de datos forenses criminales: almacenan datos procedentes de personas que han sido procesadas o condenadas, así como de indicios biológicos encontrados en la escena del crimen. En algunos casos pueden considerarse perfiles de víctimas conocidas, con objeto de facilitar la resolución de delitos. Su característica principal es que algunas de las muestras y datos considerados se obtienen sin el consentimiento de las personas implicadas.
- Bases de datos forenses civiles: su único fin es la identificación de personas desaparecidas, lo cual se hace comparando el ADN de las personas no identificadas con el de los familiares. Por sus características humanitarias, se requiere que los familiares colaboren de modo voluntario y tras firmar un consentimiento informado, cosa que las diferencia de las bases de datos criminales siguiendo las consideraciones del protocolo de Helsinki.
Actualmente, la creación de bases de datos forense civiles permite el uso de éstas con fines humanitarios. Éste es el caso de los proyectos que se desarrollan actualmente en el Laboratorio de Identificación Genética de la Universidad de Granada; el programa FÉNIX y la iniciativa DNA-PROKIDS.
Programa FÉNIX
Se han hecho grandes esfuerzos en la identificación de cadáveres y restos humanos tras guerras, problemas socio-políticos y desastres en masa. En muchos de estos casos, el uso de la identificación a través de ADN ha ofrecido una respuesta definitiva en la identificación de las víctimas. La creación de una base de datos civil de personas desaparecidas no compromete los derechos de los individuos ya que la donación de las muestras incluidas en ella es voluntaria. España fue el primer país en establecer una base de datos forenses civil. Debido a la dinámica de las sociedades modernas, constantemente se presentan denuncias de personas desaparecidas así como se encuentran cadáveres y restos humanos. Se trata de un problema universal, una situación común que no solo está relacionada con guerras o catástrofes naturales. Es obvio que muchos de los cadáveres y restos encontrados corresponderán con personas desaparecidas denunciadas.
En 1998, el grupo de investigación del Departamento de Medicina Legal y Toxicología de la Universidad de Granada ideó un programa de identificación genética de personas desaparecidas (Programa FÉNIX) para la identificación de cadáveres o restos cadavéricos que no se pudiesen identificar empleando métodos tradicionales. Su finalidad era prestar ayuda a los familiares de las personas desaparecidas. Éste programa fue pionero en el mundo entero ya que anteriormente no existía ni existe ninguna base de datos con estos fines. Ésta iniciativa fue apoyada por la Guardia Civil y el Ministerio del Interior2,3, lo que permitió la creación de un programa nacional de identificación de cadáveres y restos de personas desaparecidas.
El programa FÉNIX contiene dos bases de datos independientes donde se pueden comparar automáticamente secuencias de ADN para identificar perfiles relacionados entre sí. Una se conoce como Base de Datos de Referencia que contiene información de microsatétiles de ADN nuclear y cromosoma Y así como de secuencias de ADN mitocondrial de parientes que provienen de la línea materna. Debido a que esta base de datos es una base no criminal, sólo se encuentran en ella aquellos individuos que donan su ADN de forma voluntaria y siempre tras firmar un documento de consentimiento informado. La otra base de datos se denomina Base de Datos Dubitada y contiene todos los perfiles de restos encontrados que no han podido ser identificados por métodos clásicos como son, identificación de huellas dactilares, parámetros antropológicos, odontología, rayos X Actualmente, 800 familias de personas desaparecidas participan en el programa FÉNIX y se han logrado 350 identificaciones de restos encontrados3 (http://www.guardiacivil.org/prensa/actividades/FÉNIX/presentacion.jsp) (Figura 1).
Estrategias similares se están implantando cada vez más en todo el mundo. En Brasil, donde en 2007 se determinó que existían 7287 cadáveres y restos humanos sin identificar, se ha desarrollado una herramienta bioinformática para registrar y comparar la información genética y no genética de las personas desaparecidas, sus familiares y cadáveres no identificados, Missing person Database-gen4. Tras la guerra de la antigua Yugoslavia, el ICMP (International Commission of Missing Persons) desarrolló un programa de recogida de muestras de familiares de personas desaparecidas para la identificación de todos aquellos cadáveres y restos desconocidos5.
Desde los primeros pasos del programa FÉNIX diversos países del área iberoamericana se mostraron interesados en su desarrollo. Algunos, incluso, pretenden poner en marcha un programa similar, dándose el caso de que, como en Colombia, se pretende que se denomine "FÉNIX-Colombia". En este país, responsables del Laboratorio de Genética del CTI (Centro Técnico de Investigación), adscrito a la Fiscalía General de la Nación, pretenden instaurar un sistema de identificación genética de personas desaparecidas para paliar el gravísimo problema que en este sentido genera el elevado índice de violencia producido tanto por la actuación de la guerrilla como por otras bandas de delincuencia organizada fundamentalmente relacionadas con el narcotráfico. En Chile, el Servicio Médico Legal, por orden del Parlamento de este país, pondrá en marcha una base de datos de muestras de detenidos-desaparecidos, habiéndose solicitado la colaboración del Programa FÉNIX. En USA, el UNT Health Science Center creó el Texas Missing Persons DNA Database en 2001, una base de datos similar a FÉNIX española.
Iniciativa DNA-PROKIDS
Esta iniciativa, quiere ser una herramienta para luchar contra el tráfico de seres humanos, en especial de los más débiles, los niños. Según la UNODC, "Por trata de personas' se entenderá la captación, el transporte, el traslado, la acogida o la recepción de personas, recurriendo a la amenaza o al uso de la fuerza u otras formas de coacción, al rapto, al fraude, al engaño, al abuso de poder o de una situación de vulnerabilidad o a la concesión o recepción de pagos o beneficios para obtener el consentimiento de una persona que tenga autoridad sobre otra, con fines de explotación"6-8.
Los niños, los individuos más inocentes y frágiles de la sociedad, pueden ser sujeto de muchos tipos de abusos. Uno de éstos es el tráfico humano que se ha convertido en una actividad criminal lucrativa. Según UNICEF, 300 millones de niños en todo el mundo sufren violencia, explotación y abusos incluyendo las peores formas de explotación laboral infantil, conflictos armados y prácticas dañinas. En los Estados Unidos se denuncian 800000 desapariciones de niños al año. De éstos, aproximadamente 360000 se escapan, 340000 se clasifican como "desapariciones sin explicación plausible" y unos 100000 son secuestrados por miembros de su familia u otros individuos conocidos o han sufrido algún daño6,8.
Los gobiernos son conscientes de que el problema del tráfico humano es real y que se han de tomar medidas inmediatas. Sin embargo, es conveniente llegar a acuerdos internacionales para identificar las posibles víctimas y los responsables de estos delitos. Pero las dificultades legales y sociales que existen ralentizan el proceso de implementar un sistema contra el tráfico humano. Esto puede ser muy frustrante para los científicos que quieran ayudar desde una perspectiva humanitaria y disponen de las aplicaciones técnicas necesarias para asistir en la identificación de personas desaparecidas. Aunque las infraestructuras legales se desarrollen de forma lenta, no hay razón alguna para no implementar las herramientas analíticas y las bases de datos para la identificación de los niños desaparecidos6,7.
DNA-PROKIDS es un proyecto internacional centrado en la lucha contra el tráfico de seres humanos a través de la identificación genética de las víctimas y sus familiares, especialmente en niños. Su meta no se centra únicamente en la resolución de crímenes domésticos sino que se pretende extender los resultados a nivel mundial con el objetivo de estimular la lucha contra el tráfico de seres humanos y ayudar a prevenir la misma, http://www.dna-prokids.org/9.
El proyecto DNA-PROKIDS se inició en 2004 a través del Laboratorio de Identificación Genética de la Universidad de Granada. Tras un estudio piloto con países de Centroamérica y Asia llevado a cabo entre 2006 y 2008, se decidió la ampliación definitiva con alcance mundial. En 2009, con la colaboración de los Profs. Bruce Budowle y Arthur Eisenberg del Centro de Identificación Humana de la Universidad del Norte de Texas (UNT-HIC) y con la financiación de BBVA, Caja Granada, Fundación Marcelino Botín (Banco Santander) y Life Tecnologies (EEUU) comenzó la expansión internacional del proyecto. Actualmente se ha ampliado la colaboración -inicio de la expansión mundial- a China, India, Indonesia, Filipinas, Nepal, Sri Lanka, Tailandia, Brasil, Guatemala y México.
La principal misión de DNA-PROKIDS es identificar a las víctimas y devolverlas a su familia (reunificación); dificultar el tráfico de seres humanos, incluyendo las adopciones ilegales, gracias a la identificación de las víctimas (prevención); y obtener información sobre los orígenes, las rutas y los medios de comisión del delito, claves para el trabajo de las fuerzas policiales y judiciales (inteligencia policial). Por otro lado, los objetivos de la iniciativa DNA-PROKIDS son:
- Promover una colaboración internacional sistemática y automática a través de la integración en una misma base de datos mundial de datos genéticos para lograr:
- Reintegrar a estos niños desaparecidos en sus familias gracias a la comparación de su ADN con el de los familiares de personas desaparecidas susceptibles de haber sido víctimas de tráfico de seres humanos.
- Luchar contra las adopciones ilegales al comprobar si las personas que entregan a un menor en adopción son los verdaderos familiares biológicos (madre, padre, abuelos), para evitar que menores procedentes de raptos, robos o tráfico de personas puedan ser dados en adopciones presuntamente legales.
- Estudiar y aplicar mejoras a los sistemas policiales y judiciales de todo el mundo para que luchen más eficazmente contra el tráfico de seres humanos, especialmente de niños y mujeres.
- Analizar y proponer una base legislativa común para la resolución de este problema.
- Evaluar e intentar solventar los problemas sociales y de comunicación que impiden una lucha más eficaz contra el tráfico de seres humanos.
- Ofrecer colaboración para la coordinación y la formación de especialistas en identificación de los diferentes países.
DNA-PROKIDS emplea métodos acreditados y aceptados por la comunidad científica internacional para la identificación genética de las personas desaparecidas. Los marcadores genéticos de rutina que se estudian son aquellos STR incluidos en sistemas como CODIS, ADN mitocondrial para confirmar la línea materna y STR del cromosoma Y para establecer relaciones de tipo paterno-filial. Actualmente, se están desarrollando paneles de SNPs (Single Nucleotide Polymorphism) que nos van a permitir determinar características fenotípicas de los individuos así como su procedencia geográfica y étnica. Con toda esta gran cantidad de información se podrán crear bases de datos que nos permitirán la identificación de los menores víctimas de tráfico humano y la reunificación de éstos con sus familias (Figura 2).
Desde 2005, el Laboratorio de Identificación Genética de la Universidad de Granada ha procesado aproximadamente unas 1200 muestras dentro del programa DNA-PROKIDS. En 2008 y 2009 se registraron 107 casos englobados en el programa procedentes de Guatemala. Por otro lado, durante este mismo tiempo el Centro de Identificación Humana de la Universidad del Norte de Texas también ha llevado a cabo la identificación de menores víctimas del tráfico humano. Desde principios de 2009, gracias al programa DNA-PROKIDS se han podido identificar a 230 menores en 12 países de todo el mundo. Durante el mes de Octubre de 2009 se celebró en Granada la primera reunión científica DNA-PROKIDS donde expertos en identificación genética de todo el mundo se reunieron para elaborar un plan de actuación conjunto frente a casos de tráfico humano. En Enero de 2010, se reunieron en Filipinas el Dr. José Antonio Lorente y el Prof. Arthur Eisenberg con los expertos del "DNA Analysis Laboratory of the Natural Sciences Research Institute" y del "Philippine Council for Advanced Science and Technology Research and Development" con el fin de informar y asesorar a estos organismos sobre esta iniciativa. El día 01 de Febrero de 2010 se presentó en Madrid en una rueda de prensa organizada por la AECID y el Ministerio de Asuntos Exteriores la oferta formal del Gobierno español al de Haití para que se desarrolle de modo urgente y especial el programa DNA-PROKIDS, tratando de detener el tráfico ilegal de menores (incluidas adopciones ilegales) y el apoyo a la reunificación familiar en este país tras el terremoto del 12 de Enero de 2010.
Aplicaciones en muestras antiguas
Las técnicas empleadas en genética forense están principalmente basadas en la determinación de fragmentos y secuencias de ADN. Las muestras que normalmente se encuentran en el campo de la criminalística no suelen ser el material idóneo para trabajar con las técnicas clásicas de biología molecular. Con mucha frecuencia el ADN que se consigue de estas muestras tiene unas características que hacen necesario el desarrollo de nuevos protocolos y tecnologías para obtener resultados.
Las características del ADN en los casos de criminalística son: su bajo número de copias (material biológico en escasa cantidad y dañado), fragmentación (segmentación del ADN en distintos tamaños inferiores al inicial), inhibición (aparición de partículas que dificultan hacer copias del contenido genético) y modificaciones estructurales en la secuencia de bases del ADN10.
Los grupos de investigación que se enfrentan a este tipo de ADN solucionan las dificultades empleando nuevos procedimientos y tecnologías para solventar los problemas citados anteriormente.
- Obtener la máxima cantidad de ADN posible: Es fundamental en este tipo de casos no perder ADN de la muestra de partida11, ajustando los protocolos de extracción hasta el punto de finalizar este tratamiento obteniendo una concentración de ADN similar a la de partida. Esto se realiza mediante el uso de filtros concentradores o diseñando Kits específicos para cada muestra de partida.
- Diseñar estrategias que reduzcan el tamaño del ADN problema: eliminando los problemas de fragmentación del ADN, utilizando primers dentro de un pequeño rango en pares de bases o diseñando parejas de primers o kit multiplex con rangos de amplificación no mayores a los 250pb12,13.
- Purificar el extracto de ADN: mediante detergentes, agentes quelantes, lavados con soluciones hipotónicas, materiales absorbentes, Dependiendo del tipo de iones o moléculas que nos impidan que la amplificación del ADN de nuestra muestra, se procederá al tratamiento adecuado con una técnica u otra14-16.
- Conocer los procesos que ocurren en el ADN antiguo y degradado10,17-19: las reacciones de oxidación (cambios en los azúcares de las bases), la radiación, las reacciones hidrolíticas, los procesos de degradación enzimática,... Resultados que pueden aparecer en este tipo de ADN: mezclas o contaminación con otros ADN, pérdida de la heterozigosidad, uniones cruzadas de fragmentos o fragmentos y primer, introducción de bases erróneas,... Este último punto ha hecho que se estudien de manera exhaustiva los protocolos de amplificación y secuenciación, número de ciclos, temperaturas, diseño de primers; características que debe de tener un laboratorio forense y qué criterios de calidad debe seguir, así como, qué tipo de ADN y zonas de éste, de toda nuestra dotación genética, son más útiles para la realización de un estudio determinado.
El conocimiento y puesta a punto de estos protocolos y técnicas, así como la investigación en cada uno de los campos citados, ha permitido a nuestro grupo el desarrollo de multitud de proyectos y colaboraciones con otros grupos en este campo.
Cuando se tiene la capacidad de hacer análisis de ADN antiguo, se abre un nuevo campo de acción. Al igual que en el campo de la criminalística, el estudio del ADN en casos antiguos puede hacer cambiar una decisión judicial. El estudio de éste en nuevos proyectos con tejidos biológicos antiguos puede cambiar la historia de un personaje, de una especie La historia es una ciencia que ha sabido aprovechar las nuevas tecnologías para aumentar el número de elementos que apoyen una teoría. El ADN no es la primera, se ha utilizado el carbono 14 desde hace años para datar piezas y así recomponer hipótesis en investigaciones de sucesos históricos. Dentro de las metodologías de la historia, el ADN no es una herramienta para reinterpretarla, sino un instrumento más para ayudar al resto de las fuentes de trabajo clásicas (textos, grabados, materiales, ).
Estos proyectos, como vemos, no están incluidos dentro del campo forense, pero hacen de ésta ciencia, cada día más, una herramienta multidisciplinar.
Identificación de personajes históricos
Uno de los primeros casos históricos donde la identificación genética se ha convertido en una herramienta decisiva ha sido en el caso del Zar Nicolás II de Rusia. El ADN mitocondrial y el cromosoma Y permitieron confirmar el fallecimiento del Zar Nicolás II Rusia y de sus familiares, siendo este el fin de su dinastía20-22.
Pocos años después se comenzó desde la Universidad de Granada el proyecto de identificación de algunos de los personajes más importantes de la historia de nuestro país, Blanca de Navarra y el Príncipe de Viana. El estudio puso de manifiesto que ninguno de los restos encontrados en la momia que se asumía como el cuerpo del Príncipe de Viana coincidía con los de Blanca de Navarra, madre del príncipe. Todas las secuencias de ADN mitocondrial editadas pertenecían a individuos distintos. Se encontraron hasta tres huesos con distinta procedencia en la momia del Príncipe de Viana. Estos resultados se pueden explicar debido a que en el sepulcro de Poblet existía un caos de huesos, fruto de los distintos saqueos que había sufrido. Estudios realizados en el año 2008 por otros grupos corroboran los datos que se obtuvieron en nuestro laboratorio a finales de la década de los 90 confirmando la existencia de más de un individuo en la momia del Príncipe.
Además del citado, uno de los proyectos más trascendentes y de mayor repercusión fue la identificación genética de Cristóbal Colón y sus familiares, un proyecto internacional y multidisciplinar con el objetivo de descifrar algunos de los enigmas del famoso Almirante. El objetivo principal de este proyecto fue determinar el lugar donde se encontraban sus restos, República Dominicana y/o Sevilla.
El estado de los huesos, de Cristóbal Colón y de su hermano Diego, era mucho peor del esperado por el tiempo trascurrido, los distintos viajes del féretro de Colón y la poca cantidad de material encontrado en la tumba de la Catedral de Sevilla. Igualmente los restos de Diego, debido a las filtraciones de agua, se encontraban en un estado muy deteriorado. De todos los laboratorios participantes en el proyecto, solo en algunos se obtuvieron resultados. Se encontraron coincidencia en los resultados, pero los fragmentos de ADN mitocondrial obtenidos fueron muy pequeños, con lo que fue difícil afirmar una inclusión. La suma de los datos obtenidos por todos los participantes antropólogos, historiadores, fueron los que nos permitieron afirmar que los restos encontrados en la catedral de Sevilla, pertenecían al almirante. De todas formas este proyecto no se finalizará hasta que las autoridades de la República Dominicana no permitan contrastar estos datos con los restos del mausoleo levantado en Santo Domingo. La cantidad de huesos encontrados en la catedral de Sevilla no descarta que ambas tumbas compartieran la posesión del los restos de Colón, algo que sucedía frecuentemente en los traslados de cadáveres de personajes famosos y reliquias.
En este momento, nuestro grupo de investigación realiza, en esta misma línea, la identificación de los restos de «El Primer Venezolano Universal», Francisco de Miranda. El general Venezolano, S. Francisco de Miranda Rodríguez (Caracas, 1750 - San Fernando (Cádiz), 1816) se considera como el precursor de la independencia americana del Imperio español. Participó en tres de las guerras más importantes a favor de la democracia: la Independencia de los Estados Unidos, la Revolución francesa y las Guerras de Independencia Hispanoamericana. Francisco de Miranda debido a la firma de un acuerdo con los españoles, temiendo una derrota en una de sus batallas, fue traicionado y entregado a las autoridades españolas, por lo que estuvo apresado hasta el día de su muerte, en La Carraca, prisión en San Fernando, Cádiz. Sus restos han compartido un osario común con otros muchos restos de esos mismos años. Estudios preliminares de antropología física han delimitado el número de restos ha clasificar.
Hasta ahora, se han podido obtener los perfiles de ADN mitocondrial de muchos de estos restos al igual que de algunos de los familiares del panteón común de sus descendientes. Pero aún queda un largo camino hasta finalizar este estudio. Son muchos los restos encontrados en ambos lugares y existe incertidumbre tanto en la muestra problema como en la de los familiares de referencia.
Biorrecuperación de especies
Uno de los problemas que se están ocasionando en la sociedad actual es la pérdida de biodiversidad. La extinción de muchas de las especies, que hace solo unas décadas habitaban nuestros bosques y ríos, es algo que ocurre cada vez con más frecuencia.
Una de las especies con mayor peligro de extinción es el esturión. Gracias a la colaboración con el Departamento de Genética, se ha podido participar en un proyecto para la recuperación de esta especie en el río Guadalquivir.
Los esturiones (Familia Acipenseridae) se distribuyen exclusivamente en el hemisferio Norte. Están considerados fósiles vivientes, existiendo actualmente una disminución continua de estos peces en las poblaciones, hasta el punto de ser consideradas especies en peligro de extinción.
Se conoce la existencia de la especie A. sturio en la península Ibérica, pero hasta hace poco se creía que la especie A. naccarii era endémica del Mar Adriático, y que no formaba parte de las especies características en la península Ibérica.
Al estar extinguidas estas especies en el Guadalquivir, se recurrió a piezas de museo disecadas o guardadas en formol. Se analizaron tres especies capturadas en el río Guadalquivir de mediados de los 70 y principios de los 80, conservadas en la Estación Biológica de Doñana, una del río Ebro, del Instituto Botánico de Barcelona y uno más del Departamento de Zoología de la Universidad de Granada.
Se creó un protocolo específico para la extracción de ADN de estos esturiones, consiguiendo resultados en todos ellos. Así, dos de ellos fueron A. naccarii, otros dos A. sturio y el quinto un híbrido de ambos. Con lo que se concluyó que la especie A. naccarii coexistía en España con la A. sturio.
Estos datos permitieron comenzar la recuperación de la especie en el Guadalquivir con la especie A. naccarii. En este momento se está trabajando con éxito en cautividad en la piscifactoría "Sierra Nevada".
Tras estos primeros éxitos, durante este último año se ha comenzado una nueva línea de investigación a partir de restos fósiles de esturiones encontrados en yacimientos arqueológicos. De esta manera se intenta resolver la disyuntiva de cuál de las especies actuales era la autóctona de la península Ibérica (Figura 3).
Caracterización de elementos históricos
Gracias a la colaboración con el Departamento de Pintura de la Universidad de Granada se ha podido trabajar con uno de los dos ejemplares del Corán más antiguos que se conservan en España. Se trata de la edición facsímil de un Corán Andalusí del siglo XIII, recuperado en un hallazgo fortuito en la localidad malagueña de Cútar.
El trabajo de nuestro grupo ha consistido en la caracterización del pergamino en el que estaba editado este ejemplar y determinar el origen de la piel con el que se había realizado la edición del Corán.
El pergamino es un material hecho a partir de la piel de una res u otros animales, fabricado para poder escribir sobre él, mediante un proceso de eliminación del vellón, adobado, blanqueado y tras un estiramiento final se consiguen las láminas con las que se elabora el libro.
La antigüedad y el valor de éste no permitían tomar un fragmento completo de una hoja del Corán, así que, utilizando las técnicas más avanzadas dentro del campo de la genética forense, se tomaron como muestras de partida los pelos del animal que subsistían sobre el pellejo manufacturado.
Se ajustaron los protocolos de extracción de pelos23 adaptándolos a este tipo de material tan manipulado. Aún más crítico fue el diseño de primers24, se escogió el ADN mitocondrial de las distintas especies candidatas "Ovis aries, Bos taurus, Sus scrofa domestica y Capra aegagru". El tamaño del amplicón escogido fue menor a las 200pb. Se ajustaron todos los protocolos de amplificación y secuenciación. Una vez obtenida la secuencia del material amplificado, se introdujo el resultado obtenido en BLAST (NCBI), Basic Local Alignment Search Tool, una herramienta gratuita que permite comparar las secuencias problema con una base de datos que contiene todas las especies caracterizadas hasta la fecha. Las secuencias obtenidas a partir de los pelos del pergamino produjeron una homología del 100% con la especie Bos taurus. Estos resultados nos permitieron caracterizar el tejido y determinar la especie con la que se había producido el soporte de esta edición del Corán.
Los resultados obtenidos han proporcionando el conocimiento necesario para ubicar su elaboración y el tratamiento de los materiales, tanto en este ejemplar como en otros que se elaboraron en torno al siglo XIII (Figura 4).
Aplicaciones en biomedicina
En el seno de las ciencias biomédicas, se veía a la medicina Legal como un campo poco relacionado con el resto, sin embargo, disciplinas como la genética han conseguido aunar y cooperar con el resto. Dentro del campo forense, uno de los pilares más relevantes y determinantes en ciertos casos y tipos de muestras es la genética. Si bien, en el campo de la biomedicina, se han observado también cada vez más tipos de enfermedades en las que juega un papel fundamental y pueden ser explicadas por esta ciencia. Ejemplos de esto son, enfermedades como la diabetes, el cáncer, la esquizofrenia Gracias a que en el campo de la genética forense se trabaja con muestras en pequeña cantidad y normalmente en condiciones poco óptimas, se han conseguido optimizar al máximo las técnicas de extracción y procesamiento de las muestras forenses para obtener un mayor rendimiento de las mismas. Estas técnicas tan eficientes pueden extrapolarse en los casos más complejos de muestras biomédicas (como por ejemplo en cáncer), obteniendo unos resultados con una calidad muy elevada, difícil de igualar con otras técnicas de extracción o procesamiento. Sin embargo, no sólo nos valdremos de las técnicas mediante las que tratamos las muestras para la obtención de la información genética, sino que también se emplearán las mismas regiones del ADN. Los STRs (Short Tandem Repeats), o microsatélites, que se han usado en medicina forense por mostrar elevados niveles de polimorfismos a nivel poblacional, no obstante el análisis de los STRs es también ampliamente usado en el mapeado de enfermedades genéticas o en estudios de inestabilidad genética de tumores25.
Caracterización de muestras provenientes de tumores
En las muestras tomadas a partir de tejidos cancerígenos o muestras oncológicas se pueden destacar dos tipos de alteraciones frecuentes. Una de las alteraciones genéticas más comúnmente encontradas en los tejidos cancerosos son las LOH (pérdida de heterocigosidad, del inglés Loss of heterocigosity) y las MSI (inestabilidad de microsatélites, del inglés Microsatellite inestability). Como se sabe, los microsatélites son repeticiones múltiples de 1 a 5 nucleótidos que están distribuidas por todo el genoma. Debido a su estructura repetitiva, son susceptibles a errores durante el proceso de replicación. La inestabilidad en microsatélites (MSI) se origina por un mecanismo de reparación postreplicativo erróneo. Se caracteriza por una contracción o expansión en la longitud de los STR. Hoy en día, se conocen varios genes cuyos productos están involucrados en este mecanismo y que están mutados en determinados tumores, como es el caso del cáncer colorrectal hereditario no polipoide. En clínica, se han diferenciado ya dos tipos de MSI, MSI-low (MSI-L) y MSI-high (MSI-H), según la incidencia del loci inestable26.
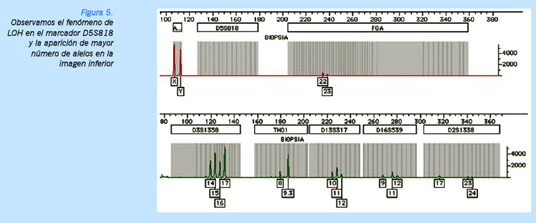
El fenómeno de LOH es relativamente frecuente en tejidos tumorales, y está ocasionado por deleciones que causan inestabilidad cromosómica manifestada en el desarrollo de tumores con aneuploidía y pérdidas frecuentes de heterocigosidad (LOH) en múltiples locus, así como mutaciones que activan oncogenes e inactivan o bloquean genes supresores (Figura 5).
Todo esto es interesante conocerlo ya que va a suponer un problema a la hora de intentar obtener información genética del tejido o muestra en cuestión. Si se analizan los STRs típicamente estudiados en genética forense, nos podemos encontrar ante la situación de que algunos de ellos se vean afectados por el proceso de LOH, y por tanto obtener falsos homocigóticos o heterocigóticos para determinados marcadores.
Gracias a que se ha ampliado el análisis y uso de los STRs al mapeado genético de enfermedades y al estudio genético de inestabilidad en tumores, se ha comprobado que la incidencia de inestabilidad genética afecta al conjunto de STRs que se usan comúnmente en identificación forense Esto se ha corroborado con varios estudios en muestras de pacientes con cáncer27. De hecho, se han encontrado alteraciones en STRs en los que existe un tamaño de nuevos STRs que oscila entre -4 y +4 unidades de repetición, y otras muestras en las que se han encontrado alelos intermedios en determinados marcadores. Incluso el marcador de la amelogenina, se ha hallado alterado en determinadas situaciones, encontrándose casos en los que determinados varones se han genotipado como XX. Este último ejemplo, ocurre en situaciones en las que la altura del cromosoma Y desciende por debajo de un 20% con respecto al alelo correspondiente al cromosoma X, por lo que si se analiza la muestra con un rango del filtro elevado, perderíamos el cromosoma Y obteniendo un genotipo erróneo en ese marcador. Vemos que hay que mantener precauciones tanto en el filtrado como en la eliminación de extrapicos en los casos de muestras oncológicas ya que vamos a encontrar con una mayor asiduidad degradaciones de la muestra en gran cantidad de marcadores alélicos 25.
En estudios de caracterización realizados por nuestro grupo de investigación en colaboración con la red de Bancos de Tumores de la Junta de Andalucía, se ha observado la aparición de los fenómenos anteriormente citados al estudiar las líneas tumorales. El genotipado de cada una de las distintas muestras de estas líneas ofrece un perfil característico. Se comprueba la aparición de gran cantidad de extrapicos, que no alcanzan una altura muy elevada, pero que no deben excluirse puesto que son los que nos indican de la presencia de fenómenos tumorales en esa muestra, manifestada por la alteración de las regiones de ADN analizadas.
Un segundo fenómeno que puede aparecer en estas muestras es la alteración en sus genotipos. En los casos de LOH, no se han detectado, hasta el momento, que ambos alelos se delecionen, pero en MSI, ambos alelos pueden verse afectados. Dependiendo de la etapa de expansión del tumor, los alelos originales pueden perderse y el tumor puede originar un nuevo genotipo.
Se han descrito, otro tipo de alteraciones al trabajar con muestras tumorales. Existen casos en los que, al tratar un tumor y suministrar al paciente inmunosupresores, esta situación de disminución de células linfocíticas hace que un tumor inactivo se manifieste. Con este proceso observamos dos situaciones; una de ellas, es la importante presión selectiva que realiza el sistema inmune evitando que se active una población celular tumoral existente en un individuo. Otra, es la problemática al realizar un genotipado del individuo, pues se ha comprobado en un caso de tumor renal que determinados alelos del individuo afectado se ven alterados, dificultando por tanto obtener un genotipo fiable. Sin embargo, esta situación se puede complicar más, se ha comprobado que tras un periodo de trasplante y tratamiento con inmunodepresores, el perfil genético que se obtiene del individuo trasplantado, coincide con el donante en determinadas regiones alélicas y en determinados tejidos28. Otro fenómeno similar se ha detectado al realizar la caracterización de una biopsia en el laboratorio de identificación genética de la Universidad de Granada. Desde la unidad de Genética, del hospital Infanta Cristina, se enviaron muestras de biopsias fijadas en parafina. Se recibieron estas muestras de las que existían sospechas previas de que un paciente trasplantado hubiese podido desarrollar un tumor proveniente de un tumor inicialmente del donante. Los estudios previos realizados al analizar la biopsia del tejido tumoral del paciente trasplantado revelaron que el tumor de este no era un tumor primario. El análisis realizado en Badajoz indicó de igual manera que su origen era embrionario. Los análisis realizados en nuestro laboratorio manifestaron que la muestra cancerígena del paciente poseía un perfil que presentaba un cambio con respecto a la muestra del paciente. En esta muestra de la biopsia aparecieron nuevos picos que no se podían explicar con los fenómenos descritos anteriormente (LOH y MSI). La muestra una vez extraída ofreció un perfil de mezcla. El trabajo forense habitual del grupo, al estar acostumbrados a manipular muestras con mezclas más o menos equilibradas, nos hizo ver que en la muestra de la biopsia aparecía el perfil de otro individuo. Este perfil coincidía con el perfil genético del órgano trasplantado (el perfil del donante). Las sospechas de nuestro equipo y de la unidad de Genética se hicieron firmes al ver los perfiles de la biopsia del tumor, del órgano trasplantado y del paciente, poniendo de manifiesto que el tumor que sufría el paciente provenía de un tumor metastásico del donante. Este caso alerta de la necesidad de nuevas metodologías para el cribado de órganos trasplantados, para poder asegurarnos que junto con el órgano no exista ningún tipo de célula maligna acompañante, ya que el órgano del donante había pasado los protocolos previos necesarios para el trasplante. En los casos de determinadas muestras oncológicas el procedimiento de análisis de las mismas es muy similar al usado con muestras de mezclas, típicas de agresiones sexuales. En estos casos, también vamos a encontrar mezcla de varios perfiles genéticos y debemos de ser precavidos a la hora de realizar un filtrado al analizar la muestra, pues podemos eliminar el perfil que está presente en un menor porcentaje y por tanto perder información genética de relevante importancia. Situaciones análogas las encontramos en los casos en que tratemos con muestras oncológicas similares a las descritas con anterioridad.
En el caso del cáncer, el ejemplo anterior no es el único donde se ha demostrado la existencia de células tumorales circulantes. Varios estudios presentan resultados que apoyan la hipótesis de la existencia de células tumorales circulantes en sangre periférica o médula ósea en pacientes con cáncer de mama que se encuentran en la fase previa a la manifestación de metástasis29. Esto puede complicar un proceso de identificación genética cuando se recurra a una muestra de sangre y se obtenga información genética afectada por este fenómeno patológico, donde se pueden encontrar poblaciones de células afectadas por el tumor y poblaciones inalteradas. Integrantes de nuestro grupo de investigación en colaboración con investigadores del centro GENyO están desarrollando con éxito una línea de investigación en esta materia, consiguiendo caracterizar células tumorales circulantes.
Como vemos, ante muestras procedentes de individuos con cáncer debemos de tomar muchas precauciones a la hora de interpretar los resultados genéticos. Esto se agrava sabiendo que esta patología ocupa un porcentaje elevado en la población, y si se recurre al genotipado en el caso en el que el individuo desconoce estar afectado. Otros de los factores a tener en cuenta es la posible problemática a la hora de la repetición de la muestra, ya que tal vez el material afectado puede ser limitado. Si bien, en los casos en los que se trabaja con muestras cancerosas, se recomienda el cotejado de los resultados con más de una extracción para evitar dar información genética errónea, en estos casos en los que la interpretación de los perfiles genéticos de un individuo puede ser muy compleja.
Asociación de enfermedades a haplotipos genéticos
En determinadas patologías, como en la diabetes tipo-2, se han encontrado mutaciones puntuales que parecen jugar un papel principal en el desarrollo de la enfermedad. Así mediante la búsqueda y caracterización de SNPs, podemos realizar un estudio detallado de la enfermedad al comparar individuos control con pacientes afectados30. Casos similares se han reportado en un estudio de esquizofrenia en los que cuatro SNPs y haplotipos determinados se han demostrado que generan un aumento en la asociación con esquizofrenia31. El papel de los haplogrupos (grupos poblacionales relacionados con el ADNmt) ha servido como clave para eventos como el origen del hombre moderno. A su vez, nos da idea de las similitudes entre unas poblaciones y otras por compartir haplogrupos mitocondriales. Actualmente, aunque siguen teniendo gran importancia en el campo de la genética de poblaciones, están ganando relevancia en el área de la biomedicina, ya que se ha visto que determinadas enfermedades tienen una mayor prevalencia en algunos haplogrupos.
Estudios enfocados con este nuevo punto de vista se están llevando a cabo con el cáncer de próstata en el Laboratorio de Identificación Genética de la Universidad de Granada en colaboración con LORGEN G.P. Se poseen muestras de saliva y de sangre de individuos controles y de individuos afectados por esta patología, intentando realizar un estudio para señalar los SNPs más predominantes entre la población afectada. Gracias a este estudio, se podrá realizar un panel de SNPs muy útil para prevenir y diagnosticar precozmente el cáncer de próstata familiar, sustituyendo el uso actual del PSA como predictor de esta patología (Figura 6).
Agradecimientos
Todos estos proyectos no podrían haber sido realizados sin las personas que han trabajado, y actualmente siguen colaborando con nuestro equipo en todos y cada uno de los estudios e investigaciones citados anteriormente:
- Ramón Cortés Márquez, Teniente coronel, Jefe de la Comandancia de la Guardia Civil de Ceuta y José A. García Sánchez-Molero, Jefe del Servicio de Criminalística.
- Profs. Bruce Budowle y Arthur Eisenberg, Centro de Identificación Humana de la Universidad del Norte de Texas.
- Marcial Castro y Sergio Algarrada, Impulsores del proyecto Colón.
- Miguel Botella, e Inmaculada Alemán del Laboratorio de Antropología Física.
- Francisca Robles y Manuel Ruiz Rejón, Departamento de Genética de la Universidad de Granada.
- Teresa Espejo Arias, Departamento de Pintura de la Universidad de Granada.
- Raquel Rodríguez López. Unidad de Genética del Hospital Infanta Cristina, Badajoz.
- José Manuel Cózar. Hospital Universitario Virgen de las Nieves. Granada.
- María José Serrano y Ana Fernández. GENyO - Pfizer-Universidad de Granada-Junta de Andalucía Centro de Genómica y Oncología.
- Y especialmente a nuestros compañeros Carmen Entrala Bernal, Francisco Fernando Rosado, Encarnación García Ruíz y Esther Martínez Espín. LORGEN GP.
Bibliografía
1. Lorente JA, Alvarez JC, Entrala C, Martinez-Espin E, Fernandez-Rosado F, Lorente M, et al. Bases de datos de ADN: Su uso en la investigación criminal y en la identificación civil. Anotaciones prácticas para su desarrollo. Forénsica 2002;1:31-44. [ Links ]
2. Lorente JA, Entrala C, Alvarez JC, Lorente M, Arce B, Heinrich B, et al. Social benefits of non-criminal genetic databases: Missing persons and human remains identification. Int J Legal Med 2002;116:187-90. [ Links ]
3. Lorente JA, Entrala C, Alvarez JC, Arce B, Heinrichs B, Lorente M, et al. Identification of missing persons: The Spanish "Phoenix" program. Croat Med J 2001;42:267-70. [ Links ]
4. da Silva LAF, Vilaça W, Azevedo D, Majella G, Silva IF, Silva BF. Missing and unidentified persons database. Forensic Sci Int: Genetics Supplement Series 2009;2:255-7. [ Links ]
5. Huffine E, Crews J, Kennedy B, Bomberger K, Zinbo A. Mass identification of persons missing from the break-up of the former Yugoslavia: Structure, function, and role of the international commission on missing persons. Croat Med J 2001;42:271-5. [ Links ]
6. United Nations. Office of Drugs and Crimes. Toolkit to Combat Trafficking in Persons, 2009. [ Links ]
7. UNODD, UN. GIFT - Global Initiative to Fight Human Trafficking, 2009. [ Links ]
8. UNICEF, UNICEF-Unite for Children, 2004. [ Links ]
9. S.L. AgA System, DNA-Prokids, 2009. [ Links ]
10. Alaeddini R, Walsh SJ, Abbas A. Forensic implications of genetic analyses from degraded DNA-A review. Forensic Sci Int: Genetics 2010;4:148-57. [ Links ]
11. Nascimento E, Cerqueira E, Azevedo E, Freitas V, Souza G, Pinheiro M. Genotyping of DNA samples under adverse conditions of Low Copy Number-LCN (formolisados tissue samples and embedded in paraffin). Forensic Sci Int: Genetics Supplement Series 2009;2:155-6. [ Links ]
12. Gamba C, Baeza C, Fernández E, Tirado M, López-Parra AM, Arroyo-Pardo E. Validation of the MiniFiler Kit in archaeological samples. Forensic Sci Int: Genetics Supplement Series 2009;2:17-8. [ Links ]
13. Nakamura S, Murakami C, Maeda K, Kobayashi M, Irie W, Wada B, et al. Analysis of AmpFlSTR® MiniFiler loci and its forensic application. Forensic Sci Int: Genetics Supplement Series 2009;2:19-20. [ Links ]
14. Loreille OM, Diegoli TM, Irwin JA, Coble MD, Parsons TJ. High efficiency DNA extraction from bone by total demineralization. Forensic Sci Int: Genetics 2007;1:191-5. [ Links ]
15. Kim W, Yoo T, Kim S, Shin D, Tyler-Smith C, Jin H, et al. Lack of association between Y-chromosomal haplogroups and prostate cancer in the Korean population. PLoS ONE 2007;2. [ Links ]
16. Seo SB, Zhang A, Kim HY, Yi JA, Lee HY, Shin DH, et al. Technical note: Efficiency of total demineralization and ion-exchange column for DNA extraction from bone. Am J Phys Anthropol 2010;141:158-62. [ Links ]
17. Mitchell D, Willerslev E, Hansen A. Damage and repair of ancient DNA. Mutation Research- Fundamental and Molecular Mechanisms of Mutagenesis 2005;571:265-76. [ Links ]
18. Dobberstein RC, Huppertz J, von Wurmb-Schwark N, Ritz-Timme S. Degradation of biomolecules in artificially and naturally aged teeth: Implications for age estimation based on aspartic acid racemization and DNA analysis. Forensic Sci Int 2008;179:181-91. [ Links ]
19. Rubio L, Martínez LJ, Martínez E, De Las Heras SM. Study of short- and long-term storage of teeth and its influence on DNA. J Forensic Sci 2009;54:1411-3. [ Links ]
20. Gill P, Ivanov PL, Kimpton C, Piercy R, Benson N, Tully G, et al. Identification of the remains of the Romanov family by DNA analysis. Nat Genet 1994;6:130-5. [ Links ]
21. Ivanov PL. Identification of the remains of the Romanov family: contribution of molecular genetics. Vestnik Rossijkoj Akademii Nauk 1996;66:310-6. [ Links ]
22. Ivanov PL, Wadhams MJ, Roby RK, Holland MM, Weedn VW, Parsons TJ. Mitochondrial DNA sequence heteroplasmy in the Grand Duke of Russia Georgij Romanov establishes the authenticity of the remains of Tsar Nicholas II. Nat Genet 1996;12:417-20. [ Links ]
23. Wilson MR, Polanskey D, Butler J, DiZinno JA, Replogle J, Budowle B. Extraction, PCR amplification, and sequencing of mitochondrial DNA from human hair shafts. BioTechniques 1995;14:992-9. [ Links ]
24. Ramadan HAI, El-Hefnawi MM. Phylogenetic analysis and comparison between cow and buffalo (including Egyptian buffaloes) mitochondrial displacement-loop regions. Mitochondrial DNA 2008;19:401-10. [ Links ]
25. Vauhkonen H, Hedman M, Vauhkonen M, Kataja M, Sipponen P, Sajantila A. Evaluation of gastrointestinal cancer tissues as a source of genetic information for forensic investigations by using STRs. Forensic Sci Int 2004;139:159-67. [ Links ]
26. Edelmann J, Lessig R, Hering S, Horn LC. Loss of heterozygosity and microsatellite inestability of forensically used STR markers in Human Cervical Carcinoma. Int Congr Ser 2004;1261:499-501. [ Links ]
27. Serrano Fernandez MJ, Alvarez Merino JC, Martínez Zubiaurre I, Fernández García A, Sánchez Rovira P, Lorente Acosta JA. Clinical relevance associated to the analysis of circulating tumour cells in patients with solid tumours. Clinical & translational oncology. 2009;11:659-68. [ Links ]
28. Cozar JM, Aptsiauri N, Tallada M, Garrido F, Ruiz-Cabello F. Late pulmonary metastases of renal cell carcinoma immediately after post-transplantation immunosuppressive treatment: A case report. Journal of Medical Case Reports 2008;2. [ Links ]
29. Serrano Fernández MJ, Fernández García A, Álvarez JC, Lorente JA. Células tumorales circulantes en pacientes con cáncer de mama. Impacto Clínico 2009:25-6. [ Links ]
30. Martínez-Marignac VL, Valladares A, Cameron E, Chan A, Perera A, Globus-Goldberg R, et al. Admixture in Mexico City: Implications for admixture mapping of Type 2 diabetes genetic risk factors. Hum Genet 2007;120:807-19. [ Links ]
31. Liu J, Shi Y, Tang J, Guo T, Li X, Yang Y, et al. SNPs and haplotypes in the S100B gene reveal association with schizophrenia. Biochem Biophys Res Commun 2005;328:335-41. [ Links ]
![]() Dirección para correspondencia:
Dirección para correspondencia:
Dr. José A. Lorente Acosta
Laboratorio de Identificación Genética.
Departamento de Medicina Legal.
Avda. Madrid 11
18012 - Granada.
E-mail: jlorente@ugr.es
Recibido: 8.MAR.2010
Aceptado: 8.MAR.2010