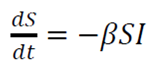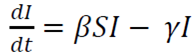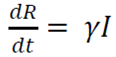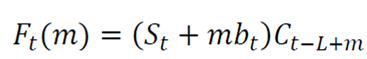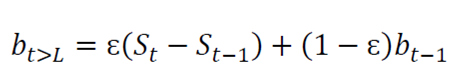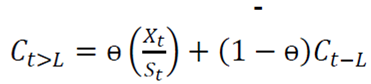INTRODUCCIÓN
Los modelos dinámicos son útiles para intuir el comportamiento de una infección cuando se manifiesta en una colectividad e investigar bajo qué escenarios será mitigada o erradicada 1. En la actualidad, COVID-19 es de gran preocupación para la ciencia, los gobiernos centrales, locales y todas las personas debido a la rápida propagación de la infección y la cantidad significativa de muertes que se producen 2. El virus que causa COVID-19 se transmite primariamente a través de gotitas generadas cuando un individuo infectado tose, estornuda o exhala. Estas gotas son densas para colgar en el aire y por efecto gravedad caen en la superficie 3. Los casos confirmados por coronavirus llegaron a casi cuatro millones en 187 países, y aproximadamente 295.000 personas han perdido la vida debido a este virus.
La evolución de la infección entre finales de enero e inicios de febrero de 2020 marca el inicio de un nuevo modelo de comportamiento para una nueva enfermedad de tipo infecciosa en el mundo, tomando protagonismo el Comité de Emergencia para el Reglamento Sanitario Internacional de la Organización Mundial de la Salud. El ente emitió el brote de SARS-CoV-2 como una Emergencia de Salud Pública de Importancia Internacional, ESPII. 4. Para el 06 de febrero, ya habían sido reportados 24554 casos confirmados en el planeta, de los cuales el 99,2 % se ubicaban en China, y el 0,8 % restante en Asia, Europa y Estados Unidos. A partir de ese momento se inicia la propagación del virus a nivel global.
Para el año 2020, la población peruana se estima en 32 millones 626 mil habitantes. La demografía del país indica que se tendrá 568 mil nacimientos y 192 mil defunciones sin incluir la infección de la COVID-19, a su vez una migración neta positiva de 57 mil pobladores. Por tanto, en el año 2020 la totalidad de población crecerá exponencialmente en 433 mil habitantes 5.
Los primeros casos de COVID-19 en Perú fueron confirmados el 10 de marzo, en la región Lima Metropolitana. Para el inicio de la investigación, el Perú registra 738020 infectados y 30927 muertos en su acumulado 6.
Los investigadores han estado observando la propagación del virus, se han congregado para acelerar diagnósticos lo más asertivos y están trabajando en una serie de vacunas para proteger contra el COVID-19 7. En el presente estudio se desarrolla un modelo epidémico de Kermack-McKendrick o también denominado SIR modificado para proyectar el número real de casos infectados; este medido con el parámetro umbral epidemiológico (R0) 8,9. La investigación tiene como objetivo, evaluar un modelo matemático de tipo diferencial, propio del comportamiento del COVID-19 para el colectivo peruano; partiendo de la experiencia en el control de infecciones de Kermack-McKendrick.
METODOLOGÍA
La investigación aplicó metodología de nivel teórico y empírico para la comprensión del conjunto de datos estadísticos COVID-19 de la OMS para el Perú, sustentado en el análisis documental y bibliográfico 10. Se utilizó la observación directa para la construcción de los indicadores, en conjunto con la modelación de Kermack-McKendrick.
El modelo SIR, que conecta el número de susceptibles S, infectados y diseminando la infección I y personas recuperadas R, se aplicó. Los parámetros de este modelo se estimaron con el uso del número individual y acumulado de casos 11. Validando los resultados con el suavizado exponencial Holt-Winters y así mesurar la bondad de ajuste del modelo 12. La hipótesis de la investigación es: El modelo de Kermack-McKendrick explica la velocidad de propagación de las cepas COVID-19 (B.1.617.2/P.1/C.37/B.1.621) en el contexto del colectivo peruano.
Modelo Kermack-McKendrick
El modelo Kermack-McKendrick es un modelo SIR (susceptibles, infectados, recuperados) para la cantidad de personas infectadas con COVID-19, aplicado en la República del Perú, partiendo del histórico entre el 07 de marzo y el 12 de septiembre de 2020, y proyectado hasta el 11 de septiembre de 2021. Se propuso para explicar el crecimiento y decrecimiento del número de pacientes infectados, tomando como valor representativo el umbral epidemiológico R013. Se asume que el tamaño de la población es fijo, el período de incubación del agente infeccioso de 5 a 14 días y la duración de la infectividad es la misma que la duración de la enfermedad 14.
El modelo se constituye de un sistema de tres ecuaciones diferenciales ordinarias no lineales articuladas:
Donde
t es el tiempo
S es el número de personas susceptibles
I es el número de pacientes infectados
R es el número de personas que se han recuperado y desarrollado, inmunidad a la infección
β es la tasa de infección
γ es la tasa de recuperación
Las condiciones para la interpretación del modelo son estas:
- Cuando R0 <1, cada persona que contrae la enfermedad infectará a menos de una persona antes de morir o recuperarse, por lo que el brote desaparecerá (dI/dt <0).
- Cuando R0> 1, cada persona que contraiga la enfermedad infectará a más de una, por lo que la epidemia se extenderá (dI/dt>0) 15.
El valor clave que rige la evolución temporal de estas ecuaciones es el llamado umbral epidemiológico,
El suavizado exponencial Holt-Winters
Holt-Winters es una técnica robusta para construir series de temporales con tendencia aditiva 16. La forma recursiva de la ecuación de suavizado exponencial de Holt-Winters se expresa así:
Donde:
Xt es el valor de la serie de tiempo en el tiempo t.
L es la longitud de la estación o duración.
St es un estimado del suavizado del componente de nivel.
bt es un estimado del suavizado del componente de tendencia.
Ct es un estimado del suavizado del componente de índice de temporada.
α es el nivel de coeficiente de suavizado.
ɛ es la tendencia del coeficiente de suavizado.
ɵ es el coeficiente de suavizado estacional.
Ft (m) es el valor pronóstico de suavizado en el paso adelantado m para X en el tiempo t.
Se tasaron tres series exponenciales simples no dependientes de nivel, tendencia y estacionales. Son interdependientes en el sentido de que los tres componentes deben actualizarse cada período. En el estudio se partió del histórico de infectados, muertes y recuperados en el Perú, con proyección de 52 semanas 17.
RESULTADO
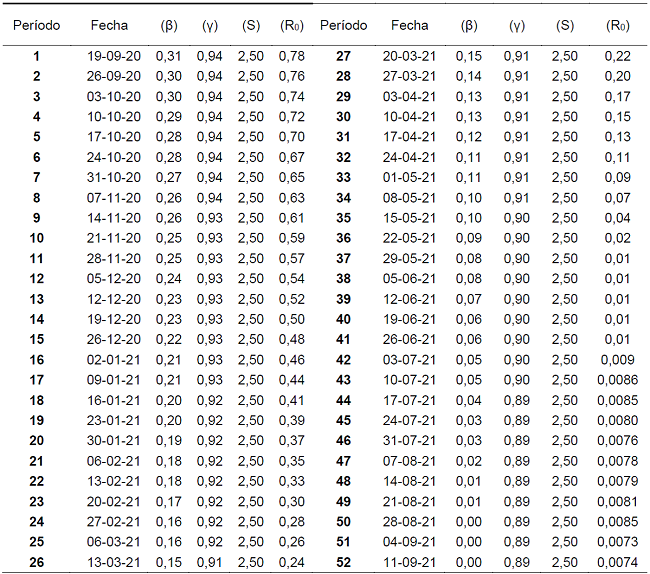
En la Figura 1, se evidencia la tendencia en comportamiento de la tasa de infectados β, tasa de recuperación γ, número de personas susceptibles S y el umbral epidemiológico R, calculado por el modelo de Kermack-McKendrick; para 28 semanas de pandemia COVID-19 desde el 07 de marzo al 12 de septiembre de 2020. La semana 1 representa un R0 de 2.50, pero que a la semana 5 se ubicó en 1,62, incrementándose en la semana 7 en 1,81 y así sucesivamente hasta llegar a la semana 28 con un valor de 0,86. Este valor representa la cantidad de personas que son infectadas por un contagiado. Por lo tanto, como R0 <1, cada persona que contrae la enfermedad infectará a menos de una persona antes de morir o recuperarse, por lo que el brote desaparecerá.
Posteriormente, con los datos obtenidos de Kermack-McKendrick se proyectó el comportamiento del COVID-19, con suavización exponencial durante 52 semanas; estimando el umbral R0 y predecir la mitigación y erradicación de la enfermedad.
>Modelo de proyección SIR
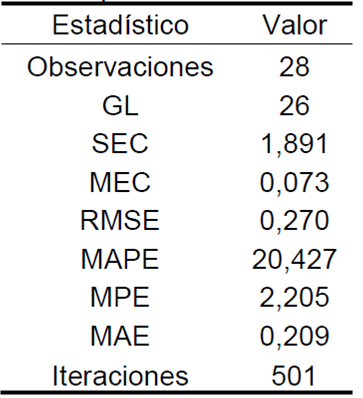
En la Tabla 1, el MAPE estima en 20,427 el porcentaje promedio del error de pronóstico un paso adelante para el Umbral epidemiológico R0, en promedio el pronóstico es acertado en una diferencia porcentual de 79,573. El valor MPE para las series es 2,205 y manifiesta el error porcentual medio, es decir entre la predicción y los valores reales. En este sentido, el pronóstico futuro para finales del año 2021 cuantifica una exactitud del modelo en 97,795 %, estos son los estadísticos que hacen fiable la predicción.
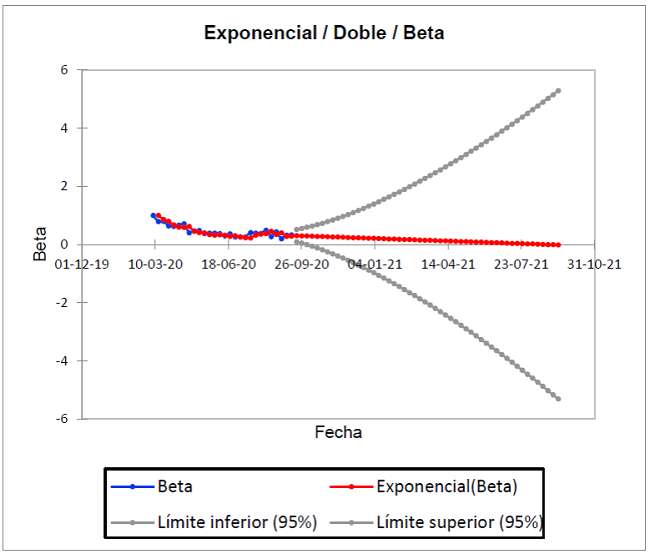
En la Figura 2, se demuestra la predicción de la tasa de infección β a partir del histórico de la pandemia, desde el 7 de marzo hasta el 12 septiembre de 2020 y proyectada hasta el 11 de septiembre del año 2021. Como se ve en los puntos de atenuación azul; pues hay una tendencia de decrecimiento en la pendiente de distribución desde el 08 de agosto del año 2020 (β=0.379), con un segundo pico dos semanas después al 22 de agosto (β=0.354), que varía según la cantidad de infectados diarios.
Los puntos grises muestran el mejor y el peor escenario. Es importante observar en el modelo ideal de Kermack-McKendrick, que estima para el mejor, una disminución de los infectados a partir del 16 de enero del 2021 y para el peor, un incremento a partir del 21 de diciembre del 2021. Ahora, la tendencia real está explicada en la línea de tendencia roja, cuyo pronóstico con base en el histórico es de 0 infectados para el 28 de agosto de 2021 (β=0.001). Se debe explicar que el número de personas susceptibles es constante 2,5, es decir, si las medidas de confinamiento se flexibilizan; hay mayor probabilidad de contagios.
En la Figura 3, se demuestra la predicción de recuperados γ a partir del histórico de la pandemia, desde el 7 de marzo hasta el 12 septiembre de 2020 y proyectada hasta el 11 de septiembre del año 2021. Como se ve en los puntos de atenuación azul; pues hay un incremento pronunciado en la pendiente de distribución en dos fechas; 22 de agosto (γ=0.987) y 05 de septiembre (γ=0.984) de 2020, pero que desciende el 12 de septiembre (γ=0.972).
Los puntos grises muestran el mejor y el peor escenario. Es importante observar en el modelo ideal de Kermack-McKendrick, que estima para el mejor, una disminución de los recuperados a partir del 24 de octubre de 2020 y para el peor, un incremento a partir del 16 de enero del 2021. Ahora, la tendencia real está explicada en la línea de tendencia roja, cuyo pronóstico con base en el histórico es de 88,50% de recuperados para el 11 de septiembre de 2021. Esto quiere decir, que al menos 88,50 personas se recuperaran por cada 100 infectados.
En la Figura 4, se demuestra la predicción de defunciones a partir del histórico de la pandemia, desde el 7 de marzo hasta el 12 septiembre de 2020 y proyectada hasta el 11 de septiembre del año 2021. Como se ve en los puntos de atenuación azul; pues hay una disminución pronunciada en la pendiente de distribución desde el 05 de septiembre del año 2020 (R0=0.724), pero que desciende desde el 5 de septiembre, incrementándose en la semana del 12 de septiembre nuevamente (R0=0.738).
En el modelo ideal de Kermack-McKendrick, la tendencia real está explicada en la línea roja, cuyo pronóstico sobre la base del histórico es (R0=0.000) para el 29 de mayo de 2021. Cuando R0 <1, cada persona que contrae la enfermedad infectará a menos de una persona antes de morir o recuperarse, por lo que el brote desaparecerá; si R0> 1, cada persona que contraiga la enfermedad infectará a más de una. Sobre la base de esto, el resultado de la simulación exponencial para el 29 de mayo explica que no habrá contagiados ni susceptibles, considerándose erradicada la enfermedad. Estos resultados son representativos de 501 iteraciones del modelo.
La Tabla 2 evidencia las predicciones de las próximas 52 semanas de comportamiento del COVID-19, en relación con el histórico de datos del modelo Kermack-McKendrick. Los resultados son estadísticamente significativos, pues el valor del umbral epidemiológico (R0=0,00) para la semana del 29 de mayo de 2021, representa la erradicación de la enfermedad, con una tasa de infectados (β=0,08). Esta a su vez disminuirá por completo (β=0,00) para la semana del 28 de agosto de 2021.
En la Figura 5, se modela la curva de contagio del COVID-19 desde el 14 de marzo de 2020 al 11 de septiembre de 2021 para la República del Perú; con la representatividad de tres picos de crecimiento en un mismo mes, específicamente para agosto de 2020, con una tendencia decreciente para los primeros días de septiembre. Sin embargo, vuelve a experimentar un pico el 19 del mismo mes. Este ha sido el comportamiento de la pandemia durante seis meses continuos de medidas de confinamiento de parte del gobierno central.
Finalmente, el pronóstico de la simulación Holt-Winters, explica que si se mantiene el decrecimiento en la curva de contagio; esta experimentará su final para el 29 de mayo de 2021, cuyo punto de corte con el eje de fechas se observa en la Figura. Las estimaciones son consistentes con el análisis epidemiológico de las primeras 28 semanas de pandemia en el Perú 18. En este sentido, se confirma la hipótesis de investigación: El modelo de Kermack-McKendrick, explica la velocidad de propagación del COVID-19 en el contexto del colectivo peruano.
Con base en los resultados del estudio, se recomienda adoptar medidas de mitigación flexibilizadas a partir del 10 de abril de 2021, donde se observa la menor transmisibilidad de las cepas (B.1.617.2/P.1/C.37/B.1.621). El modelo de Kermack-McKendrick desde el 12 de septiembre de 2020, mantiene el umbral de contagio con un valor R0 <1, por lo tanto, cada persona que contrae la enfermedad infectará a menos de una persona antes de morir o recuperarse, por lo que la transmisibilidad decrecerá.
DISCUSIÓN
Los autores han desarrollado un modelo de simulación dinámica para la República del Perú en el período 2020-2021; este es un simulado analítico basado en los principios SIR de Kermack-McKendrick, con datos históricos de la COVID-19 Dashboard by the Center for Systems Science and Engineering (CSSE) at Johns Hopkins University (JHU) 19, y proyectado con tendencia aditiva del suavizado exponencial de Holt-Winters. El modelo diferencial del estudio explicó las tasas de susceptibles, infectados y recuperados con un nivel de precisión del 79,5 %, una diferencia de un punto porcentual en comparación con los resultados de Viña y Pernalete 20, quienes demostraron un acierto del 80,5 %. La diferencia entre ambos modelos radicó en las cepas, para esta investigación se tomaron a (B.1.617.2/P.1/C.37/B.1.621), mientras el comparado solo tomó a (B.1.617.2/P.1/C.37). Es decir, se incluyó a la variante B.1.621 (Mu), detectada en enero de 2021 en Colombia. Es razonable que, al incluir una cepa adicional, el porcentaje de mesura disminuyera.
Los resultados están en concordancia con los de Kucharski et al. 21, el modelo aplicado en Wuhan, China; registró un crecimiento exponencial en los casos que surgieron a principios de enero 2020, un número creciente de casos de exportación entre el 15 y el 23 de enero de 2020, y la tasa de infección se midió en diez vuelos de evacuación desde Wuhan a siete países; donde el 94,8 % de la población seguía siendo susceptible al 31 de enero de 2020. Para el caso de susceptibles en Perú, la dinámica arrojó un 94% hasta el primer semestre de 2020, con disminución a 91% hasta septiembre de 2021. Estos resultados pueden estar influenciados por los protocolos de emergencia en la atención primaria de pacientes con COVID-19, ya que la evolución de la infección hizo necesario comprobar que el paciente es negativo en coronavirus por Reacción en Cadena de la Polimerasa (PCR) y, por tanto, corroborar que ya no tiene el virus.
En consecuencia, los autores Ramírez et al. 22 describieron los comportamientos del segundo y tercer rebrote epidémico del COVID-19 para la provincia Santiago de Cuba aplicando el modelo SEIR. Los resultados de la simulación dan evidencia en la disminución de los casos activos, con variación en la curva de reproducción a una pendiente más negativa, descubrimiento que se observó a partir del 26 de septiembre de 2021. Este cambio de pendiente sugiere la preexistencia de factores sinérgicos que contrapesan el crecimiento de la infección, acaecen a la par y preponderan en escalas prospectivas.
De esta manera, en Perú la tendencia real está explicada en el pronóstico de 88,50% de recuperados para el 11 de septiembre de 2021, en concordancia con los autores. Esto quiere decir, que al menos 88,50 personas se recuperaran por cada 100 infectados. Ambos estudios permiten inferir que el pico de la infección (cresta de la ola) toma en promedio 12 semanas, con una constante en su reproducción de 26 semanas desde la aparición, y un descenso o menor transmisibilidad a partir de la semana 40.
En estudio llevado a cabo en Uganda por Mugisha et al. 23, los resultados mostraron que esta enfermedad no va a desaparecer, incluso plantearon que el escenario de reducir a la mitad el número de importaciones no tendrá un efecto inmediato, ya que la epidemia seguirá creciendo exponencialmente durante varios meses, la enfermedad persistirá en el país más allá del tiempo de simulación de 1 año. Aún más preocupante es el hecho de que la prevalencia fue mayor en los niveles más altos de la población susceptible. Similares hallazgos ocurrieron en Perú, el modelo SIR aplicado durante 52 semanas evidenció una tendencia de infectados en decrecimiento de 31% a 15%, bajo el fiel cumplimiento de las normas de confinamiento sociales de importación del virus.
En el estudió se confirmó que las cepas (B.1.617.2/P.1/C.37/B.1.621), tuvieron una menor transmisibilidad de la infección hasta noviembre de 2021, donde su agresividad disminuyó en promedio a (Ro = 0,24). No obstante, en esa fecha da inicio la variante Ómicron (B.1.1.529), específicamente el 24 de noviembre en Sudáfrica y el 19 de diciembre de 2021 en Perú 24. La aparición de Ómicron en Perú quedó registrada el 19 de diciembre de 2021 en 10 regiones según Se debe destacar que para utilizar de manera inteligente las tasas de crecimiento basadas en valores específicos de R y tomar decisiones de salud pública asertivas, se deben abordar otras cuestiones, como la recopilación de datos que son necesarias para generar estimaciones estandarizadas con intervalos de confianza estrictos Ridenhour et al. 25.
Basado en la diferencia aleatoria de cepas mencionadas en esta investigación, Hellewell et al. 26 predijeron recientemente la eficacia del rastreo de contactos y aislamiento de casos de COVID-19 empleando patrones distintos de Ro. En analogía con los autores, la probabilidad de control disminuye la transmisibilidad con un mayor aislamiento, un menor número de casos comprobados por el rastreo de contactos, y el aumento de la transmisión antes de los síntomas.
Por lo tanto, el modelo de Kermack-McKendrick explicó la velocidad de propagación de las cepas COVID-19 (B.1.617.2/P.1/C.37/B.1.621) en el contexto del colectivo peruano, y da cabida a continuar con este modelado dinámico, pero con la inclusión de la cepa B.1.1.529 (ómicron). Los autores recomiendan hacer esta estimación para el segundo semestre del año 2022, fecha en la que los datos serían más precisos para el estudio.
CONCLUSIÓN
El patrón de comportamiento de las cepas (B.1.617.2/P.1/C.37/B.1.621) COVID-19 para la República de Perú, se modeló por medio de ecuaciones diferenciales de Kermack-McKendrick, específicamente para mesurar la variable umbral epidemiológico R0, también conocido como índice de la curva de contagio, que coadyuven en la valoración epidemiológica, es decir, determinar el número de personas infectadas por contacto con una sola persona infectada antes de su muerte o recuperación. La modelación se inició desde el 12 de septiembre de 2020; el pronóstico de las 52 semanas de pandemia evidenció un pronóstico de erradicación para el 29 de mayo de 2021 con una tasa de infectados casi nula (β=0.08), considerando la transmisión por contacto como una de las principales rutas del COVID-19.
En este sentido, el pronóstico para mediados del año 2021 cuantifica una exactitud del modelo en 97,795 %, estos son los estadísticos que hacen fiable la predicción; con un valor promedio R0 <1, por lo tanto, cada persona que contrae la enfermedad infectará a menos de una persona antes de morir o recuperarse, por lo que el brote desaparecerá. Como resultado, se confirma que el modelo de Kermack-McKendrick, explica la velocidad de propagación del COVID-19 en el contexto del colectivo peruano. La curva de contagio para el Perú dependerá directamente de las medidas de mitigación que tienen como fin, disminuir la velocidad de la propagación de la infección y anticiparse a la transmisión sostenida hasta tener una vacuna para suprimir el COVID-19; siempre que se implementen medidas preventivas por parte de la población.
En líneas generales, el mejor pronóstico del modelo de Kermack-McKendrick explicó que la menor tasa de infectados ocurrirá a partir del 3 de abril de 2021, donde la curva de contagio empezará a mostrar una pendiente de decrecimiento abrupta, hasta desaparecer por completo a mediados del mes de mayo de 2021. El modelo se ajusta al contexto de propagación de las cepas (B.1.617.2/P.1/C.37/B.1.621). No obstante, el inicio de ómicron en noviembre de 2021, con un patrón de transmisibilidad distinto a lo valorado en este estudio, es materia de estudio a mediano plazo, con la premisa de conocer la dinámica poblacional del virus SARS-CoV-2.











 texto en
texto en