My SciELO
Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Cited by Google
Cited by Google -
 Similars in
SciELO
Similars in
SciELO -
 Similars in Google
Similars in Google
Share
Sanidad Militar
Print version ISSN 1887-8571
Sanid. Mil. vol.68 n.2 Madrid Apr./Jun. 2012
https://dx.doi.org/10.4321/S1887-85712012000200004
Perfiles genéticos en muestras de orina en identificación sanitaria militar
Genetic profiles in urine samples in military medical identification
Marqués Negredo, M.L.1, Álvarez-Maldonado Paramés T.2, Villa Rodríguez L.3, García Tejerina R.3, Sanz Zamarro M.I.4, Coca Menchero S.5
1 Lda. en Biología (Genetista).
2 Lda. en Biología.
3 Tte. Enfermera.
4 Dra en Biología (Genetista).
5 Col. Médico.
Hospital Central de la Defensa "Gómez Ulla". 1-3, 5Servicio de Anatomía Patológica. 4Servicios Centrales. Madrid. España.
Dirección para correspondencia
RESUMEN
Antecedentes: Las muestras de orina que con frecuencia son utilizadas para análisis clínicos, toxicológicos o tets de control de dopaje, pueden verse mezcladas de forma inconsciente o manipuladas intencionalmente.
Objetivos: Disponer de una técnica que permita asegurar la identidad de las muestras de orina. Se propone la identificación del donante a partir de la comparación de perfiles genéticos en muestras de orina y sangre. Comparamos dos métodos de extracción de DNA nuclear.
Material y métodos: Extracción de DNA a partir de dos métodos, el protocolo Master Diagnostica (VITRO, S.A.) y el método automático MagNa Pure® de Roche. Se establece los perfiles genéticos según el AmpFlSTR® Identifiler® PCR Amplification Kit (Applied Biosystems).
Resultados: Se obtuvo 100 % de concordancias entre los perfiles de los dos tipos de muestras.
Conclusiones: Tanto de forma manual como automática se puede obtener DNA a partir de muestras de orina. Ambos métodos son válidos, si bien con el manual, se obtiene mayor cantidad de DNA. Se puede identificar al donante de una muestra de orina comparando los perfiles genéticos de la orina y de la sangre. La extracción del DNA de la orina y su posterior almacenamiento a -80oC permite el establecimiento del perfil genético a posteriori sin alteraciones.
Palabras clave: Perfil genético, extracción de DNA, identificación sanitaria.
SUMMARY
Antecedents: Urine samples that are often utilized for clinical, toxicological or doping control tests can be mistaken involuntarily or intentionally tampered with.
Objective: To make available a technique that ensures the identity of urine samples. We propose the identification of the donor through genetic profiling in blood and urine samples. Two methods of nuclear DNA extraction are compared.
Material and methods: DNA is extracted by two methods, the method of Master Diagnostica (VITRO S.A.) and the automatic method MagNa Pure® of Roche. The genetic profiles are established utilizing the AmpFlSTR® Identifiler® PCR Amplification Kit (Applied Biosystems).
Results: The correspondence between the profiles of both types of samples was 100%.
Conclusions: DNA can be obtained from urine samples manually or automatically. Both methods are valid although the manual method yields more DNA. The donor of a urine sample can be identified comparing the genetic profiles of urine and blood. The extraction of DNA from urine and its subsequent storage at -80oC allows for establishing later a genetic profile without alterations.
Key words: Genetic profile, DNA extraction, medical identification.
Introducción
La orina contiene sales y productos de desecho del organismo disueltos o en suspensión en agua, y como tales las drogas, ya sean naturales (cocaína, marihuana, etc) o de diseño (anfetaminas, ácido lisérgico, barbitúricos, etc) también se excretan y detectan fácilmente en orina.
En el caso del sedimento de la orina, en los individuos normales, es escaso y está formado por sales (uratos, fosfatos y carbonatos) con algunas células epiteliales de la vesícula y la uretra. En la mujer pueden presentarse también algunas células vaginales1.
Las células epiteliales de descamación de las vías urinarias se extraen de la orina por centrifugación y eliminando el sobrenadante. Con las técnicas actuales, se puede extraer DNA de cualquier vestigio humano por muy pequeña que sea la muestra2. En el estudio de grandes grupos de población es importante la recogida de muestras por métodos no invasivos3.
Las muestras de orina son con frecuencia utilizadas para análisis clínicos, toxicológicos o tests de control de dopaje. Estas muestras pueden verse mezcladas de forma inconsciente o manipuladas intencionalmente. Por lo tanto es importante proporcionar una técnica que permita asegurar la identidad de las mismas. También es importante la elección del marcador genético que se use en la identificación4.
En casos de dopaje en deportistas surge la cuestión de si el estudio de polimorfismos de DNA puede individualizar las muestras de orina5. La manipulación de las muestras de orina en los tests de drogas de los deportistas está considerado como una violación de las reglas anti-doping y consecuentemente es sancionado por la autoridad reguladora6. Las muestras son consideradas sospechosas de haberse manipulado cuando muestran perfiles esteroideos idénticos.
Hemos encontrado pocos estudios sobre identificación forense de muestras de orina y que ensayan si se puede determinar el perfil genético de las mismas para poder aplicarlo a la identificación posterior del donante. En el estudio de Brinkmann et al5 se propone este método como útil para la identificación del donante en muestras dudosas de orina. El estudio de Castella et al4 compara dos tipos de marcadores (DNA nuclear frente a DNA mitocondrial) para establecer el perfil genético de muestras de orina. Thevis et al6 muestran en su estudio una aproximación bastante similar a la establecida en nuestro estudio. En todos ellos las conclusiones son similares, a pesar de que actualmente, año 2011, no hemos encontrado que se utilice el perfil genético en orina de forma más rutinaria para la identificación de donantes.
Otros estudios como el de Cannas et al7 están más enfocados al aislamiento del DNA y a las consideraciones de almacenamiento y deterioro que sufre éste en las muestras de orina con el tiempo. Nosotros proponemos el aislamiento temprano a partir de la toma de la muestra y su reserva a baja temperatura (-80oC).
Objetivos
Elaboración del perfil genético del DNA obtenido a partir de las células de descamación que se encuentran en el sedimento de la orina y compararlo con la huella genética obtenida a partir del DNA extraído de muestras de sangre de los mismos individuos a fin de relacionar cada orina con el sujeto donante.
Comparar la extracción de DNA obtenido por método automático (MagNa Pure de Roche) frente al obtenido manualmente tras precipitación con fenol-cloroformo-isoamílico, mediante cuantificación con el kit Quantifiler (Applied Biosystems) en una reacción de PCR (reacción en cadena de la polimerasa) a tiempo real.
Material y métodos
En nuestro laboratorio (Unidad de Identificación Sanitaria - Servicio de Anatomía Patológica. Hospital Central de la Defensa "Gómez Ulla" de Madrid) se ha procedido a la recogida de orina y de sangre en soporte GSM (Gene Storage Matrix™) de personal voluntario, previamente informado sobre el estudio.
1. Primera prueba.
Se tomaron muestras de orina en recipientes estériles de 4 personas del Servicio de Anatomía Patológica. Tres mujeres y un varón. Asimismo se tomaron muestras de sangre de los mismos, recogidas en tarjetas GSM (Control-Biogen, S.L.), como las que se utilizan para la huella genética.
En un primer experimento, las muestras de orina se procesaron de dos formas diferentes. La extracción del DNA en una de las muestras se hizo por procedimiento automático (MagNa Pure® de Roche) y el resto según el protocolo para la extracción de DNA a partir de muestras de citologías líquidas de Master Diagnostica (VITRO, S.A.).
Siguiendo el protocolo para la extracción automática por el sistema MagNa Pure® de Roche, se tomaron 15 mL de orina que se centrifugó (1200 rpm, 10 minutos) y descartó el sobrenadante. Al pellet restante se le lavó tres veces en suero fisiológico, centrifugando y descartando el sobrenadante cada vez. Finalmente se resuspendió en 100 μL de suero fisiológico y esta alícuota se procesó en el MagNa Pure®.
Según el protocolo del "kit para extracción de DNA total" (No catálogo 003951M) de Master Diagnostica (VITRO, S.A.), se tomaron 500 μL de las muestras de orina y se centrifugaron a 12000 rpm durante 3 minutos. El proceso se repitió de tres a cuatro veces, hasta ver un sedimento de células epiteliales suficiente para la posterior extracción del DNA.
Estos sedimentos celulares fueron lavados dos veces con 500 μL de PBS (buffer salino fosfato), centrifugados a 12000 rpm durante 3 minutos y sometidos posteriormente a digestión con buffer de lisis y solución de proteasa-K. Se las mantuvo a 55oC en baño-maría durante toda la noche.
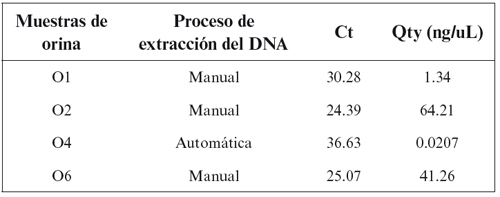
Al día siguiente se inactivó la proteasa-K por calor (95oC, 10 minutos), se centrifugó (12000 rpm durante 5 minutos) y se recogieron 50 μL de sobrenadante, que contiene el DNA de las células de descamación de las vías urinarias (Tabla 1).
Tabla 1. Cuantificación de las muestras de orina del primer experimento.
Threshold: 0.200000; Línea basal: 3-15; Slope: -3.504356; R2: 0.98946.
2. Segunda prueba.
Tras obtener los primeros resultados se repitió el proceso unificando las muestras de orina: se tomaron dos alícuotas de 10 mL de cada voluntario y se procesaron para la extracción del DNA de dos formas diferentes. Una extracción automática con el sistema MagNa Pure de Roche y otra mediante extracción manual que conllevó digestión enzimática, precipitación con fenol-cloroformo-isoamílico y lavado por columnas de membrana (Microcon 100).
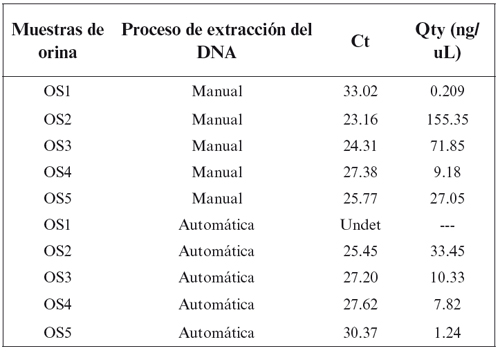
Ambas muestras se cuantificaron mediante una reacción de PCR a tiempo real y los reactivos de Applied Biosystems (Quantifiler). La reacción se llevó a cabo en un termociclador 7500 de Applied Biosystems, a fin de optimizar los procesos posteriores1,3. Los reactivos empleados para la reacción de PCR fueron el kit "Quantifiler™ Human DNA Quantification Kit" que incluye los controles de referencia, primers, buffer de reacción con dd-NTP, cloruro de magnesio y la enzima Taq polimerasa, y un control interno de la reacción (Tabla 2).
Tabla 2. Cuantificación de las muestras de orina. Valores obtenidos según
el proceso de extracción del DNA a partir de dos alícuotas de 10 mL de orina cada una.
Threshold: 0.201534; línea basal: 3-15; Slope: -3.43333; R2: 0.991991.
Según la concentración obtenida en la cuantificación para cada muestra, se tomaron hasta 10 μL del sobrenadante inicial para amplificar mediante PCR convencional el DNA obtenido de la lisis celular, siguiendo el protocolo del kit "AmpFlSTR® Identifiler®" (Applied Biosystems) y así determinar la huella genética del DNA de la orina. El AmpFlSTR® Identifiler® PCR Amplification Kit contiene el buffer de reacción con todo lo necesario para realizar la amplificación del DNA mediante PCR y los primers o cebadores correspondientes a los 203 STR que es capaz de determinar.
Los STR8 son polimorfismos de secuencia del DNA, llamados también microsatélites. Se trata de repeticiones en tándem de bloques de entre 1 y 7 pares de bases. Son altamente polimórficos y de herencia mendeliana simple. Son estables y pueden amplificarse mediante una reacción de PCR multiplex, la cual permite amplificar varios loci simultáneamente a partir de una muestra muy pequeña de DNA. Este polimorfismo surge porque el número de repeticiones que forma cada alelo (secuencia de DNA de un locus determinado) no es el mismo.
La estabilidad a la degradación de los STR es importante porque las muestras de orina presentan un grado de degradación alto debido a la contaminación bacteriana, confirmado en el estudio de Brinkmann et al5, siendo esta contaminación debida también al almacenaje de las muestras por más de cinco semanas a 4oC. Sin embargo la repercusión en el estudio con AmpFlSTR es prácticamente nula5.
Una vez amplificado el DNA de las células epiteliales de las vías urinarias, se realizó una electroforesis capilar con el analizador genético 3130 de Applied Biosystems. Los electroferogramas de STR correspondientes a las huellas genéticas de dichos DNA se procesaron con el software GeneMapper ID v3.2.
Los DNA obtenidos a partir de la orina se guardaron a -80oC con el fin de recuperarlos un tiempo después de su extracción, que en este caso fue de dos meses, para comprobar la viabilidad de los mismos al cabo de un tiempo.
Asimismo se realizó el perfil genético a las muestras de sangre de los individuos voluntarios, según protocolos habituales usando el AmpFlSTR® Identifiler® PCR Amplification Kit. Este proceso requiere la toma de dos discos o punches de 1 mm de diámetro del soporte GSM de las muestras de sangre. Estos discos se pasaron a un eppendorf limpio y se lavaron dos veces con un reactivo de purificación para FTA. Se les mantuvo 5 minutos con 200 μL de reactivo a temperatura ambiente, agitando suavemente. Al cabo de ese tiempo se eliminó el sobrenadante y se añade otros 200 μL del reactivo de purificación, manteniéndoles otros 5 minutos a temperatura ambiente con agitación suave. Se hizo lo mismo con 200 μL de buffer TE hasta que los discos aoarezcan blancos, se eliminó todo el sobrenadante y se dejaron secar al menos dos horas.
Sobre los discos secos, se añadió 10 μL de agua destilada y los reactivos para la PCR de amplificación de los STR: AmpFlSTR ® Identifiler® PCR Amplification Kit.
Una vez amplificadas las muestras de sangre, se llevaron al analizador genético 3130 (Applied Biosystems) y se obtuvieron los electroforogramas de los marcadores genéticos9,10.
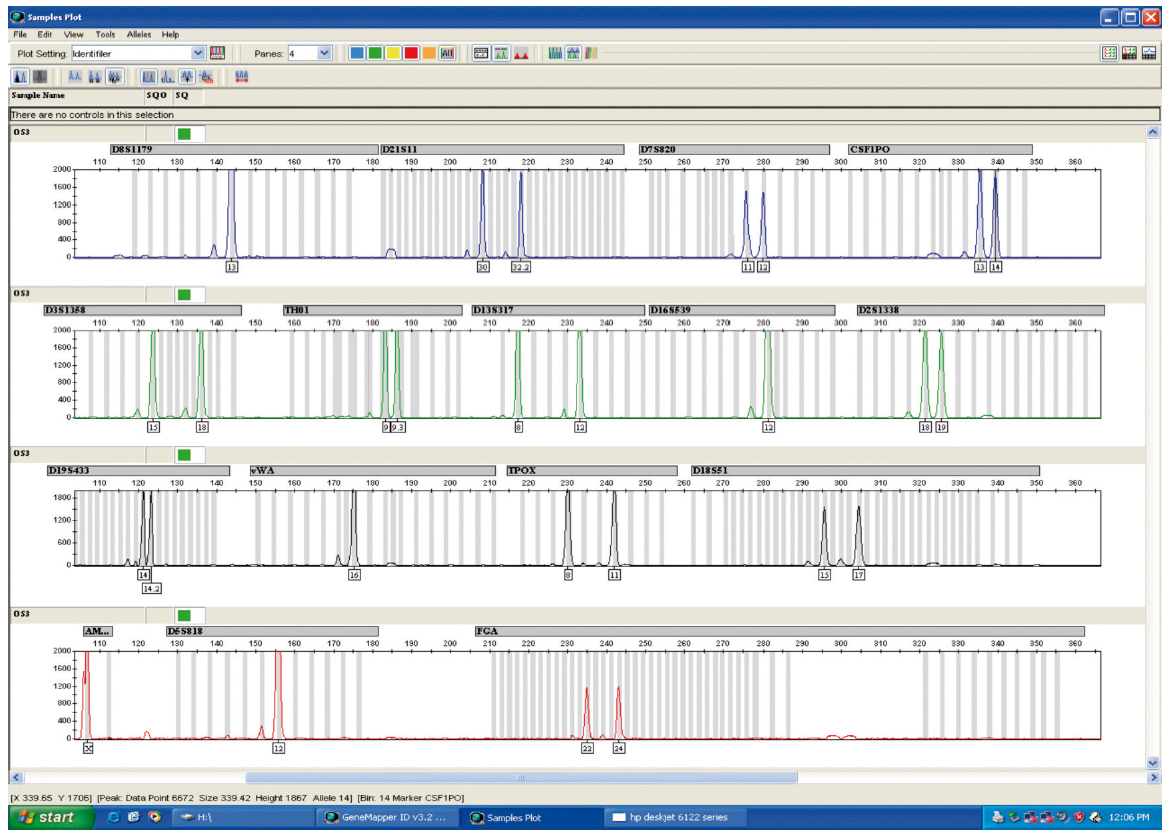
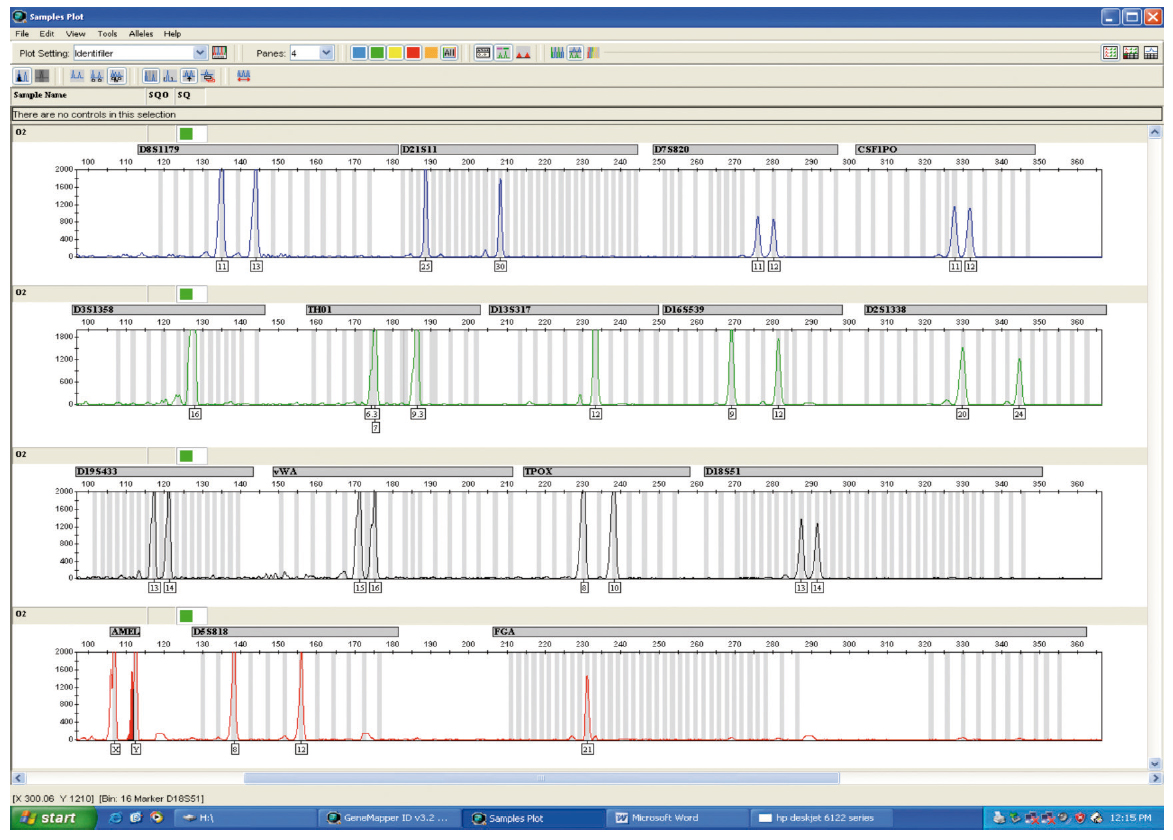
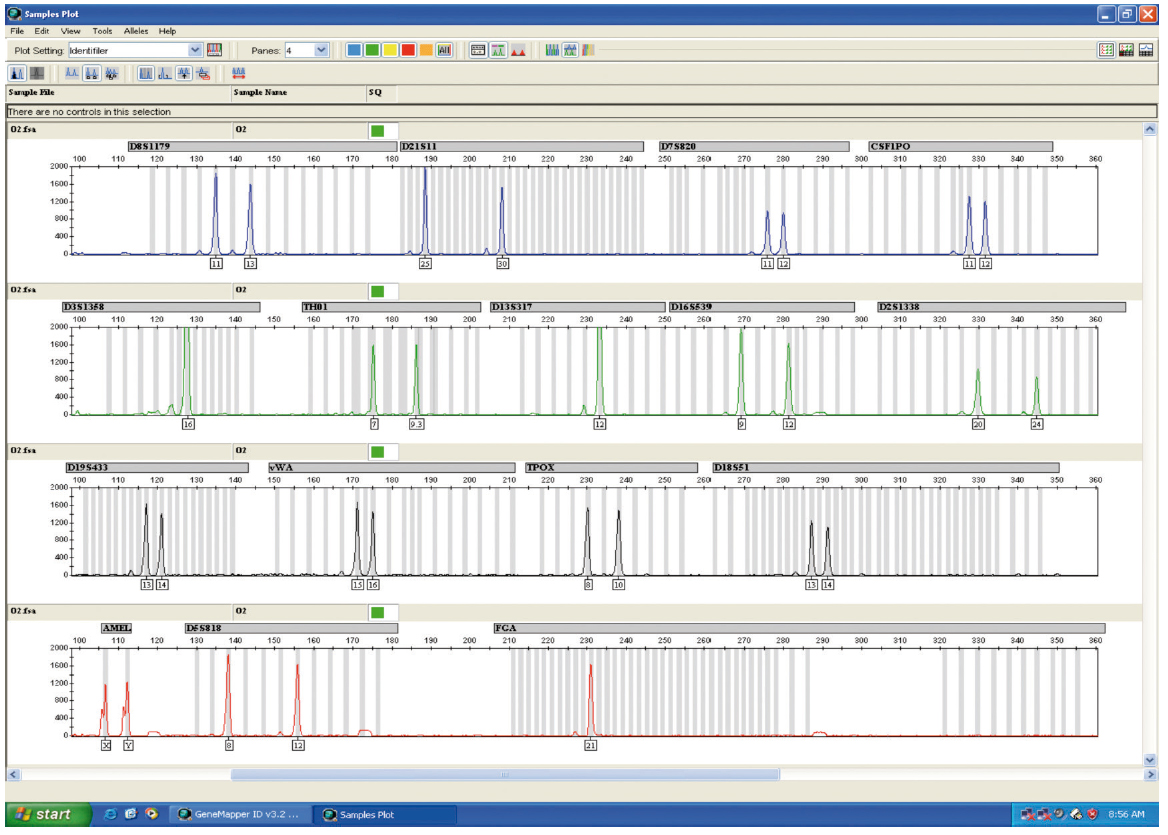
Finalmente se compararon los electroforogramas de las muestras de orina y de las muestras de sangre de los voluntarios. (Figuras 1 y 2; Figuras 3 y 4).
Figura 1. Electroferograma de la muestra de orina del individuo 3.
Figura 2. Electroferograma de la muestra de sangre del voluntario 3.
Figura 3. Electroferograma de la muestra de orina del voluntario 2.
Figura 4. Electroferograma de la muestra de sangre del voluntario 2.
Resultados
Comparando los patrones STR obtenidos a partir de las muestras de orina con los obtenidos a partir de las muestras de sangre, sólo cabía esperar dos posibilidades:
• Exclusión cuando las muestras no fuesen coincidentes.
• No-exclusión cuando los patrones de bandas fuesen coincidentes.
Los primeros resultados, de los cuatro primeros voluntarios, mostraron mezclas en los perfiles de orina. Se achacó a problemas de contaminación, pues salvando las vías de posible contaminación, se obtuvieron perfiles coincidentes 100 % de orina frente a las sangres.
De una muestra de orina no se pudo elaborar el perfil puesto que no se consiguió aislar DNA suficiente por los métodos empleados.
Para la segunda prueba, se recogieron muestras nuevas de cinco voluntarios, tres de ellos coincidentes con los de la primera prueba. Se extrajo DNA por ambos métodos a partir de la misma cantidad de orina (10 mL) y se obtuvieron los mismos resultados que en la primera prueba.
En ambas pruebas, el rendimiento del método manual dio mejores resultados en cuanto a cuantificación de DNA (Tabla 2). Los resultados mostraron un 100 % de coincidencias, pudiendo individualizar las muestras de orina para cada voluntario a partir de la comparación de ambos electroforogramas (sangreorina) (Figuras 1, 2, 3 y 4).
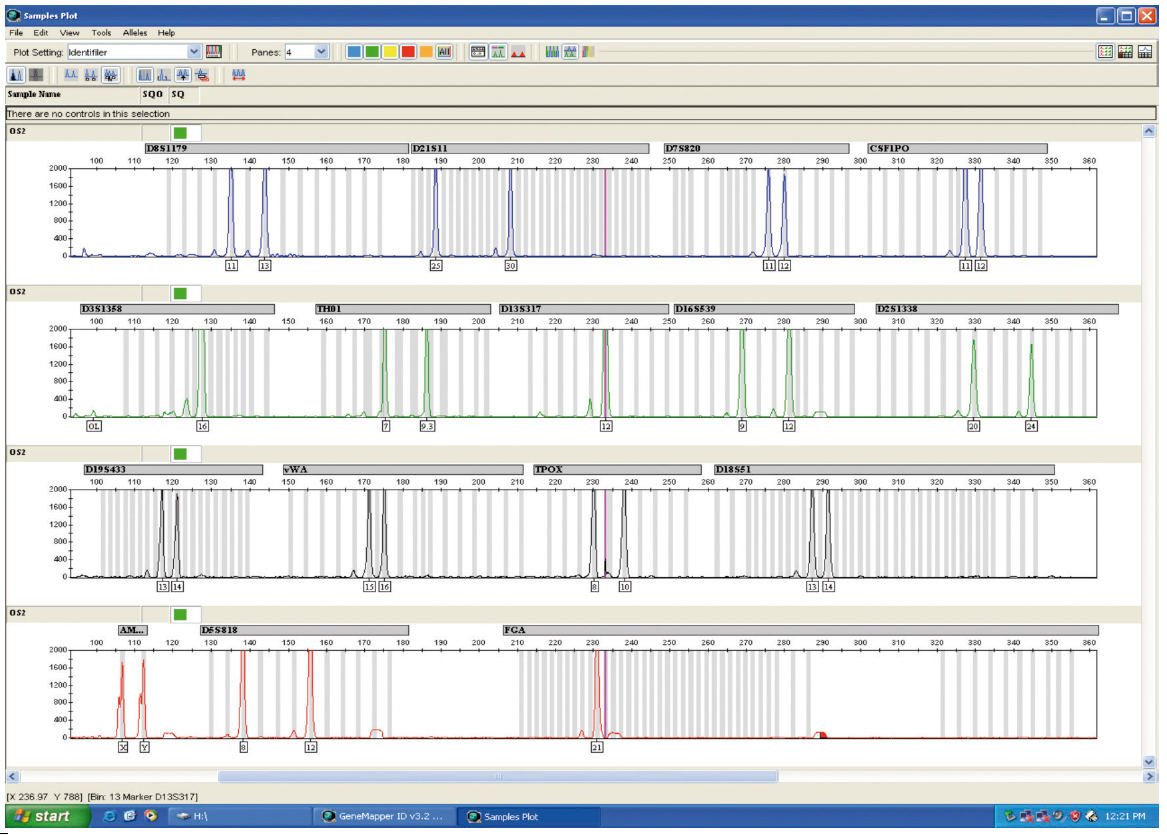
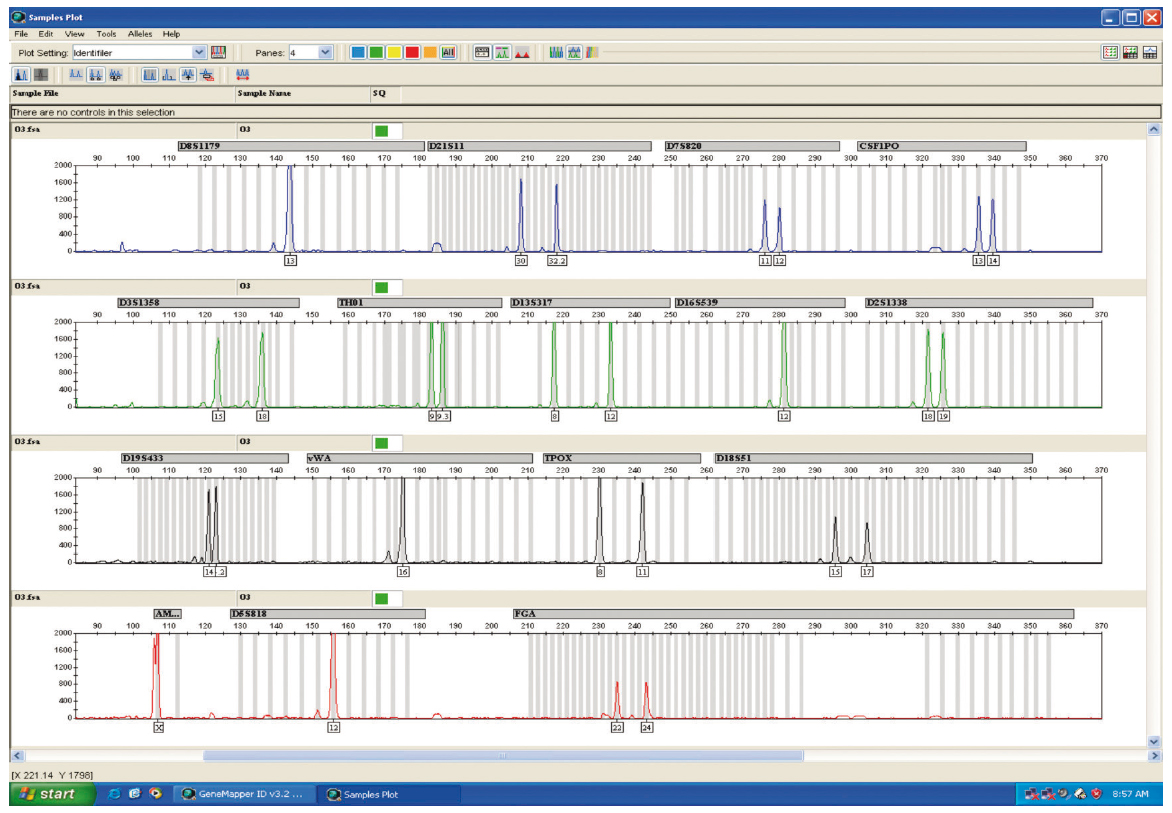
Al cabo de dos meses se realizaron los perfiles del DNA obtenido de las muestras de orina mediante ambos métodos. Se obtuvieron idénticos resultados (Figuras 5 y 6).
Figura 5. Electroferograma del DNA del voluntario 3 tras dos meses de almacenaje a -80oC.
Figura 6. Electroferogframa del DNA del voluntario 2 al cabo de dos meses de su almacenaje a -80oC.
Discusión
Dado que hay métodos de genotipado de STR de DNA que están validados para estudios forenses9,10 y entre ellos el AmpFlSTR® Identifiler® PCR Amplification Kit, podemos usar dicha tecnología en investigaciones de dopaje para establecer la identidad del donante5.
Hay que enfatizar que esto sólo es posible si tanto las muestras de orina como las de sangre son custodiadas y codificadas de forma unívoca. Para ello se han editado guías prácticas que recomiendan entre otras cosas, un lugar de recogida de muestras limpio y seguro con sólo acceso a personal autorizado y en donde la cadena de custodia de la recogida de la muestra implique a la persona recolectora y al donante, finalizando esta custodia con el correcto empaquetamiento en contenedores que minimicen los riesgos de daño para la muestra en el caso de que se envíe a los centros de análisis, o que finaliza en el caso del apropiado almacenaje de la muestra en frío. También ha de contemplarse la privacidad del proceso de micción y el mantenimiento de la integridad de la muestra.
En cuanto a la integridad de la muestra, se deben evitar procesos de dilución o alteración de la misma en el momento de la micción, riesgos de contaminación, el recolector debe comprobar si el volumen, la temperatura, el color y la apariencia de la muestra son los correctos, siempre a la vista del donante y éste deberá a su vez estar informado del test a realizar con su muestra y deberá dar la información únicamente correspondiente a la prueba a realizar con su muestra. Por supuesto, el donante debe estar identificado, con foto por ejemplo, o si la identificación no es posible, se recomienda descartar la muestra.
Nosotros recibimos las muestras de voluntarios a los que les pedimos unas precauciones de recogida mínimas. De una de las muestras de orina no se obtuvo DNA a pesar de ser una voluntaria femenina, cuestión que no se tuvo en cuenta ya que pretendíamos saber si podríamos obtener perfiles genéticos de orina con nuestros medios y capacitaciones.
En la práctica, tanto para Johnson et al1 como para Brinkmann et al5, 10 mL de orina fresca son suficientes para extraer DNA. También se recomienda que sea la primera orina de la mañana. Para nuestro primer experimento se extrajo DNA a partir de 3 mL orina de media mañana. Después repetimos el estudio homogeneizando las muestras a alícuotas de 10 mL de orina y tener una cantidad igual de orina de partida. Con la orina fresca, obtuvimos DNA en todos los casos.
Según el estudio de Cannas et al7, la composición de la orina afecta a la estabilidad del DNA durante su almacenamiento. Dado que la muestra se va degradando con el tiempo se puede sugerir que se extraiga el DNA para posible identificación en el caso de que sea necesario, en el momento de su procesamiento inicial. Es cierto que en estos estudios las muestras de orina se recogen y almacenan a temperatura ambiente. Nosotros almacenamos el DNA extraído a -80oC y lo recuperamos dos meses después para comprobar su estabilidad. Hecho que se probó satisfactoriamente.
Dado que la muestra se va degradando con el tiempo se puede sugerir la extracción del DNA para posible identificación en el caso de que sea necesario. Se comprobó que el DNA se puede guardar a -80oC sin riesgo de posibles contaminaciones y con la calidad necesaria para posibilitar una identificación mediante análisis de STR a posteriori.
Otro punto importante es la elección del marcador que se utilice para individualizar las muestras de orina. Nuestro equipo utilizó el DNA nuclear por tener el aparataje y reactivos apropiados pero por ser también un proceso más sencillo, validado y automatizado.
Para Castella et al4, y desde un punto de vista práctico, el perfil genético que se obtiene a partir del DNA nuclear (nDNA) requiere una reacción de PCR multiplex con un kit comercial mientras que para obtener la secuenciación de los dos loci del DNA mitocondrial (mtDNA) es necesario, al menos, dos reacciones de PCR y dos reacciones de secuenciación.
Los marcadores genéticos que se analizan en mtDNA aparecen en todas las muestras independientemente del tiempo de almacenamiento de las mismas y son bastante constantes4. En el estudio de Junge et al11, la determinación de los STR en DNA nuclear falló a excepción de la amelogenina tras orina almacenada durante 4 meses y su estudio de las regiones mitocondiales HVR I y HVR II aunque exitoso y coincidente tanto en las muestras de orina como en las de sangre, no pudieron discernir si las muestras pertenecían al mismo individuo o a individuos diferentes con el mismo linaje materno.
Actualmente no hemos encontrado estudios que permitan la identificación del donante en muestras de orina sospechosas. Nosotros hemos comprobado que se puede hacer de forma semiautomática usando los perfiles genéticos a partir del sedimento urinario.
Conclusiones
La extracción de DNA realizada en nuestro estudio de las muestras de orina por el sistema del kit de Master Diagnóstica (VITRO, S.A.) necesita de incubación en baño María toda la noche, al igual que la extracción manual y posterior precipitación en fenol-cloroformo-isoamílico, mientras que el sistema automático MagNa Pure de Roche puede hacer la extracción en el mismo día, lo que permitiría amplificar tanto la muestra de orina como la de sangre a la vez.
Sin embargo, el rendimiento de DNA de las muestras de orina fue mejor con el sistema del buffer de lisis y digestión con proteinasa K durante toda la noche que con el sistema automático. Al cuantificar el DNA extraído pudimos ver mayor cantidad por el método manual.
Se ha comprobado que el DNA se puede guardar a -80oC sin riesgo de posibles contaminaciones y con la calidad necesaria para posibilitar una identificación mediante análisis de STR a posteriori. Por otro lado, la utilización de DNA nuclear da mucha información y representa un método semiautomático sencillo y rápido con los kits comerciales de los que se dispone actualmente.
El perfil genético del sedimento urinario de orinas conservadas en congelación y custodiadas, nos permite asociarlas a sus donantes.
Agradecimientos
A todos los voluntarios donantes pertenecientes al Servicio de Anatomía Patológica (incluyendo el laboratorio de Identificación Sanitaria y el laboratorio de Genética) sin cuya colaboración desinteresada no se hubiera llevado a cabo este estudio.
Bibliografía
1. Johnson, D.J., Calderaro, A.C., Roberts, K. A. Variation in nuclear DNA concentrations during urination. J Forensic Sci. January 2007. Vol.52 (1):110-113. [ Links ]
2. Malson, S. Flanagan, N., McAlister, C. and Dixon, L. The prevalence of mixed DNA profiles in fingernail samples taken from couples who co-habit using autosomal and Y-STRs. For. Sci. Inter. Genert. (2009) 3: 57-62. [ Links ]
3. Hausknecht, R., Gula, R., Pirga, B., Kuehn, R. Urine - a source for noninvasive monitoring in wildlife. Mol Eco Notes. March 2007. Vol.7, 2: 208-212. [ Links ]
4. Castella, V., Dimo-Simonin, N., Brandt-Casadevall, N., Robinson, N., Saugy, M., Tqaroni, F., Mangin, P. Forensic identification of urine simples: a comparison between nuclear and mitochondrial DNA markers. Int J Leg Med (2006) 120: 67-72. [ Links ]
5. Brinkmann, B., Rand, S. and Bajanowsky, T. Forensic identification of urine samples. Int J Leg Med (1992) 105:59-61. [ Links ]
6. Thevis, M. et al. Detection of manipulation in doping control urine sample collection: a multidisciplinary approach to determine identical urine samples. Anal Bioanal Chem, Aug 2008; 388(7):1539-43. [ Links ]
7. Cannas, A., Kalunga, G., Green, C., Calvo, L., Katemangwe, P., Reither, K. et al for the TB trDNA consortium. Implications of storing urinary DNA from different populations for molecular analyses. Sep 2009. PLoS ONE. 4 (9): e6985. doi:10.1371/journal.pone.0006985 [ Links ]
8. José Luque y Ángel Herráez. Texto Ilustrado de Biología Molecular e Ingeniería Genética. Conceptos, Técnicas y Aplicaciones en Ciencias de la Salud. Ediciones Harcourt, S.A.. 2001. ISBN:84-8174-505-7. [ Links ]
9. Caragine, T. et al. Validation of testing and interpretation protocols for low template DNA samples using AmpFlSTR® Identifiler®. For. Sci. Croat Med J, 2009; 50: 250-67. [ Links ]
10. Collins, P.J. et al. Developmental validation of a single-tube amplification of the 13 CODIS STR loci, D2S13338, D19S433 and Amelogenin: The AmpFlSTR® Identifiler® PCR Amplification Kit. J Forensic Sci, Nov. 2004, Vol.49. No.6: 1-13. [ Links ]
11. Junge, A.,Steevens, M., Madea, B. Successful DNA typing of a urine sample in a doping control case using human mitochondrial DNA analysis. J Forensic Sci. 2002 Sep; 47 (5): 1022-4. [ Links ]
![]() Dirección para correspondencia:
Dirección para correspondencia:
Ma Luisa Marqués Negredo.
Servicio de Anatomía Patológica.
Unidad de Identificación Sanitaria.
Hospital Central de la Defensa "Gómez Ulla" de Madrid.
Glorieta del Ejército, s/n.
28047 Madrid.
Tno.: 91 422 25 47.
Fax: 91 422 25 44.
E-mail: mmarneg@oc.mde.es.
Recibido: 4 de abril de 2011
Aceptado: 2 de noviembre de 2011