INTRODUCCIÓN
Numerosos estudios de asociación de genoma completo (GWAS) y estudios de asociación de genes candidatos han encontrado algunos polimorfismos bialélicos (single nucleotide polymorphisms, SNPs) en el gen SOST, que codifica la esclerostina, asociados con la densidad mineral ósea (DMO) y la predisposición a fracturas1 2 3-4. Sin embargo, se desconoce el mecanismo responsable de esa asociación. Entre los mecanismos generales por los que las variantes genéticas predisponen a las enfermedades complejas se encuentran los mecanismos epigenéticos, como la metilación del ADN, que modulan la transcripción de genes de forma directa (localmente) o indirecta (remotamente)5. En este sentido, cabe destacar que la metilación del promotor de SOST está inversamente relacionada con la actividad transcripcional, es decir, con los niveles de expresión, de este gen6.
La metilación de ADN es una marca epigenética, que consiste en la adición de un grupo metilo en la posición 5' del anillo de citosina, generalmente en citosinas que preceden a la guanina, formando los denominados sitios CpG. Los sitios CpG se distribuyen a lo largo de todo el genoma y son abundantes en algunas regiones específicas, como los promotores, denominadas islas CpG. Los niveles de metilación de los sitios y/o islas CpG tienen perfiles específicos de acuerdo con el tejido de origen y modulan la expresión de genes en muchos tejidos, incluido el hueso7 8 9-10.
El ADN libre circulante (cell freeDNA, cfDNA) está presente en fluidos, como la orina, el líquido sinovial, el plasma o el suero, y es un biomarcador molecular interesante porque es fácil de obtener sin utilizar procedimientos invasivos11. Por ejemplo, es un biomarcador muy estudiado en tumores, ya que la cantidad de cfDNA aumenta con la presencia de estos. Además, los tumores acumulan mutaciones específicas, que permiten diferenciarlos de otras cadenas de ADN con distinto origen12,13. El cfDNA es, por tanto, un marcador prometedor para la detección, el diagnóstico, el pronóstico, el seguimiento y el tratamiento futuros de diversas enfermedades14.
Previamente, hemos demostrado ciertos patrones específicos de metilación del ADN de osteoblastos y de células madre mesenquimales (hMSCs) en la osteoporosis. Estas regiones diferencialmente metiladas están enriquecidas en genes asociados a la diferenciación celular, en el caso de las hMSCs15, y en la formación del esqueleto en el caso de los osteoblastos16. Específicamente, hemos comprobado previamente que el grado de metilación del promotor de SOST regula la expresión génica en osteoblastos. En esa línea, la desmetilación induce la expresión de SOST, incluso en células en las que normalmente este gen está reprimido17,18.
El objetivo de este estudio fue determinar la metilación del ADN en el promotor de SOST en ADN de hueso y en ADN libre en suero y la posible relación con algunos polimorfismos previamente asociados con la DMO. Además, estudiar los efectos de dichos polimorfismos sobre la expresión de la esclerostina.
MATERIAL Y MÉTODOS
Selección de pacientes
Se obtuvieron las cabezas femorales de 33 pacientes sometidos a cirugía de reemplazo de cadera por fractura osteoporótica (FRX; n=15) o artrosis (OA; n=18). Se excluyeron los pacientes con osteoporosis secundaria, artrosis secundaria o fracturas debidas a traumatismos de alta energía, con edades comprendidas entre 61 y 91 años. De cada paciente, se obtuvieron muestras de tejido óseo, sangre y suero. El suero se utilizó para aislar ADN libre y estudiar la metilación del promotor de esclerostina. La sangre se utilizó para la obtención de ADN genómico con el objetivo de analizar los polimorfismos de interés.
El estudio fue aprobado por el Comité de Ética en Investigación Clínica de Cantabria y los pacientes dieron su consentimiento informado por escrito.
Aislamiento de ADN
Las muestras de hueso trabecular de la parte central de las cabezas femorales se obtuvieron con un trócar. Se congelaron instantáneamente con nitrógeno líquido y se homogeneizaron con un politrón en tampón de lisis y proteinasa K. Después de una incubación toda la noche a 55º, se extrajo el ADN con fenol:cloroformo:alcohol isoamílico, según se ha publicado previamente16. El ADN libre en suero se obtuvo de dos alícuotas de 1 ml de suero, procedimiento doble realizado en paralelo (2 ml de suero por paciente para el análisis). A cada alícuota, en un tubo falcon de 15 ml, agregamos 500 µl de tampón de lisis (Tris-HCl, EDTA, acetato de sodio y SDS) y 5 µl de proteinasa K (20 mg/ml). La mezcla se incubó durante 1 hora en un baño de agua a 56ºC. El aislamiento del ADN se realizó, al igual que con el hueso, por el procedimiento con fenol:cloroformo:alcohol isoamílico. El pellet(no visible) se dejó secar a temperatura ambiente y se resuspendió con 20 µl de agua destilada. El ADN de las células sanguíneas se extrajo con el kit Illustra blood genomic Prep Mini Spin (GE Healthcare Life Sciences, Marlborough, EE.UU.).
Genotipado
En el ADN de sangre total, analizamos un total de 4 SNPs previamente asociados con SOST y la densidad mineral ósea: rs851054, rs851056, rs1234612 y rs10534024. El ADN se cuantificó por el procedimiento de Qubit (Thermofisher Scientific, Waltham, EE.UU.) y se realizó una PCR mediante ensayos con sondas Taqman, para el análisis de los polimorfismos previamente mencionados.
Análisis de metilación de ADN
Se utilizaron 500 ng de ADN de hueso por muestra, para modificar con bisulfito con el kit de metilación EZ DNA Methylation-Gold (ZymoResearch, Irvine, EE.UU.), siguiendo las instrucciones del fabricante. Por otro lado, con el ADN aislado de suero, se utilizó todo el volumen de la suspensión (20 µl del ADN resuspendido) y se sometió igualmente a modificación con bisulfito con el kit de metilación EZ DNA Methylation-Gold. El nivel de metilación de la CpG seleccionada para el estudio, en la región del promotor SOST, se analizó mediante pirosecuenciación (PyromarkQ24 Advanced System®). Los cebadores utilizados para la amplificación por PCR y la secuenciación se diseñaron con el diseñador de ensayos PyroMark (Qiagen N.V., Hilden, Alemania) (Cebador sentido. 5'-TGGTGGGGTGATAAATGAATT-3'; Cebador antisentido. 5'-TGGTGGGGTGATAAATGAATT-3'; Cebador de secuenciación. 5'-ATTTGGTTTGAGAAATGG-3'). La PCR se realizó con un cebador biotinilado, que permite su purificación en una plantilla de ADN de una sola hebra, utilizando la estación de trabajo de vacío PyromarkQ24 (Qiagen N.V., Hilden, Alemania) (según las instrucciones del fabricante). Finalmente, las reacciones de pirosecuenciación y la cuantificación de la metilación se realizaron en PyroMark Q24 Advance System (Qiagen N.V., Hilden, Alemania).
Se analizaron los niveles de metilación de la CpG en la región seleccionada, cercana a los polimorfismos examinados, situada aproximadamente a 300 pares de bases antes del sitio de inicio de la transcripción (Figura 1A).
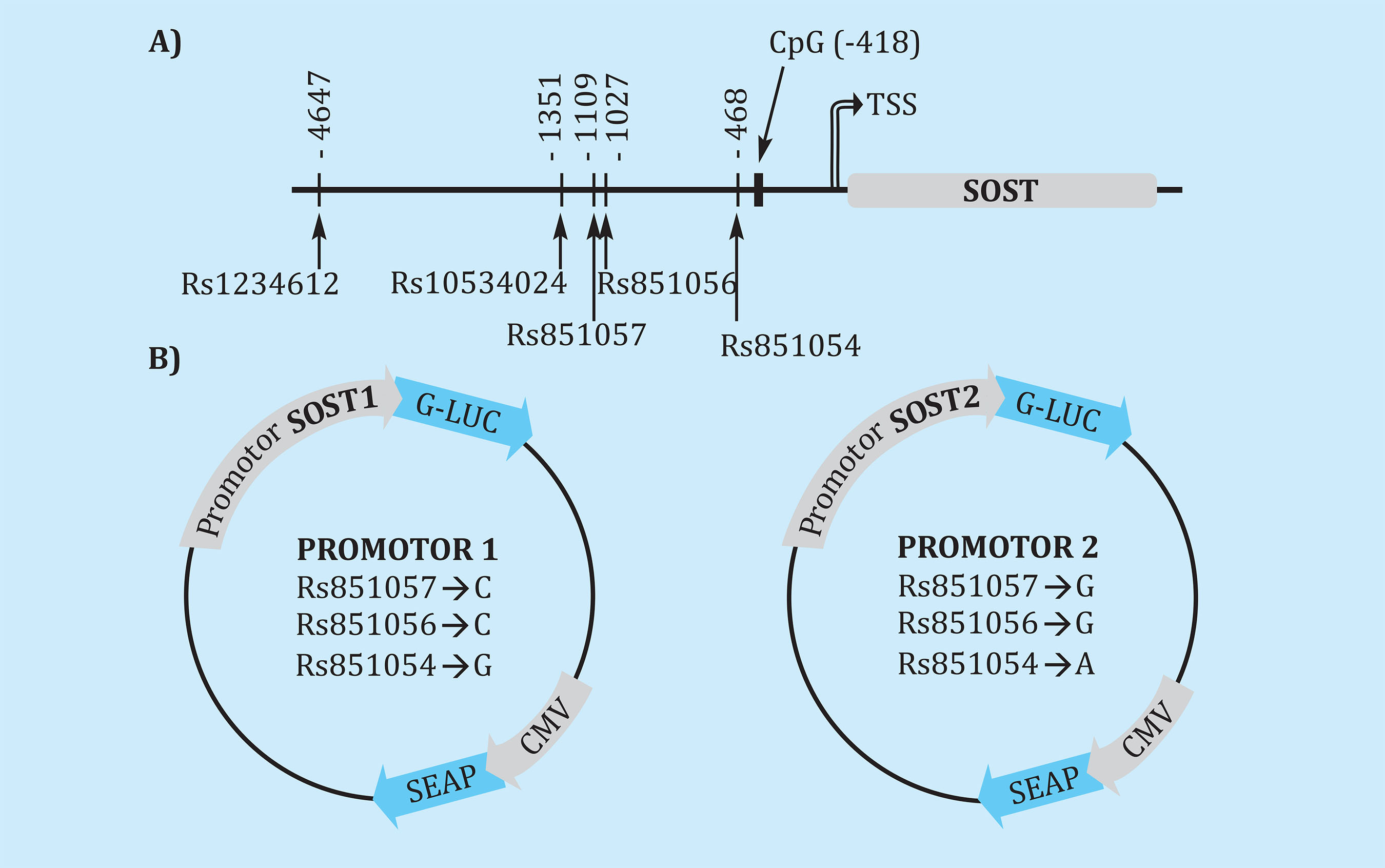
Figura 1. (A) Esquema de la región promotora del gen SOST y localización de los polimorfismos analizados, señalando la distancia al punto de inicio de la transcripción (TSS). El dinucleótido estudiado para los niveles de metilación de la región promotora está expuesto como 'CpG', con la distancia al TSS. (B) Vectores reporteros con el gen de la luciferasa (G-LUC) hacia la región 5' del promotor de esclerostina y la actividad fosfatasa alcalina (SEAP) bajo el promotor constitutivo del citomegalovirus (CMV). Se encuentran dos vectores, cada uno con un haplotipo distinto con respecto a los polimorfismos frecuentes de la región (rs851054, rs851056 y rs851057)
Expresión de SOST y niveles de esclerostina
Los niveles séricos de esclerostina se analizaron mediante ELISA (Teco Medical Group, Sissach, Suiza). La sensibilidad de este kit es de 0,05-3 ng/ml.
La expresión de SOST en hueso se analizó por PCR cuantitativa a tiempo real (RT-qPCR). Para ello, se extrajo ARN de las biopsias de hueso congeladas homogeneizando con trizol, aislando con cloroformo y precipitando el ARN con isopropanol. El ADN complementario (ADNc) se sintetizó con el kit TaKaRa PrimeScript RT (TaKaRa, Shiga, Japón). Utilizamos 1 µg de partida de ARN, hexámeros aleatorios y oligo-dT como cebadores, con las cantidades recomendadas por el fabricante. La abundancia de la transcripción de los ARN mensajeros se evaluó mediante RT-qPCR utilizando ensayos Taqman disponibles comercialmente (Thermofisher Scientific) en un sistema de PCR en tiempo real Applied Biosystems 7300. Utilizamos como genes de referencia, GAPDH y TBP.
Vectores reporteros y análisis de la actividad transcripcional
Se adquirió el vector reportero del promotor de SOST (HPRM50859-PG04; GeneCopoeia, Rockville, EE.UU.). Además, se obtuvo un segundo vector con la misma secuencia, pero variando el haplotipo (rs851054 G/A; rs851056 C/G; rs851057 C/G). Ambos vectores tienen el gen de la luciferasa bajo la secuencia promotora de SOST, y el gen de fosfatasa alcalina bioluminiscente bajo un promotor constitutivo (Figura 1B). Este vector dual permite normalizar la señal y cuantificar la señal generada por cada promotor transfectado. Asimismo, obtuvimos un vector con un promotor vacío (pEZX-PG04; GeneCopoeia, Rockville, EE.UU.) sobre la secuencia de la luciferasa, como control negativo para la transfección.
La transfección de los distintos vectores se desarrolló en la línea celular HEK-293T, con lipofectamina 3000 (Thermofisher Scientific, Waltham, EE.UU.). Para la transfección se sembraron 50.000 células (HEK-293T) por pocillo en una placa de 24 pocillos, por triplicado. Al día siguiente, con una confluencia aproximada de un 80%, se transfectaron 500 ng totales de cada uno de los vectores, en pocillos independientes, utilizando lipofectamina 3000, según las recomendaciones del fabricante. Se analizó la señal de luciferasa y fosfatasa alcalina a las 24 h, 48 h y 72 h. Para esto, se utilizó el kit Secrete-Pair Dual Luminescence Assay Kit (GeneCopoeia, Rockville, EE.UU.) y se midió por luminometría.
Análisis de los resultados
La presencia de desequilibrio de ligamiento y la distribución haplotípica se analizó con el programa Haploview19.
Los análisis estadísticos realizados en este estudio se llevaron a cabo utilizando la versión 3.6.0 del software R. Se compararon los alelos con respecto a su nivel de metilación del promotor de SOST y/o expresión de SOST en hueso mediante análisis de varianza (ANOVA). La comparación entre grupos de pacientes (FRX vs.OA) se realizó mediante T de Student. En todos los casos se tomaron como significativos los valores p menores que 0,05.
RESULTADOS
Todas las muestras de suero se analizaron por duplicado y los niveles de metilación por pirosecuenciación se tomaron como fiables cuando la intensidad de señal era íntegra. La variabilidad entre los duplicados de suero fue pequeña, con un error estándar promedio de ±3,89%.
El análisis de metilación de ADN libre en suero no reveló diferencias estadísticamente significativas en relación con los diversos alelos de los polimorfismos analizados (rs851054, rs851056, rs1234612 y rs10534024) (Figura 2). Tampoco se encontraron diferencias en la metilación de ADN de hueso en asociación con los citados polimorfismos (Figura 3).

Figura 2. Porcentaje de metilación de ADN libre en suero de la región promotora del gen SOST, en los distintos individuos (n=33) genotipados para cada uno de los 4 polimorfismos (rs851054, rs851056, rs10534024 y rs1234612). El valor de p señala el análisis de la varianza entre los distintos alelos y el valor de metilación

Figura 3. Porcentaje de metilación de ADN de hueso de la región promotora del gen SOST, en los distintos individuos (n=33) genotipados para cada uno de los 4 polimorfismos (rs851054, rs851056, rs10534024 y rs1234612). El valor de p señala el análisis de la varianza entre los distintos alelos y el valor de metilación
Además, con las biopsias de hueso se obtuvo también el ARN con el objetivo de estudiar la expresión endógena de SOST en hueso. Los resultados obtenidos por PCR cuantitativa a tiempo real no revelaron diferencias estadísticamente significativas de la expresión endógena de SOST en hueso, en relación con los polimorfismos analizados (Figura 4).

Figura 4. Expresión del gen SOST en hueso, de los individuos (n=33) genotipados para cada uno de los 4 polimorfismos (rs851054, rs851056, rs10534024 y rs1234612). Los niveles de expresión se calcularon por RT-qPCR, normalizados por los genes de referencia (GAPDH y TBP) y están expresados como deltaCt. El valor de p señala el análisis de la varianza entre los distintos alelos y los niveles de expresión
Es de señalar que 3 de los 4 SNPs se encontraban en fuerte desequilibrio de ligamiento, con D' de 1 y estrecha correlación entre sus alelos (r2de 0,83-1), El otro polimorfismo, rs1234612, no formaba sin embargo parte de ese bloque (Figura 5). El análisis por haplotipos o genotipos combinados tampoco reveló asociaciones estadísticamente significativas con la metilación o la expresión génica (datos no mostrados).
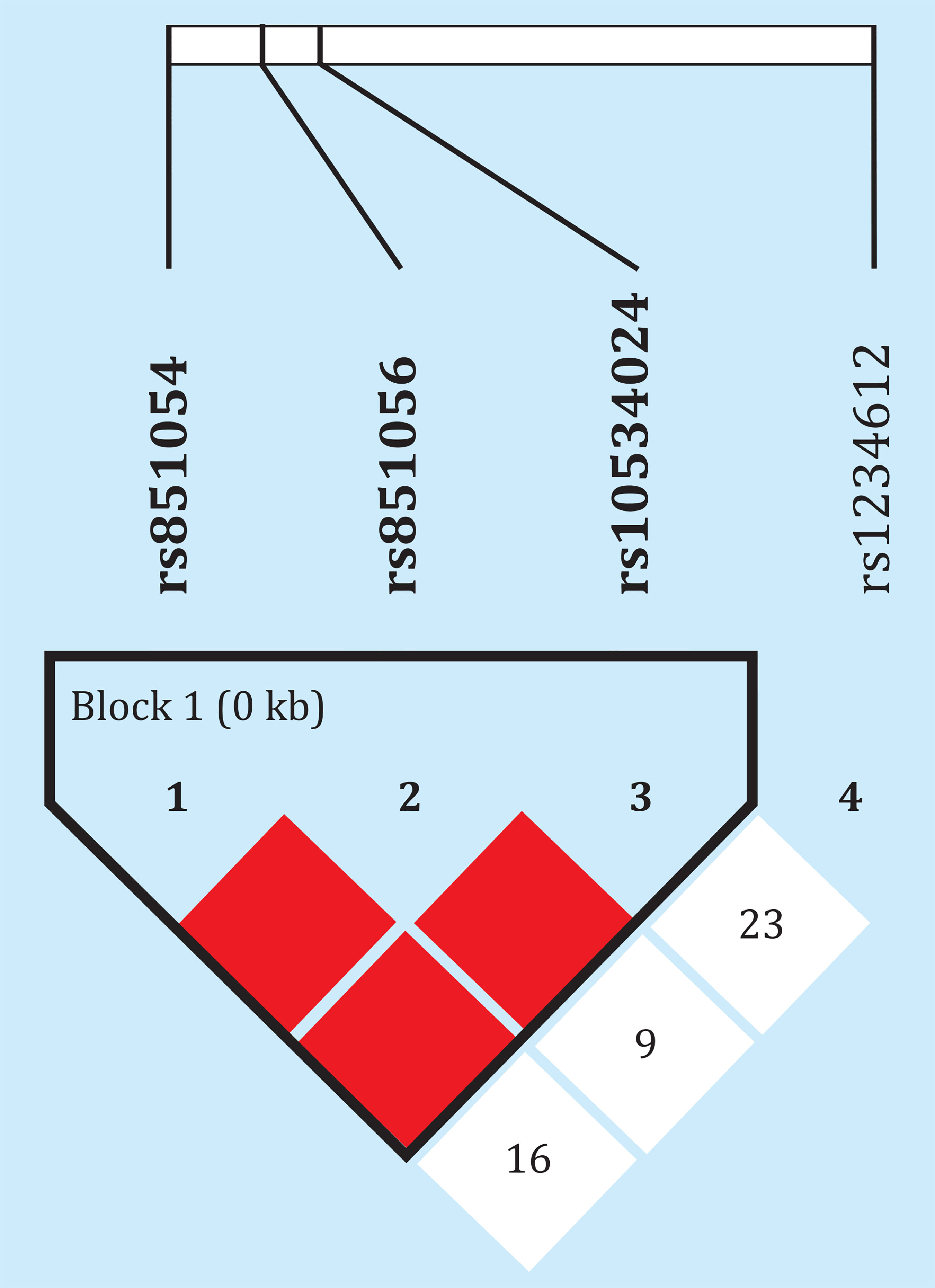
Figura 5. Desequilibrio de ligamiento entre los polimorfismos analizados. Los númerosrepresentan los valores de distancia D', que pueden variar entre 0 y 1. En la figura se representan los valores multiplicados por 100. En el caso de los cuadros de color el valor es de 1
Las transfecciones con los vectores reporteros, que portan la región promotora del gen de la SOST, mostraron una elevada actividad transcripcional, independientemente del vector utilizado. De hecho, aumentó hasta 20 veces a las 24 horas con respecto al vector vacío. Sin embargo, ambas construcciones, con alelos opuestos, mostraron una actividad similar (Figura 6)

Figura 6. Medición de la actividad del promotor de SOST con transfecciones de los distintos reporteros (promotor 1 y promotor 2). Cada uno de los reporteros tiene el haplotipo contrario para los polimorfismos (rs851054, rs851056 y rs851057). La señal relativa se ha calculado normalizando cada valor de absorbancia de luciferasa por el cociente de la actividad fosfatasa alcalina (SEAP). Pösteriormente se ha comparado con el cociente de las transfecciones realizadas con un vector vacío (sin el promotor, pero con actividad SEAP). Las barras de error muestran el error estándar
DISCUSIÓN
La esclerostina es un potente inhibidor de la vía Wnt, a través del bloqueo de los co-receptores Lrp4, 5 y 6, e impidiendo así la señalización de esta vía. Esta molécula tiene un marcado papel en la biología ósea. Los ratones con la delección de SOST tienen mayor formación de hueso y masa ósea20. Por el contrario, la sobreexpresión de SOST en osteoblastos disminuye la masa ósea21. Además, ciertas mutaciones del gen SOST que causan una pérdida de la esclerostina en humanos, se asocian con una alta actividad de formación ósea y elevada DMO, causando la enfermedad de Van Buchem o la esclerosteosis22,23. A la inversa, un anticuerpo monoclonal que bloquea la acción de la esclerostina (romosozumab) ha sido recientemente aprobado por la FDA (U.S. Food and Drug Administration)para el tratamiento de la osteoporosis, tras observar que mejoraba la masa ósea en estudios en animales y en huma nos24,25
Varios estudios sugieren que algunas variantes alélicas del gen SOST pueden influir en la DMO y el riesgo de osteoporosis26,27. Puesto que se trata de variantes no codificantes, presumiblemente su efecto ha de basarse en influir en la expresión de dicho gen. Por otro lado, previamente hemos podido demostrar la importancia de la metilación de ADN en la regulación de la expresión de esclerostina en el linaje osteoblástico18. Asimismo, en diversos estudios se ha comprobado que las variantes genéticas pueden influir sobre la metilación de ADN y con esto sobre la expresión del gen afectado5. De ahí que el objetivo de este estudio fue explorar la repercusión funcional de algunos polimorfismos frecuentes en la región promotora del gen SOST sobre la metilación y la expresión génica. Sin embargo, a pesar de su asociación con la DMO15, no hemos encontrado ninguna asociación significativa entre la frecuencia alélica y los niveles de metilación de ADN, o entre la frecuencia alélica y los niveles de expresión génica, ni analizando los polimorfismos aisladamente, ni al combinarlos según los genotipos o los haplotipos. Concordantemente con ello, los experimentos de transfección con vectores reporteros no han revelado diferencias entre las variantes alélicas de la región promotora y la actividad transcripcional. Por tanto, nuestro estudio no apoya que exista una influencia de estos polimorfismos sobre la expresión del gen de la esclerostina, ni directa, ni mediada a través de cambios en la metilación del promotor.
Hay diversas limitaciones que pueden condicionar estos resultados negativos. En primer lugar, el estudio de asociación entre las variables alélicas y la metilación de ADN está acotada a una región concreta del promotor. Para mejorar esto, habría que hacer un barrido mucho más amplio de distintas marcas de metilación de ADN. En segundo lugar, el efecto de los polimorfismos estudiados puede depender de otros polimorfismos frecuentes en desequilibrio de ligamiento. Además, estos posibles polimorfismos pueden estar en regiones alejadas del promotor, como regiones reguladoras (enhancer)o, incluso, interacciones con otros cromosomas. Este último hecho condiciona también el análisis con vectores reporteros, donde solo se encuentra la región promotora de SOST. Los vectores reporteros se transfectan en experimentos in vitro, donde la metilación de ADN se pierde y, por lo tanto, no se refleja la situación in vivoapropiadamente. Otra limitación de este estudio es la presencia de muestras con distintas patologías (osteoporosis y artrosis), ya que estas patologías pueden estar influyendo distintamente sobre los niveles de metilación. No obstante, los análisis por separado no han mostrado diferencias en los resultados. Finalmente, el tamaño muestral condiciona la capacidad para demostrar diferencias sutiles entre los polimorfismos, especialmente en los análisis de polimorfismos combinados. En todo caso, es importante remarcar que estos resultados no cuestionan la importancia de la esclerostina en la regulación de la actividad de las células óseas, que ha sido demostrada en numerosos estudios experimentales y clínicos.
Como conclusión, no hemos visto una asociación clara entre los distintos alelos y la metilación de ADN de la región promotora del gen de SOST. Por tanto, no parece que la asociación de estos polimorfismos con la DMO se debe a influencias directas sobre la actividad del promotor, ni a cambios en la metilación de este. Cabe presuponer, pues, que esté mediada por interacciones complejas con regiones distantes de la cromatina. Por otro lado, este estudio plantea la posibilidad de utilizar el ADN libre en suero como biomarcador en algunos trastornos esqueléticos.











 texto en
texto en 


