My SciELO
Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Cited by Google
Cited by Google -
 Similars in
SciELO
Similars in
SciELO -
 Similars in Google
Similars in Google
Share
Revista Española de Enfermedades Digestivas
Print version ISSN 1130-0108
Rev. esp. enferm. dig. vol.108 n.7 Madrid Jul. 2016
https://dx.doi.org/10.17235/reed.2015.3894/2015
REVISIÓN
Causas del fracaso del tratamiento de la hepatitis C en la era de la terapia antiviral directa
Causes of treatment failure for hepatitis C in the era of direct-acting antiviral therapy
Joaquín Cabezas, Susana Llerena, Ángela Puente, Emilio Fábrega y Javier Crespo
Servicio de Aparato Digestivo. Hospital Universitario Marqués de Valdecilla. IDIVAL. Facultad de Medicina. Universidad de Cantabria. Santander
Dirección para correspondencia
RESUMEN
El tratamiento de la hepatitis C en la era de los nuevos agentes antivirales de acción directa ha cambiado radicalmente nuestros esquemas de tratamiento, consiguiendo tasas de respuesta virológica sostenida muy elevadas. Sin embargo, en un subgrupo de pacientes el tratamiento con agentes antivirales directos fracasa. Este colectivo de pacientes a los que podemos denominar como difíciles de curar constituyen el motivo de este artículo, que revisa las causas virológicas del fracaso virológico, sus implicaciones clínicas y algunas recomendaciones finales.
Palabras clave: Hepatitis C. Agentes antivirales de acción directa. Fracaso de la terapia.
ABSTRACT
Hepatitis C therapy in the era of the newer direct-acting antiviral agents has radically changed our treatment schemes by achieving very high rates of sustained virological response. However, treatment with direct antiviral agents fails in a subgroup of patients. This group of so-called difficult-to-treat individuals is the subject of this paper, which reviews the causes of virological failure, their clinical implications, and some final recommendations.
Key words: Hepatitis C. Direct-acting antiviral therapy. Resistance-associated variants. Treatment failure.
Introducción
El descubrimiento del virus de la hepatitis C (VHC) se produjo en 1989 (1). Dos décadas después, el profundo conocimiento del ciclo vital de este virus ha conducido a una impresionante innovación terapéutica.
En este momento, la terapia antiviral es capaz de curar la infección en la mayoría de los pacientes, con tratamientos cortos, libres de interferón (IFN) y con mínimos efectos secundarios. Y, aunque hay múltiples aspectos de la terapia en investigación, es probable que consigamos disminuir de manera significativa la morbimortalidad tanto de causa hepática como extrahepática, particularmente cardiovascular y renal. Esta revolución, ha venido acompañada de algunos cambios de paradigma en relación con esta infección. En primer lugar, su propio descubrimiento, que utilizó una estrategia desconocida hasta entonces para la búsqueda de una infección viral sospechada desde hacía años pero que permanecía oculta a los ojos de los investigadores. En segundo término, y con el objetivo de acelerar el desarrollo de los nuevos agentes antivirales directos (AAD), los organismos reguladores más importantes, la FDA y la EMA, aprobaron ensayos clínicos muy rápidos, evitando la rama de terapia comparadora estándar. En tercer lugar, en muchas regiones del mundo, y particularmente en España, se ha producido una intensa movilización social, sólo comparable a la acontecida en los primeros años de la pandemia por el VIH. Las sociedades científicas han tenido que cambiar la estrategia de sus conferencias de consenso, dado que la rapidez en la innovación farmacológica hace que nazcan anticuadas. Incluso desde el punto de vista económico, tanto la industria farmacéutica como las autoridades han hecho un notable esfuerzo, sustituyendo la habitual compra de pequeños volúmenes y a un precio elevado por una táctica inversa. Y, finalmente, se han elaborado planes estratégicos que abordan de una forma integral el control de la infección a corto y a largo plazo (2).
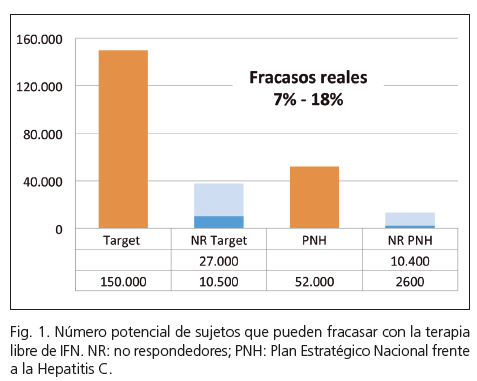
Todos los hechos relatados conducen al optimismo. Cuando observamos los resultados de las nuevas terapias libres de IFN, nuestra mirada se detiene de forma irresistible en su enorme eficacia. De hecho, esta es superior al 90% en la práctica totalidad de los ensayos clínicos independientemente de la carga viral, del haplotipo de la IL28B, del genotipo o subgenotipo, de la existencia de cirrosis, de la ausencia de respuesta a un régimen terapéutico previo que contenga IFN, de la coinfección por el VIH o de la existencia de una insuficiencia renal avanzada. Incluso, cuando analizamos las tasas de respuesta viral sostenida (RVS) en la práctica real, los resultados no difieren mucho de los obtenidos en ensayos clínicos, con tasas de efectividad que oscilan entre el 82 y el 93% (3). Pero si nos fijamos en el negativo de la fotografía, los pacientes que fracasan a esta terapia no son, en modo alguno, una población residual. Se estima que en Estados Unidos cerca de 150.000 personas se han tratado con la combinación sofosbuvir y simprevir. Aplicando una sencilla regla de tres, si de estos 150.000 pacientes no se consigue curar entre el 7 y el 18%, tendremos entre 10.500 y 27.000 enfermos en los que la terapia ha fracasado. Si trasladamos las cifras a nuestro país, el resultado tampoco es tranquilizador. Gracias al Plan Estratégico Nacional Frente a la Hepatitis C (2) estamos en disposición de tratar a más de 50.000 pacientes en un tiempo breve, probablemente inferior a los 3 años previstos. Y operando con la misma regla de tres y asumiendo que los resultados en la practica real en España serán similares a los de EE. UU., el número de pacientes con fallo a la terapia antiviral libre de IFN afectará a entre 2.600 y 10.400 pacientes (Fig. 1).
Los términos "difícil de curar" y "difícil de tratar" se utilizan indistintamente en la literatura, lo que representa una notable dificultad a la hora de establecer el motivo por el que en determinados pacientes no se consigue la eliminación de la infección (4-7). En esta revisión, nos referiremos como pacientes "difíciles de curar" a aquellos con un fracaso relacionado con las características virológicas de la infección, las cuales analizaremos en profundidad, y como "pacientes difíciles de tratar", a aquellos en los que el fracaso se debe a uno de estos tres motivos: a) una mala adherencia terapéutica; b) la suspensión precoz del tratamiento, bien por un improbable efecto secundario o por una no tan improbable interacción clínicamente relevante; y c) la pérdida de seguimiento, en íntima relación con la mala adherencia y que impide valorar la consecución del objetivo de RVS (Tabla I).
Cómo debemos realizar el diagnóstico diferencial del fracaso con terapias libres de IFN
Habitualmente categorizamos el fracaso terapéutico en función del momento en que se produce este: recaída intratratamiento (rebote virológico o breakthrough), recaída postratamiento (recidiva de la infección) y ausencia primaria de respuesta. Pero esta clasificación, que ha sido muy útil durante muchos años, carece de verdadero valor en este momento en el que el fracaso terapéutico con AAD suele aparecer postratamiento, siendo excepcionales los breakthroughs. Las causas virológicas de fracaso terapéutico se pueden agrupar en (Tabla II): a) errores de genotipado; b) fenómenos de recombinación genética; c) variantes resistentes al tratamiento (pre-existentes o adquiridas tras una exposición inicial a AAD); d) persistencia de la infección, en general con la emergencia de nuevos aislados predominantes; e) reinfección; y f) sobreinfección. Vamos a analizarlas con cierto detenimiento.
Error en el genotipado
El VHC tiene una elevada heterogeneidad genética (8). En función del grado de homología, los aislados del VHC se han agrupado en siete genotipos que muestran hasta un 30% de divergencia en su secuencia nucleotídica. A su vez, los diferentes genotipos muestran notables divergencias de su secuencia (hasta el 20% de la misma) que permite la clasificación en subtipos. Su distribución geográfica es diferente, predominando el genotipo 1 en Estados Unidos, Japón y Europa, mientras que el genotipo 4 se encuentra en Egipto y el genotipo 3 domina en el subcontinente indio. Hace años que conocemos que el genotipo condiciona la RVS, sobre todo en los regímenes basados en IFN, aunque la razón nunca se aclaró de forma definitiva. En la era de la terapia antiviral directa, sigue siendo imprescindible un correcto genotipado del aislado viral, dado que la mayoría de los AAD contra el VHC muestran una diferente sensibilidad en función del genotipo e, incluso, del subtipo, lo que conlleva diferentes tasas de RVS y de patrones de resistencia (9).
El genotipado del VHC es una técnica bien establecida que implica la evaluación de una región altamente conservada del virus pero en la que existen diferencias entre los diversos genotipos y subtipos. Técnicamente, existen tres formas de efectuar el genotipado: mediante la secuenciación directa y posterior análisis filogenético, mediante PCR y posterior hibridación con sondas especificas de genotipo y subtipo y, por último, con técnicas serológicas. El genotipado por métodos serológicos adolece de una pobre sensibilidad y especificidad, lo que hace que su utilización sea marginal y no lo analizaremos. La secuenciación y el posterior análisis filogenético de la secuencia obtenida es la técnica de referencia; no obstante, su utilización de forma rutinaria no se recomienda debido a la elevada variabilidad genética de las regiones habitualmente secuenciadas (NS5b, NS3), lo que dificulta la interpretación de los resultados. Por lo tanto, para genotipar una muestra generalmente recurrimos a una PCR que amplifica una región altamente conservada del genoma del VHC, la región 5' no codificante o región 5' UTR. Además, en los últimos años se han desarrollado ensayos comerciales que son capaces de genotipar en función de las diferencias existentes en la región NS5b (10). Pero a pesar de los avances en la genotipificación del VHC, no es excepcional la imposibilidad de genotipar o subtipar una muestra. Veamos algunos ejemplos recientes: a) sobre una muestra de 1.052 pacientes, Benedet y cols. (10) demuestran que los ensayos comerciales no pueden discriminar en cerca del 9-10% de los casos examinados; b) del mismo modo, Josep Quer y cols. (11), asumiendo la hipótesis de que las técnicas actuales de genotipado producen identificaciones incorrectas, analizan el valor de la secuenciación profunda y el análisis filogenético para la identificación correcta del genotipo, subtipo y las eventuales infecciones mixtas. Tras el análisis de 32 muestras con genotipo no determinado y 81 muestras con genotipo 1 y subtipo no determinado, observan que, aunque el Lipa 2 mejora la identificación de genotipo y subtipo, la secuenciación profunda consituye el método más valioso para un genotipado correcto, consiguiendo que este se haga de forma satisfactoria en todos los casos; y c) finalmente, Silberstein y cols. (12) efectúan un análisis filogenético en 343 pacientes infectados por el VHC antes del inicio de una terapia antiviral libre de IFN. En este subgrupo de pacientes se efectuó el genotipado clásico y, además, se procedió a efectuar una secuenciación de la región NS3 en la mayoría de los casos (NS5a en el 9% y NS5b en el 14% de las muestras). Cuando se analizó más de una región genómica por paciente (n = 52), los resultados de secuenciación fueron concordantes al 100%, lo que confirma la asignación específica de genotipo/subtipo. Por el contrario, la concordancia entre genotipado comercial y secuenciación fue del 91,8%. Además, en todos los pacientes en los que el resultado del genotipado fue indeterminado o mixto, se consiguió tiparlos de forma correcta mediante secuenciación. Por lo tanto, cerca del 8% de los pacientes necesitaron la secuenciación para una asignación correcta de genotipo y/o subtipo. Unos resultados muy similares a estos se habían publicado con anterioridad (13). Aunque no está establecido en la actualidad, probablemente sea interesante caracterizar el genotipo mediante secuenciación en pacientes con genotipos indeterminados o con genotipo 1 en el que no sea posible el subtipado, antes de prescribir una terapia basada en AAD. Por otra parte, un grupo en el cual será muy importante la secuenciación del genoma viral es aquel en el que haya fracasado una terapia previa libre de IFN que incluya dos o más AAD.
Procesos de recombinación genética
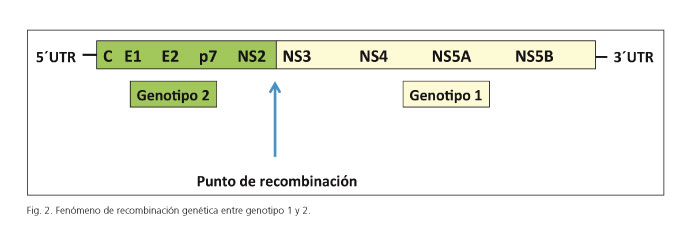
El VHC es un flavivirus que se replica a través de intermedios de ARN. Se sabe que algunos Flaviviridae son capaces de infectar una célula con dos cepas virales diferentes. Y al infectarse una misma célula con dos virus diferentes, se pueden producir fenómenos de "entrecruzamiento" o "crossover" de los intermediarios de RNA que dan lugar a híbridos, virus recombinantes que poseen características de ambos virus. La existencia de esta posibilidad genética había sido reconocida en el VHC, pero se pensó que esta eventualidad era muy rara y de dudosas implicaciones clínicas (14). Sin embargo, puede que estas afirmaciones no sean totalmente ciertas, como demuestra un reciente artículo de Hedskog y cols. (15) en el que tuvimos la oportunidad de colaborar. El trabajo evalúa la existencia de divergencias en el genotipado utilizando dos sistemas: un test que analiza la región 5'NC (InnoLipa) y una técnica de secuenciación de la región 3'NC del virus. La mayoría de las más de 2.000 muestras analizadas fueron concordantes, pero en 12 pacientes se produjeron resultados discordantes, indicando las técnicas que evaluaron el genotipo en la región 5' un genotipo 2 y la secuencia de la región 3' un genotipo 1 (Fig. 2). Posteriormente, se efectuó una secuenciación completa del virus, demostrándose la existencia de virus híbridos con un punto de recombinación en la región NS2/NS3. Y este virus híbrido tuvo importantes consecuencias clínicas. Por ejemplo, nuestro paciente fue caracterizado como genotipo 2 y tratado como tal, con un régimen corto de sofosbuvir y RBV. Tras una respuesta excelente al tratamiento, la viremia reapareció. Esta recidiva fue motivada por que nuestro virus recombinante se comportó como un genotipo 1. De hecho, el comportamiento de todos los virus híbridos fue similar: sólo 3 de los 11 pacientes teóricamente genotipo 2 respondieron al régimen anteriormente expresado. Es evidente que este proceso de recombinación genética puede explicar algunos casos (probablemente pocos) de fallo en el tratamiento antiviral de última generación, facilitando la elección de regímenes subóptimos. Además, y aunque sólo se ha descrito recombinación entre los genotipos 1 y 2, no se puede descartar la exitencia de otros fenómenos de recombinación.
Variantes asociadas a resistencias al tratamiento (RAV)
La enorme producción de viriones diarios (1012 viriones), la elevada tasa de mutación (10-4 a × 10-5 por nucleótido) y la escasa capacidad de reparación de errores de la RNA polimerasa RNA dependiente explican la gran diversidad genética del VHC. Como consecuencia de esta variabilidad, el VHC existe en forma de una multitud de variantes genéticas pero íntimamente relacionadas que se denominan cuasiespecies. Y esta variabilidad genética justifica que el desarrollo de variantes asociadas a resistencias (RAV) sea mayor que en las infecciones por VHB o VIH (16,17).
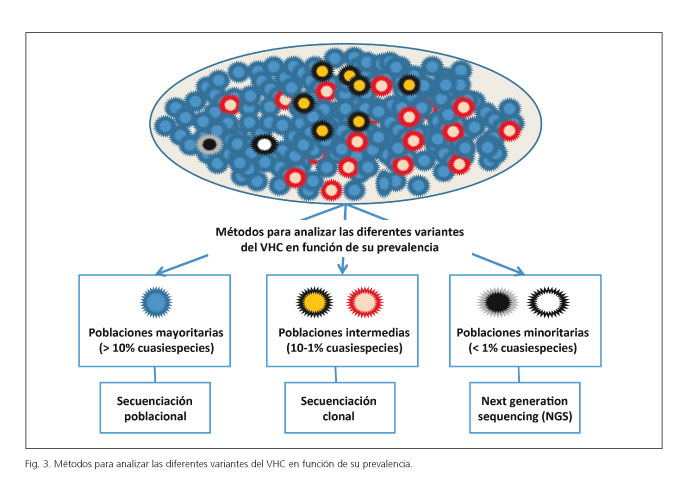
La detección de RAV depende principalmente del método utilizado (Fig. 3). Mediante la secuenciación poblacional, somos capaces de detectar variantes que representen, al menos, el 10% de la población viral; cuando aplicamos técnicas más sensibles como la secuenciación clonal, detectamos variantes que pueden suponer sólo el 1% de la población viral completa y, finalmente, cuando utilizamos técnicas de extraordinaria sensibilidad, como las de Next Generation Sequencing (NGS), podemos analizar incluso poblaciones que suponen menos del 0,5-1% de la muestra total. La propia presencia de las RAV no justifica la resistencia a un tratamiento. En efecto, esta resistencia estará relacionada con: a) la importancia cuantitativa de esta RAV; b) la potencia del régimen antiviral; c) la barrera genética del AAD (número de mutaciones necesarias para que el VHC se haga resistente al AAD); y d) su capacidad replicativa (el fitness viral). Y, aunque este aspecto no tiene todavía respaldo científico, es probable que el desarrollo de RAV aumente como consecuencia de una adherencia a la terapia subóptima.
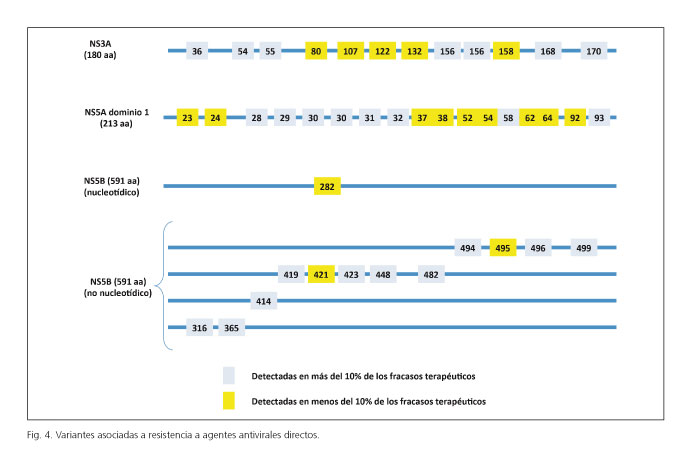
Descripción de las RAV más frecuentes (Fig. 4)
Las RAV que afectan a NS3 se observan con una frecuencia relativamente baja antes del tratamiento (< 3% en naïve y < 7% en tratados con anterioridad) (18). Las que se asocian con más frecuencia a un fallo terapéutico emergen como consecuencia del tratamiento con telaprevir y boceprevir. Quizás la más importante es la R155K/T, cuyo fitness puede mejorar cuando se asocia a otra RAV, la V36M. Las RAV asociadas a inhibidores de la proteasa de segunda generación más frecuentes son, de nuevo, la R155K y la D168A/V/E/T. El polimorfismo Q80K, frecuente en infecciones por el genotipo 1a, se asocia a resistencia a simeprevir (19). Tal y como se observa en la figura 4, existen múltiples variantes potenciales de la región NS5A que pueden inducir resistencia a los inhibidores del complejo NS5A. En el caso del daclatasvir, las más frecuentes son la L31V/M y la Y93H/N (20). Variantes resistentes a ombitasvir se detectan en las posiciones 28, 30, 58 y 93, prácticamente de forma exclusiva en infecciones por genotipo 1a. Los patrones de resistencia en ledipasvir son similares. La sustitución S282T en la región NS5B es la única mutación resistente asociada con una disminución de la susceptibilidad a los inhibidores nucleotídicos de NS5B, del que el sofosbuvir es la molécula paradigmática, la frecuencia de su aparición es mínima y no tiene relación con otras RAV de NS5B (21); fue detectada en el estudio Electron de sofosbuvir en monoterapia, en un paciente infectado con un genotipo 2 y que recidivó en la semana 4 postratamiento (22). Hasta el momento, esta RAV sólo se ha detectado en 4 pacientes. Las RAV de NS5B que confieren resistencia a los inhibidores de NS5B no nucleosídicos (como, por ejemplo, dasabuvir o tegobuvir,) son mucho más frecuentes que las que afectan a los inhibidores nucleotídicos y se relacionan con resistencia y breakthrough viral. En general, son más frecuentes en el genotipo 1a que en el genotipo 1b (23) aunque la variante C316N se observa con más frecuencia en genotipo 1b (24). La RAV C316N/H/F ha sido detectada de forma basal en 6 pacientes con VHC genotipo 1b que fallaron al tratamiento con sofosbuvir y en un paciente genotipo 1a que presentó una recidiva tras el mismo; sin embargo, se necesitan más estudios para establecer correctamente el papel de las RAV como causa de resistencia al tratamiento con sofosbuvir (25).
Significado clínico de las RAV
La información disponible acerca de las RAV que pueden conferir resistencia a los AAD es cada vez mayor; sin embargo, muchos aspectos no se conocen en profundidad en este momento (26-28). Asumiendo que nuestra información es incompleta y que, probablemente, algunas de las afirmaciones puedan matizarse en un futuro inmediato, las preguntas más relevantes son:
- ¿La presencia de RAV basales se correlaciona con una menor RVS? No existe una contestación única a esta pregunta. Como norma general, las RAV basales no tienen una influencia decisiva en la probabilidad de obtener una RVS, sobre todo las RAV de NS3 en poblaciones minoritarias (< 1%). El impacto de las RAV en NS5A es variable, siendo más frecuente la ausencia de RVS si se asocian a otros factores predictivos negativos como la presencia de cirrosis (29). Algunos autores demuestran que en genotipos 1a el compromiso de la actividad de los inhibidores de NS5a condicionado por las RAV es mayor que en genotipo 1b (30). También se ha demostrado el impacto potencial en el retratamiento, como se demuestra en dos estudios: el primero, en el que todos los fracasos terapéuticos se producen en el grupo de pacientes con RAV (31), y el segundo, en el que la práctica totalidad de los 22 pacientes (sobre 471 pacientes tratados con grazoprevir y elbasvir) que no alcanzaron la RVS tenían RAV y, además, en muchos casos ya estaban presentes antes del inicio del tratamiento (32). Pero no todos los estudios son concordantes, como lo demuestra el hecho de que en 94 casos con RAV basales (de una serie de 511, siendo el 18% cirróticos) tratados con sofosbuvir y ledipasvir, la tasa de RVS fue similar en los pacientes con y sin RAV basales (33). Por el contrario, el estudio ION-3 demuestra que en los pacientes que habían recibido tratamiento previo durante 8 semanas y no presentaron RVS, el retratamiento durante 24 semanas se asoció a una tasa muy baja de RVS (inferior al 50%) si presentaban dos o más RAV basales para NS5A (34).
- ¿Las RAV persisten o tienden a desaparecer? La persistencia a largo plazo se ha evaluado en un número muy elevado de pacientes y se ha comunicado recientemente en el último Congreso de la EASL. Las RAV a los inhibidores de las proteasas desaparecen, tanto cuando surgen en tratamientos con simeprevir (35) como cuando lo hacen con regímenes que incluyen paritaprevir (36) o grazoprevir (32). Sin embargo, las RAV asociadas al tratamiento con inhibidores de NS5A persisten a lo largo del tiempo, tanto en pacientes tratados con ledipasvir (31) como con ombitasvir (36) o elbasvir (32). Del mismo modo, las RAV asociadas a inhibidores no nucleotídicos de NS5B, como dasabuvir, también persisten en el tiempo (36). Además, esta persistencia puede ser muy prolongada, como lo demuestran Dvory-Sobol y cols., que confirman la presencia de RAV de NS5A hasta 96 semanas después de finalizado el tratamiento, lo que sugiere un fitness de estas variantes (31).
- ¿Tratamiento guiado por RAV? Un aspecto muy interesante es saber si el régimen terapéutico debe guiarse por la presencia previa de RAV. En 312 pacientes europeos, se investigaron las RAV de las regiones NS3, NS5A y NS5B potencialmente relevantes para el tratamiento con telaprevir, simeprevir, asunaprevir, daclatasvir, ledipasvir, ombitasvir y dasabuvir. No se observaron RAV basales resistentes a SOF. Se detectaron RAV de NS3 en el 20,5% de los casos, de NS5A en el 11,5% (más frecuentes en el genotipo 1b que en el 1a) y de NS5B en el 21,5% (también más frecuentes en genotipo 1b). Los autores concluyen que el estudio de las RAV basales puede ayudar en la selección del mejor tratamiento posible y en la optimización del coste de tratamiento (37). En este momento, las principales sociedades científicas no aprueban o no se posicionan acerca del estudio de las resistencias antes del inicio del tratamiento (38-40) salvo en el caso de la búsqueda del polimorfismo Q80K en pacientes con genotipo 1a que van a iniciar tratamiento con un régimen antiviral que incluye simeprevir.
Reinfecciones, superinfecciones, sobreinfecciones
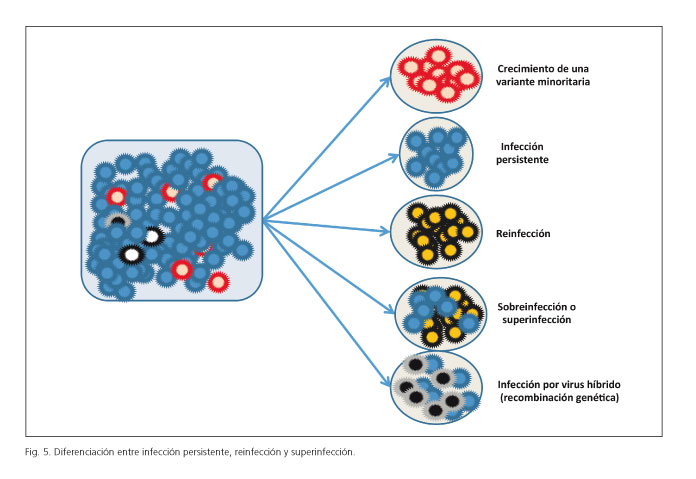
La reinfección por el VHC es frecuente en algunos colectivos, particularmente en hombres homosexuales con una coinfección por el VIH (41). Se ha producido un incremento de la incidencia de hepatitis aguda por VHC entre los hombres infectados por el VIH que tienen sexo con otros hombres (HSH), tanto en Europa como en Australia y Estados Unidos (42,43). En su congreso de noviembre de 2014, la American Association for the Study of Liver Diseases (AASLD) presentó un extenso estudio, posteriormente publicado en Clinical Infectious Diseases (44), cuyo objetivo prioritario era analizar el beneficio clínico de la RVS. Pero, además, analizó el riesgo de reinfección en una población de más de 8.000 pacientes que obtuvieron una RVS tras el tratamiento antiviral, observando que el riesgo de reinfección (con un periodo de obsevación que osciló entre 3 y 5 años) era diferente en función de la población analizada (0,9% en pacientes sin factores de riesgo, 8,2% en usuarios de drogas pro vía parenteral y/o internos en instituciones penitenciarias, y de hasta el 23% en pacientes coinfectados, particularmente en HSH). Pero los estudios citados carecen de un estudio virológico sistemático. Para establecer una correcta definición del diferente tipo de infecciones en un determinado paciente con fracaso a la terapia antiviral, es necesario el uso de una tecnología a la que, todavía, no estamos acostumbrados los clínicos: secuenciación profunda mediante NGS y análisis filogenético. De esta forma, podremos distinguir varias situaciones (Fig. 5):
- Infección persistente: se define así a aquella infección en la que somos capaces de detectar idénticas variantes antes y después del tratamiento. El peso relativo de cada variante (o dominancia) puede cambiar de forma sustancial, pero el análisis filogénetico de estas variantes demuestra que existe una divergencia inferior al 10% (un único origen filogenético). Es probable que la recidiva de la infección no sea más que una infección persistente que en algún momento es de nivel tan bajo que se haya hecho indetectable.
- Reinfección o nueva infección por VHC: se define como la presencia de variantes después del tratamiento sin ninguna relación filogenética (más de 10% de divergencia) con las variantes basales. La reinfección es obvia cuando el genotipo varía.
- Sobreinfección o superinfección: en estos casos, se detectan variantes postratamiento con clara relación filogenética con la variante pretratamiento (infección persistente), además de variantes sin relación con los aislados originales (reinfección).
Recientemente se ha demostrado de forma fehaciente la necesidad de analizar el problema del fallo virológico mediante el uso de NGS (45). Mediante el estudio de la dinámica de las cuasiespecies utilizando secuenciación convencional, clonal y profunda, en pacientes con alto riesgo de reinfección (homosexuales con coinfección VHC y VIH) en los que ha fallado el tratamiento antiviral, intentan diferenciar entre infección persitente y reinfección. De 99 tratados, 15 no alcanzaron la RVS: estos pacientes constituyen el núcleo del análisis. La interpretación clínica y la de la secuenciación convencional fue que en 10 de estos pacientes se había producido una reinfección. Sin embargo, el uso de la NGS reveló que los 15 pacientes (100%) tenían evidencia de infección persistente, aunque en 6 de 15 casos la variante no se había detectado con anterioridad. Los autores concluyen que, incluso en grupos de alto riesgo de reinfección, es más probable la persistencia de la infección que la reinfección, siendo esta sobreestimada por la secuenciación convencional. Este trabajo aporta numerosos aspectos novedosos que deben ser tenidos en cuenta en el futuro: a) es posible la detección de múltiples cepas virales antes del tratamiento; este hecho puede ser debido a una infección simultánea o a una superinfección (46). Por otro lado, la presencia de nuevas variantes (no detectadas en la muestra basal, pero del mismo origen filogenético) puede explicarse por una selección inducida por el tratamiento o por la existencia de diferentes variantes en diferentes compartimentos, como el sistema nervioso central (47); b) implicaciones terapéuticas: si el paciente tiene una infección persistente, es probable que la mejor estrategia sea un tratamiento más prolongado, mientras que en una reinfección deberemos adaptar la combinación de AAD a la nueva cepa infectante (48); y c) por último, este estudio tambien cuestiona la veracidad de la alta tasa de reinfección, postulando que una parte importante de las supuestas reinfecciones son, en realidad, infecciones persistentes. Y esta afirmación también se sustenta en algunas publicaciones que demuestran una tasa de recidivas a largo plazo muy bajas en el colectivo de coinfectados (49).
Pero si bien el estudio que hemos analizado cuestiona la existencia de, al menos, algunas reinfecciones, Sarrazin aporta una interesante aproximación a este problema. Analiza la concordancia entre la RVS en la semana 12 y 24 en más de 3.000 pacientes incluidos en las fases 3 de sofosbuvir, observando que esta es concordante en todos los pacientes salvo en 12. Y en estos 12, analiza la secuencia viral (completa en 10 y de la región NS5B en 2). Asume que cuando el genotipo es diferente, se ha producido una reinfección y cuando el genotipo es el mismo, la decisión acerca de si es una reinfección o una infección persistente dependerá de la distancia filogenética. Sus resultados demuestran que ambas situaciones son posibles (7 reinfecciones, 5 infecciones persistentes), siendo indistinguibles desde el punto de vista clínico (50)
Implicaciones potenciales y algunas recomendaciones
En este momento en que la terapia con AAD es capaz de curar la infección por el VHC en la mayoría de los pacientes, es deseable que prestemos una atención especial a los pacientes en los que no conseguimos dicha curación. Y este hecho está motivado por la dificultad intrínseca que tiene conocer el mecanismo por el cual ha fracasado el tratamiento y, lo que es más importante, por la ausencia de unas directrices claras para retratar a los pacientes en los que fracasa esta terapia antiviral libre de IFN. Tras la exposición de las causas asociadas al fracaso terapéutico quizás sea adecuado enunciar algunas reflexiones en modo alguno dogmáticas:
- Es imprescindible genotipar adecuadamente a todos los pacientes antes de iniciar la terapia antiviral. En este momento, el genotipado debe efectuarse mediante PCR convencional. Teniendo en cuenta el elevado precio de los fármacos y el potencial riesgo de establecer un régimen terapéutico inadecuado especialmente en pacientes con una enfermedad avanzada, así como el relativamente bajo precio de la secuenciación, creemos que en un futuro cercano se procederá a la secuenciación basal de un número cada vez mayor de pacientes. Antes de aplicar esta nueva tecnología, se deben efectuar estudios coste-eficacia que avalen dicha secuenciación. Además, esta secuenciacion permitiría no sólo tipar bien a todos los pacientes, sino conocer la potencial existencia de RAV, favoreciendo la aplicación de un tratamiento a la carta.
- Tal y como se expone con claridad en la última actualización de las guías de la EASL, no existe una evidencia científica suficiente para recomendar un régimen terapéutico determinado tras el fallo de una terapia antiviral libre de IFN que incluya dos o más AAD. Pero, dado que en algunas ocasiones el tratamiento deberá ser instaurado de forma urgente, sí parece razonable intentar hacer una aproximación práctica al problema:
• En primer lugar, debemos categorizar adecuadamente al paciente: existencia o no de cirrosis, función hepática, riesgo de descompensación, grado de hipertensión portal, potencial entrada en lista de espera de trasplante hepático, reinfección del injerto hepático. Es decir, debemos evaluar la urgencia del retratamiento. Además, debemos pensar en la posibilidad de una reinfección o una sobreinfección, interrogando en este sentido al paciente.
• En segundo lugar, debemos analizar con detenimiento las características del régimen antiviral prescrito, incluyendo tipo de fármacos empleados, uso o no de RBV, potenciales problemas de adherencia, momento en que se ha producido el fallo terapéutico, etc.
• Y, en tercer lugar, debemos estudiar detenidamente las características virológicas: carga viral, genotipo y subtipo. Como hemos visto con anterioridad, no son excepcionales la aparación de reinfecciones, el error en el genotipado o subtipado, la presencia de variantes resistentes al tratamiento y, aunque con menos frecuencia, pueden surgir fenómenos de recombinación genética. En este sentido, y aunque ninguna guía lo recomienda de forma explícita, creemos que en todos los pacientes con un fracaso a un régimen antiviral de última generación es deseable una secuenciación completa del genoma del VHC con el objeto de descartar/confirmar alguna de las posibles causas de fallo virológico que hemos analizado en esta revisión. Y, dado que la tecnología de secuenciación no está al alcance de todos los centros, parece imprescindible la homologación de centros de referencia con esta finalidad. En cualquier caso, lo que sí está al alcance de todos es la conservación de una muestra basal del paciente a -70 oC para efectuar un estudio virológico profundo cuando sea posible.
Teniendo en cuenta las consideraciones generales efectuadas, se pueden hacer algunas recomendaciones muy genéricas y que, presumiblemente, cambien en muy poco tiempo:
- Dado que sofosbuvir tiene una alta barrera genética para la resistencia y que la existencia de RAV es excepcional, la mayoría de los pacientes con fallos a terapia antiviral con AAD deben ser tratados con un régimen que incluya sofosbuvir. Este régimen debe ser libre de IFN y añadir si es posible un AAD de una familia que no se haya utilizado previamente.
- Probablemente el tratamiento debe incluir RBV, aunque no se conoce si la adición de esta o la prolongación del tratamiento son igual de efectivas. Se desconoce si la ribavirina es capaz de disminuir las resistencias al tratamiento.
- Es muy importante que recordemos que los fracasos a un régimen que contenga un inhibidor de NS5A, probablemente contengan RAV con resistencia cruzada a cualquier NS5A y que estas variantes no tienden a desaparecer (51-53).
- Los pacientes sin una necesidad urgente de tratamiento pueden esperar hasta que surjan más datos y/u opciones terapéuticas alternativas que estén contrastadas con ensayos clínicos dirigidos a resolver este problema.
-Quizás sea también adecuado solicitar a los promotores de los ensayos una hoja de ruta, una estrategia predefinida con respecto a la actitud a tomar ante un eventual fracaso del tratamiento antiviral, debiendo adecuar esta actitud al motivo del fracaso
![]() Dirección para correspondencia:
Dirección para correspondencia:
Susana Llerena.
Servicio Aparato Digestivo.
Hospital Universitario Marqués de Valdecilla.
IDIVAL. Facultad de Medicina.
Universidad de Cantabria.
Avda. Los Castros, s/n.
39005 Santander, Cantabria
e-mail: sllerena@humv.es
Recibido: 17-06-2015
Aceptado: 21-09-2015
Bibliografía
1. Choo QL, Kuo G, Weiner AJ, et al. Isolation of a cDNA clone derived from a blood-borne non-A, non-B viral hepatitis genome. Science 1989;2444:359-62. DOI: 10.1126/science.2523562. [ Links ]
2. Plan Estratégico Nacional frente al VHC 2015 (17/06/2015). Available from: http://aeeh.es/wp-content/uploads/2015/04/a77478e2a1147600cb9979b5992281cb.pdf. [ Links ]
3. Jensen DM, O'Leary J, Pockros P, et al. Safety and efficacy of sofosbuvir-containing regimens for hepatitis C: Real-world experience in a diverse, longitudinal observational cohort. 65th Annual Meeting of the AASLD 2014; Abstract 45. Hepatology 2014;60(Supp4):219A. [ Links ]
4. Terrault N. Difficult-to-cure populations with chronic hepatitis C: Vanishing in the DAA era? Hepatology 2015;62:4-7 DOI: 10.1002/hep.27892. [ Links ]
5. Nelson DR, Cooper JN, Lalezari JP, et al. All-oral 12-week treatment with daclatasvir plus sofosbuvir in patients with hepatitis C virus genotype 3 infection: ALLY-3 phase III study. Hepatology 2015;61:1127-35. DOI: 10.1002/hep.27726. [ Links ]
6. Foster GR, Pianko S, Cooper C, et al. Sofosbuvir + peginterferon/ribavirin for 12 weeks vs. sofosbuvir + ribavirin for 16 or 24 weeks in genotype 3 HCV infected patients and treatment-experienced cirrhotic patients with genotype 2 HCV: The BOSON study. J Hepatol 2015;62(Supp2):S259. DOI: 10.1016/S0168-8278(15)30151-3. [ Links ]
7. Saxena V NL, Pauly M, et al. Safety and efficacy of simeprevir/sofosbuvir in hepatitis C infected patients with compensated and decompensated cirrhosis: A matched analysis. J Hepatol 2015;62(Supp2):S669-S70. DOI: 10.1002/hep.27922. [ Links ]
8. Smith DB, Bukh J, Kuiken C, et al. Expanded classification of hepatitis C virus into 7 genotypes and 67 subtypes: Updated criteria and genotype assignment web resource. Hepatology 2014;59:318-27. DOI: 10.1002/hep.26744. [ Links ]
9. Pawlotsky JM. New hepatitis C therapies: The toolbox, strategies, and challenges. Gastroenterology 2014;146:1176-92. DOI: 10.1053/j.gastro.2014.03.003. [ Links ]
10. Benedet M, Adachi D, Wong A, et al. The need for a sequencing-based assay to supplement the Abbott m2000 RealTime HCV Genotype II assay: A 1 year analysis. J Clin Virol 2014;60:301-4. DOI: 10.1016/j.jcv.2014.04.005. [ Links ]
11. Quer J, Gregori, J, Rodríguez-Frías F, et al. High-resolution hepatitis C virus subtyping using NS5B deep sequencing and phylogeny, an alternative to current methods. J Clin Microbiol 2015;53:219-26. DOI: 10.1128/JCM.02093-14. [ Links ]
12. Ceccherini Silberstein F, Di Maio VC, Aragri M, et al. Hepatitis C virus gene sequencing as a tool for precise genotyping in the era of new direct antiviral agents. Hepatology 2015 (In press). DOI: 10.1002/hep.27895. [ Links ]
13. Liu CH, Liang CC, Liu CJ, et al. Comparison of Abbott RealTime HCV Genotype II with Versant Line Probe assay 2.0 for hepatitis C virus genotyping. J Clin Microbiol 2015;53:1754-7. DOI: 10.1128/JCM.03548-14. [ Links ]
14. Morel V, Fournier C, Francois C, et al. Genetic recombination of the hepatitis C virus: Clinical implications. J Viral Hepat 2011;18:77-83. DOI: 10.1111/j.1365-2893.2010.01367.x. [ Links ]
15. Hedskog C, Doehle B, Chodavarapu K, et al. Characterization of hepatitis C virus intergenotypic recombinant strains and associated virological response to sofosbuvir/ribavirin. Hepatology 2015;61:471-80. DOI: 10.1002/hep.27361. [ Links ]
16. Sarrazin C, Kieffer TL, Bartels D, et al. Dynamic hepatitis C virus genotypic and phenotypic changes in patients treated with the protease inhibitor telaprevir. Gastroenterology 2007;132:1767-77. DOI: 10.1053/j.gastro.2007.02.037. [ Links ]
17. Wyles DL, Gutiérrez JA. Importance of HCV genotype 1 subtypes for drug resistance and response to therapy. J Viral Hepat 2014;21:229-40. DOI: 10.1111/jvh.12230. [ Links ]
18. Bartels DJ, Zhou Y, Zhang EZ, et al. Natural prevalence of hepatitis C virus variants with decreased sensitivity to NS3.4A protease inhibitors in treatment-naive subjects. J Infect Dis 2008;198:800-7. DOI: 10.1086/591141. [ Links ]
19. Cabezas J, Menéndez S, Puente A, et al. Polimorfismos del Q80K en una región del norte de España. Gastroenterol Hepatol 2015;38. [ Links ]
20. Wang C, Sun JH, O'Boyle DR, et al. Persistence of resistant variants in hepatitis C virus-infected patients treated with the NS5A replication complex inhibitor daclatasvir. Antimicrob Agents Chemother 2013;57:2054-65. DOI: 10.1128/AAC.02494-12. [ Links ]
21. Svarovskaia ES, Dvory-Sobol H, Parkin N, et al. Infrequent development of resistance in genotype 1-6 hepatitis C virus-infected subjects treated with sofosbuvir in phase 2 and 3 clinical trials. Clin Infect Dis 2014;59:1666-74. DOI: 10.1093/cid/ciu697. [ Links ]
22. Gane EJ, Stedman CA, Hyland RH, et al. Nucleotide polymerase inhibitor sofosbuvir plus ribavirin for hepatitis C. N Engl J Med 2013;368:34-44. DOI: 10.1056/NEJMoa1208953. [ Links ]
23. Zeuzem S, Buggisch P, Agarwal K, et al. The protease inhibitor, GS-9256, and non-nucleoside polymerase inhibitor tegobuvir alone, with ribavirin, or pegylated interferon plus ribavirin in hepatitis C. Hepatology 2012;55:749-58. DOI: 10.1002/hep.24744. [ Links ]
24. Bartels DJ, Sullivan JC, Zhang EZ, et al. Hepatitis C virus variants with decreased sensitivity to direct-acting antivirals (DAAs) were rarely observed in DAA-naive patients prior to treatment. J Virol 2013;87:1544-53. DOI: 10.1128/JVI.02294-12. [ Links ]
25. Donaldson EF, Harrington PR, O'Rear JJ, et al. Clinical evidence and bioinformatics characterization of potential hepatitis C virus resistance pathways for sofosbuvir. Hepatology 2015;61:56-65. DOI: 10.1002/hep.27375. [ Links ]
26. Pawlotsky JM. Treatment failure and resistance with direct-acting antiviral drugs against hepatitis C virus. Hepatology 2011;53:1742-51. DOI: 10.1002/hep.24262. [ Links ]
27. Pawlotsky JM. Is hepatitis virus resistance to antiviral drugs a threat? Gastroenterology 2012;142:1369-72. DOI: 10.1053/j.gastro.2011.12.060. [ Links ]
28. Sarrazin C, Zeuzem S. Resistance to direct antiviral agents in patients with hepatitis C virus infection. Gastroenterology 2010;138:447-62. DOI: 10.1053/j.gastro.2009.11.055. [ Links ]
29. Farnik H, Vermehren J, Susser S, et al. Epidemiology of viral resistance in genotype 1 infected patients at approval of IFN-free DAA combination therapy of chronic hepatitis C in Germany. J Hepatol 2015;62:S616. DOI: 10.1016/S0168-8278(15)30969-7. [ Links ]
30. Cook J, Solberg O, Newton A, et al. Characterization of naturally occurring resistance to HCV NS5A inhibitors. (CROI Abstract 696) Conference on Retroviruses and Opportunistic Infections. Top Antivir Med 2015;23(e-1):307. [ Links ]
31. Dvory-Sobol HWD, Ouyang W, et al. Long-term persistence of HCV NS5A variants after treatment with NS5A inhibitor Ledipasvir. J Hepatol 2015;62(Supp2):S221. DOI: 10.1016/S0168-8278(15)30073-8. [ Links ]
32. Black S, Pak I, Ingravallo P, et al. Resistence analysis of virologic failures in hepatitis C genotype 1 infected patients treated with Grazoprevir/Elbasvir +/- Ribavirin: The C-Worthy study. J Hepatol 2015; 62(Supp2):S677. [ Links ]
33. Sarrazin C, Dvory-Sobol H, Svarovskaia ES, et al. The prevalence and the effect of HCV NS5A resistence associated variants in subjects with compensated cirrhosis treated with ledipasvir/sofosbuvir +/- RBV. J Hepatol 2015;62(Supp2):S620 DOI: 10.1016/S0168-8278(15)30073-8. [ Links ]
34. Lawitz E, Flamm S, Yang JC, et al. Retreatment of patients who failed 8 or 12 weeks of ledipasvir/sofosbuvir-based regimens with ledipasvir/sofosbuvir for 24 weeks. J Hepatol 2015;62(Supp2):S192. DOI: 10.1016/S0168-8278(15)30012-X. [ Links ]
35. Fevery B, Thys K, Van Eygen V, et al. Deep squencing analyses of minority baseline polymorphisms and persistence of emerging mutations in HCV senotype 1 infected patients treated with simeprevir. J Hepatol 2014;60:S139. DOI: 10.1016/S0168-8278(14)60382-2. [ Links ]
36. Krishnan PTR, Schnell G, et al. Long-term follow-up of treatment-emergent resistence-associated variants in NS3, NS5A and NS5B with paritaprevir/R-, ombitasvir- and dasabuvir-based regimens. J Hepatol 2015;62(Supp2):S220. DOI: 10.1016/S0168-8278(15)30071-4. [ Links ]
37. Dietz J, Berkowski C, Perner D, et al. Consideration of viral resistence for optimization of direct antiviral therapy of chronic hepatitis C. J Hepatol 2015;62(Supp2):S681. DOI: 10.1016/S0168-8278(15) 31103-X. [ Links ]
38. AASLD. Recommendations for testing, managing, and treating hepatitis C 2015 (17/06/2015). Available from: http://www.hcvguidelines.org/full-report-view. [ Links ]
39. EASL. Recommendations on treatment of hepatitis C 2015 2015 (17/06/2015). Available from: http://www.easl.eu/research/our-contributions/clinical-practice-guidelines/detail/recommendations-on-treatment-of-hepatitis-c-2015. [ Links ]
40. AEEH. Documento del II Consenso español sobre tratamiento de la hepatitis C 2015 (17/06/2015). Available from: http://aeeh.es/wp-content/uploads/2015/04/II-Conferencia-de-consenso-sobre-el-tratamiento-de-la-hepatitis-C-de-la-AEEH.pdf. [ Links ]
41. Serpaggi J, Chaix ML, Batisse D, et al. Sexually transmitted acute infection with a clustered genotype 4 hepatitis C virus in HIV-1-infected men and inefficacy of early antiviral therapy. Aids 2006;20:233-40. DOI: 10.1097/01.aids.0000200541.40633.56. [ Links ]
42. Thomson EC, Main J. Epidemiology of hepatitis C virus infection in HIV-infected individuals. J Viral Hepat 2008;15:773-81. [ Links ]
43. Grebely J, Pham ST, Matthews GV, et al. Hepatitis C virus reinfection and superinfection among treated and untreated participants with recent infection. Hepatology 2012;55:1058-69. DOI: 10.1002/hep.24754. [ Links ]
44. Simmons B, Saleem, J, Heath K, et al. Long-term treatment outcomes of patients infected with hepatitis C virus: A systematic review and meta-analysis of the survival benefit of achieving a sustained virological response. Clin Infect Dis 2015;61:730-40. DOI: 10.1093/cid/civ396. [ Links ]
45. Abdelrahman T, Hughes J, Main J, et al. Next-generation sequencing sheds light on the natural history of hepatitis C infection in patients who fail treatment. Hepatology 2015;61:88-97. DOI: 10.1002/hep.27192. [ Links ]
46. Pham ST, Bull RA, Bennett JM, et al. Frequent multiple hepatitis C virus infections among injection drug users in a prison setting. Hepatology 2010;52:1564-72. DOI: 10.1002/hep.23885. [ Links ]
47. Forton DM, Karayiannis P, Mahmud N, et al. Identification of unique hepatitis C virus quasispecies in the central nervous system and comparative analysis of internal translational efficiency of brain, liver, and serum variants. J Virol 2004;78:5170-83. DOI: 10.1128/JVI.78.10.5170-5183.2004. [ Links ]
48. McNaughton AL, Thomson EC, Templeton K, et al. Mixed genotype hepatitis C infections and implications for treatment. Hepatology 2014;59:1209. DOI: 10.1002/hep.26544. [ Links ]
49. Soriano V, Vispo E, De Mendoza C, et al. Very late relapse after discontinuation of antiviral therapy for chronic hepatitis C. Antivir Ther 2013;18:1033-5. DOI: 10.3851/IMP2659. [ Links ]
50. Svarovskaia E, Martin R, Chodavarapu Chodavarapu K, et al. HCV reinfection in phase 3 studies of sofosbuvir. J Hepatol 2015;62(Supp2):S222-S3. DOI: 10.1016/S0168-8278(15)30077-5. [ Links ]
51. Wang C, Sun JH, O'Boyle DR, et al. Persistence of resistant variants in hepatitis C virus-infected patients treated with the NS5A replication complex inhibitor daclatasvir. Antivir Ther 2013;57:2054-65. DOI: 10.1128/AAC.02494-12. [ Links ]
52. Krishnan P, Tripathi R, Schnell G, et al. Pooled analysis of resistance in patients treated with ombitasvir/ABT-450/r and dasabuvir with or without ribavirin in phase 2 and phase 3 clinical trials. Hepatology 2014;60:1134A. [ Links ]
53. Sarrazin C, Dvory-Sobol H, Svarovskaia ES, et al. Baseline and post-baseline resistance analyses of phase 2/3 studies of ledipasvir/sofosbuvir RBV. Hepatology 2014;60:1128A. [ Links ]











 text in
text in