INTRODUCCIÓN
Escherichia coli es un bacilo Gram negativo de entre 1,1 - 1,5 x 2 - 6 µm perteneciente a la familia Enterobacteriaceae. Aparecen aislados o en parejas, no forman esporas y pueden ser móviles gracias a flagelos perítricos. Son aerobios y anaerobios facultativos por lo que disponen de metabolismo respiratorio y fermentativo. Son oxidasa-negativo y forman ácidos y gas a partir de la mayor parte de los hidratos de carbono fermentables.
Son mesófilos típicos, ya que puede crecer a temperaturas comprendidas entre 7ºC y 50ºC, alcanzando su crecimiento óptimo a 37ºC, con valores de pH próximos a la neutralidad y un valor mínimo de actividad de agua de 0,95.
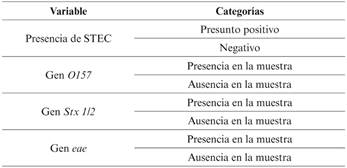
Para realizar el serotipado de las diferentes cepas, se ha utilizado la estructura antigénica formada por el antígeno somático o antígeno O, el antígeno flagelar o antígeno H y el antígeno capsular o antígeno K.
El antígeno somático se caracteriza por ser un lipopolisacárido presente en la membrana exterior de la bacteria. Si utilizamos el sistema propuesto por Kauffman, se conocen del orden de 180 antígenos somáticos (O1 a O185)1-3.
El flagelo de E. coli está formado por varias subunidades de una única proteína, denominada flagelina, que está codificada por el gen fliC. Se conocen 56 antígenos flagelares (H1 a H56)1,2,3.
El antígeno capsular radica en el polisacárido ácido capsular (CPS), clasificándose este en dos grupos, I y II4.
Hoy en día, en función de sus mecanismos de patogenicidad y factores de virulencia, las distintas cepas de E. coli se clasifican en seis grupos: E. coli enteropatogénicos (EPEC), E. coli enterotoxigénicos (ETEC), E. coli enteroinvasivos (EIEC), E. coli enteroagregativos (EAEC), E. coli con adherencia difusa (DAEC) y E. coli enterohemorrágicos, verotoxigénicos o productores de toxinas Shiga (EHEC/VTEC/STEC).
El grupo de cepas EHEC/VTEC/STEC, son capaces de producir toxinas, codificadas por bacteriófagos4,5,6,7 muy similares a la producida por Shigella dysenteriae tipo 1. Se han descrito dos tipos: la toxina Shiga 1 (Stx1) o verotoxina 1 (VT1) y la toxina Shiga 2 (Stx2) o verotoxina 2 (VT2); la capacidad de detección de las toxinas Stx activas mediante el test de toxicidad en celulas Vero explica ambas denominaciones (Stx/VT). Stx 1 se divide a su vez en tres subtipos y Stx2 en siete subtipos8.
Las cepas STEC pueden presentar factores de virulencia adicionales. Cabe destacar la intimina, proteína de la membrana externa responsable de la adhesión de las bacterias al epitelio intestinal. Se encuentra codificada en el gen eae que forma parte de la isla de patogenicidad cromosómica denominada Locus for Enterocyte Effacement (LEE). El gen eae está presente en las cepas de algunos de los serotipos más virulentos: O157:H7, O26:H11, O103:H29.
Otro factor de virulencia importante es la enterohemolisina codificada por el gen ehxA y situado en el plásmido EHEC, la presencia del LEE y el plásmido EHEC son marcadores de las cepas clásicas enterohemorrágicas, de los principales serotipos implicados en el 80% de los casos de colitis hemorrágica (CH) y síndrome urémico hemolítico (SUH) en Europa y Estados Unidos10,11,12,13.
La capacidad de adquisición y pérdida de estos factores de virulencia14, supone la principal problemática, desde el punto de vista epidemiológico, de este grupo de microorganismos.
Las toxinas Shiga de E. coli pueden producir diferentes cuadros en los seres humanos, desde diarrea leve a colitis hemorrágica (CH), pudiendo progresar a síndrome urémico hemolítico (SUH), acompañado de anemia hemolítica, trombocitopenia y fallo renal agudo grave. Este cuadro clínico es el responsable de que a este grupo se le denomine frecuentemente E. coli enterohemorrágico (EHEC)15.
La mayoría de brotes y casos de SUH se han relacionado con el serotipo E. coli O157:H7; por ello, podemos encontrar a estos patógenos clasificados en dos categorías: STEC O157 y STEC no-O15716,17.
STEC O157
El primer aislamiento de E. coli O157:H7 se llevó a cabo en 1975 a partir de una muestra de heces de un paciente con diarrea sanguinolenta siendo registrado el primer brote en 1982 por el consumo de hamburguesas17,18.
A pesar de que las cepas STEC no-O157 provocan más casos esporádicos y brotes que STEC O157, es más difícil realizar el estudio epidemiológico y encontrar los alimentos implicados debido a que E. coli O157:H7 es más patógena que la mayoría de las cepas no-O157. Ello conlleva que los brotes de E. coli O157:H7 sean reconocidos e investigados más rápidamente17.
Hoy en día hay muchos alimentos implicados en la transmisión de STEC O157, aunque inicialmente se relacionó con la carne de vacuno. Griffin y Tauxe19 establecieron que aproximadamente el 52% de los brotes alimentarios producidos por E. coli O157:H7 se relacionan con productos de origen bovino. Los productos implicados con mayor frecuencia son hamburguesas y carne picada poco cocinada20.
La leche es una fuente común de infección de E. coli O157:H7, debido fundamentalmente a la contaminación fecal21. El microorganismo se ha aislado de leche cruda, leche pasteurizada y queso22,23,24.
La prevalencia de E. coli O157:H7 en carne de ovino es muy baja, en torno a un 2%25, aunque Chapman26 concluyó que su aislamiento era mayor en productos cárnicos de ovino que en productos cárnicos de vacuno.
Respecto a las especies cinegéticas, además de haber actuado como vectores en brotes de origen alimentario dispersando E. coli O157:H7 en zonas de cultivos27, la bacteria se ha aislado de cecina y solomillo de ciervo28,29.
Se han descrito brotes de E. coli O157:H7 por consumo de espinacas, mezcla de lechugas de cuarta gama, zumo de manzana sin pasteurizar y brotes de rábano27,30,31,32.
STEC NO-O157
Desde que se produjera el primer brote en Japón en 1984 por una cepa de E. coli O145:H-33, los serogrupos no-O157 más relacionados con casos de enfermedad en humanos son el O23, O45, O103, O111, O121 y O145. Estos serogrupos se han denominado como el big six por el Centers for Disease Control and Prevention (CDC)34.
Se han aislado diferentes cepas de STEC no-O157 en carne y derivados cárnicos de todas las especies domésticas de, leche de vaca y oveja, queso de oveja, carne de caza (ciervo (Cervus elaphus Linnaeus, 1758), jabalí (Sus scrofa Linnaeus, 1758), muflón (Ovis orientalis mousimon Pallas, 1762) y carne picada de ternera24,35,36,37,38.
El brote producido por el serotipo O104:H4 en el año 2011 tuvo un gran impacto mediático en Europa. A pesar de la controversia generada en cuanto al origen del brote y, aunque no se pudo aislar la cepa causante en ninguna muestra de alimentos, se atribuyó el origen a unos brotes para ensalada39.
Se ha descrito también la transmisión de STEC no-O157 a través de bebidas tales como sidra de manzana, zumo de frutas y la variedad de lechuga iceberg34,40,41,42,43.
El uso de derivados cárnicos de especies domésticas de abasto es habitual dentro de las Fuerzas Armadas (FAS) así como, y aunque en menor medida, el consumo de carne de especies cinegéticas. Por ello y porque la notificación de casos por STEC sigue aumentando año tras año44,45, el objetivo de este estudio es evaluar la prevalencia de STEC dentro de las FAS.
MATERIAL Y MÉTODOS
Las muestras analizadas en este estudio fueron las siguientes:
-
- 108 muestras recibidas en el Servicio de Bromatología y Seguridad Alimentaria del Centro Militar de Veterinaria de la Defensa (CEMILVETDEF), clasificadas de acuerdo a la reglamentación técnica vigente como46:
-
- 62 muestras de derivados de carne de caza congelados, clasificadas como:
El procedimiento de pre-enriquecimiento fue el mismo para todas las muestras; se tomaron 25 g. y se diluyeron en 225 g. de agua de peptona tamponada, incubándose a 37ºC durante 24 h.
Para la extracción del ADN se utilizó el kit PrepSEQ™ Rapid Spin Sample Preparation Kit siguiendo las instrucciones del fabricante.
Para la investigación de STEC mediante Real Time PCR, se utilizó el kit Custom TaqMan ISO STEC Screening Assay (Thermo Scientific™), según las instrucciones del fabricante.
Las condiciones de amplificación del ensayo mediante PCR fueron:
- Desnaturalización inicial de 2 minutos a 95ºC.
- 40 ciclos de 3 segundos de desnaturalización a 95ºC y 30 segundos de hibridación y extensión a 60ºC.
El criterio para clasificar una muestra a partir de la interpretación de la curva de amplificación es el siguiente:
- Presunto positivo: La amplificación del gen O157 y/o Stx1/2 debe darse antes del ciclo 40.
- Negativo: No se produce amplificación de ningún gen o, únicamente se amplifica el gen eae antes del ciclo 40.
Para realizar el análisis estadístico se han utilizado las siguientes variables:
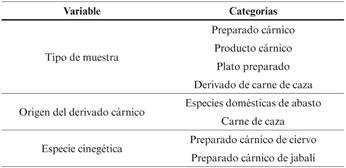
Como variables dependientes se han utilizado las variables cualitativas que se recogen en la Tabla 1 con sus diferentes categorías.
Como variables independientes se han utilizado las variables cualitativas recogidas en la Tabla 3, con sus diferentes categorías.
Se ha tratado de determinar la asociación o independencia de la distribución de las variables dependientes entre las independientes. Para ello se ha utilizado el test Χ2 de Pearson, y en aquellos casos en los que no ha sido posible, se ha utilizado la Prueba Exacta de Fischer.
El error α asumido fue del 5% y se ha utilizado el software de análisis estadístico SPSS® v.19.
RESULTADOS
Análisis descriptivo de los resultados obtenidos
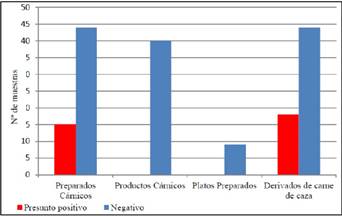
La prevalencia de presuntos positivos STEC en las 170 muestras analizadas es de un 19,41 %.
No se han encontrado presuntos positivos entre los productos cárnicos ni entre los platos preparados. La prevalencia de presuntos positivos STEC entre los preparados cárnicos es 25,42 % (15 muestras) (Figura 1).
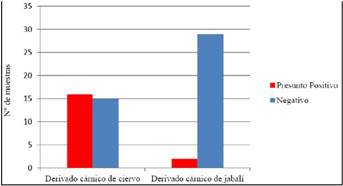
La prevalencia de STEC en derivados de carne de caza es de 27,4 %. En el caso de los preparados cárnicos de jabalí es del 6,8 % (2 muestras) y en el caso de los preparados cárnicos de ciervo es del 51,61 % (Figura 2).
En la Tabla 2 se resumen las combinaciones de genes que hemos encontrado entre las muestras presuntas positivas.
Resultados del análisis estadístico
El resultado obtenido tras realizar el Test Exacto de Fischer para evaluar la asociación entre el tipo de muestra y el resultado del cribado se recoge en la Tabla 4.
Tabla 4 Test Exacto de Fisher para evaluar asociación entre el tipo de muestra y la presencia de STEC.
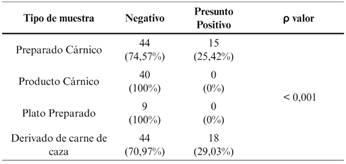
El resultado del cribado de STEC presenta una distribución diferente y estadísticamente significativa en función del tipo de muestra.
El resultado obtenido tras realizar el test Χ2 de Pearson para evaluar la asociación entre el tipo de derivado cárnico y el resultado del cribado, se recoge en la Tabla 5.
Tabla 5 Test Ji-cuadrado de Pearson para evaluar asociación entre el origen de los derivados cárnicos y la presencia de STEC.
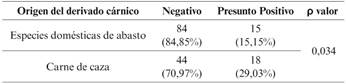
La prevalencia de STEC en los derivados cárnicos correspondientes a especies domésticas de abasto analizados en este estudio, es menor que la hallada en derivados cárnicos de especies cinegéticas, siendo esta diferencia estadísticamente significativa.
El resultado obtenido tras realizar el test Χ2 de Pearson para evaluar la asociación entre la especie cinegética y el resultado del cribado, se recoge la Tabla 6.
Tabla 6 Test Ji-cuadrado de Pearson para evaluar asociación entre la especie cinegética y la presencia de STEC.
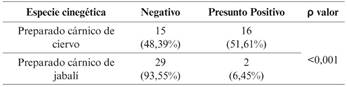
La prevalencia de STEC es diferente en función de la especie cinegética que se trate y, esa diferencia, es estadísticamente significativa.
La prevalencia de los diferentes factores de virulencia dependiendo del origen del derivado cárnico y su significación estadística, se resumen en la Tabla 7.
Tabla 7 Prevalencia de los diferentes genes codificadores de factores de virulencia según el origen de los derivados cárnicos (%).
| Gen | Especies domésticas de abasto | Carne de caza | ρ valor |
|---|---|---|---|
| eae | 22,22 | 61,29 | < 0,001 |
| Stx 1/2 | 7,07 | 27,42 | < 0,001 |
| O157 | 12,12 | 1,61 | 0,017 |
La prevalencia de los genes eae y Stx 1/2 es menor en los derivados cárnicos de especies domésticas de abasto. En cambio, la prevalencia del gen O157 es mayor en estos derivados.
DISCUSIÓN
La prevalencia de nuestros resultados en relación a la presencia de STEC en derivados cárnicos coincide con la obtenida por Auvray et al.47 y Samadpour et al.48, alrededor de un 16%. Adwan y Adwan encontraron una prevalencia similar en 300 muestras de carne de vacuno cruda49.
Entre los estudios que han obtenido una prevalencia menor podemos citar el de Pradel et al.50 con un 11% de positivos entre 411 muestras de carne y derivados cárnicos de vacuno. En el trabajo de Blanco et al.51 se obtuvo una prevalencia del 12% en diversos productos entre los que se encuentra la carne de vacuno mientras que Perelle et al.52 detectaron un 15% de STEC entre 300 muestras de carne de vacuno.
En este sentido, cabe destacar los datos publicados por la Agencia Europea de Seguridad Alimentaria (EFSA) en el año 2014. De las 2522 muestras de carne y derivados cárnicos de vacuno analizadas por 13 estados miembros y Suiza, únicamente el 3% fueron positivas45.
La prevalencia obtenida por Acheson et al.53 supera nuestros resultados en diez puntos. Esto podría explicarse debido a que Acheson et al. utilizaron diferentes caldos de cultivo para realizar el preenriquecimiento de las muestras y el método de cribado incluía un enzimo-inmunoensayo.
El papel de las especies cinegéticas en la transmisión de STEC, ya sea como reservorio o como fuente de infección, es cada vez más importante y los resultados obtenidos están en consonancia con esta afirmación27,28,29,54,55.
La prevalencia de STEC obtenida en este estudio concuerda con la notificada a la EFSA por Italia y Austria45. Sin embargo, si tenemos en cuenta únicamente a los rumiantes salvajes, nuestros resultados duplican lo notificado por estos países. Nuestra prevalencia es tres veces superior a la obtenida en los estudios de Piérard et al.56 y Miko et al.37, realizados en Bélgica y Alemania respectivamente.
No hemos encontrado ningún estudio que investigase la presencia de STEC en la carne de caza, cuya prevalencia superase a la de nuestro trabajo. Esto se debe a que la mayoría de estudios sobre prevalencia de STEC en especies cinegéticas se basan en el estudio de muestras de heces; la prevalencia de este microorganismo es mayor en el tracto intestinal y debe de producirse contaminación cruzada durante el faenado de la canal para que esta se contamine. Esto podría explicar que sólo hayamos encontrado prevalencias superiores a la nuestra en este tipo de trabajos28,57.
Además, un factor limitante en la comparabilidad de nuestro estudio frente a otros proviene de las características del diseño de nuestra técnica diagnóstica; por un lado detectamos la presencia del gen Stx independientemente del tipo (1 ó 2), y por otro, incluimos el gen O157 como factor de virulencia junto con el resto (stx, eae). Si bien es cierto que en el caso de los estudios de prevalencia en cuanto a la presencia conjunta del gen O157 y el gen eae, nuestros resultados concuerdan con lo publicado por Mora et al.58.