Mi SciELO
Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO -
 Accesos
Accesos
Links relacionados
-
 Citado por Google
Citado por Google -
 Similares en
SciELO
Similares en
SciELO -
 Similares en Google
Similares en Google
Compartir
Revista Española de Enfermedades Digestivas
versión impresa ISSN 1130-0108
Rev. esp. enferm. dig. vol.102 no.1 Madrid ene. 2010
Polimorfismos del gen del factor de crecimiento vascular endotelial en pacientes con cáncer colorrectal
Vascular endothelial growth factor gene polymorphisms in patients with colorectal cancer
M. Vidaurreta1, R. Sánchez-Muñoz2, S. Veganzones1, S. Rafael1, M. Gutiérrez1, V. de-la-Orden1, C. Fernández3, M. Arroyo1, F. J. Cerdán2 y M. Maestro1
Servicio de 1Análisis Clínicos. Sección Biología Tumoral.
2Servicio de Cirugía General y Aparato Digestivo. 3Unidad de Investigación. Hospital Clínico San Carlos. Madrid
Dirección para correspondencia
RESUMEN
Introducción: la angiogénesis juega un papel importante en la progresión de los tumores. El factor de crecimiento endotelial vascular (VEGF) es un importante regulador de la angiogénesis. En este trabajo se han analizado los polimorfismos de único nucleó-tido (SNP) -2578C > A, -1154G > A y +936C > T del gen VEGF en pacientes intervenidos de carcinoma colorrectal, así como su posible implicación pronóstica.
Pacientes y método: el estudio de estos SNP se ha realizado en 177 pacientes intervenidos quirúrgicamente de carcinoma colorrectal (CCR) en el Hospital Clínico San Carlos. El análisis de los polimorfismos se realizó con sondas específicas para cada nucleótido y se determinó mediante una reacción multiplex mediante
real time PCR.
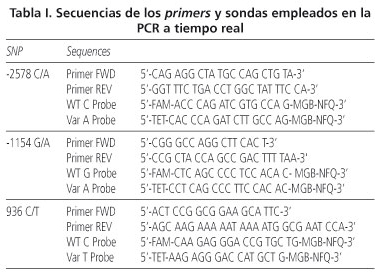
Resultados: de los 3 polimorfismos estudiados sólo encontramos relación estadísticamente significativa del SNP +936C > T con el sexo y la localización. El 10,7% de los pacientes heterocigotos para este SNP tenían como localización el tumor en colon proximal, el 35,2% en colon distal y el resto en recto (p = 0,03). La supervivencia global (SG) de los pacientes con el genotipo +936T/T fue del 100%.
Conclusión: los pacientes con el genotipo +936T/T presentan mayor supervivencia y el polimorfismo +936C > T podría ser una herramienta de ayuda en el seguimiento y terapéutica de este grupo de pacientes.
Palabras clave: VEGF polimorfismos. Cáncer colorrectal.
ABSTRACT
Background: angiogenesis plays an important role in tumor progression. The vascular endothelial growth factor (VEGF) is an important regulator of angiogenesis. In the present study we evaluated single nucleotide polymorphisms (SNPs) -2578C > A, -1154G > A, and +936C > T in the VEGF gene, and their prognostic value for patients operated on for colorectal cancer (CRC).
Patients and method: VEGF polymorphisms have been analyzed in 177 patients who had undergone surgical resection at Hospital Clínico San Carlos. The analysis of these polymorphisms was performed with specific probes for each nucleotide in a multiplex reaction using real-time PCR.
Results: we only found a statistically significant relationship for one of these three polymorphisms, +936C > T, with gender and tumor location; 10.7% of patients heterozygotes for this SNP had tumors located in proximal colon, 35.2% in distal segment and 54.1% in rectum (p = 0.03). Patients with the +936T/T genotype had 100% overall survival (OS).
Conclusion: patients with a +936T/T genotype showed increased survival, therefore the +936C > T SNP could be a useful marker in the follow-up and clinical management of patients with colorectal cancer.
Key words: VEGF polymorphisms. Colorectal cancer.
Introducción
La angiogénesis es un requisito previo para el desarrollo, crecimiento y progresión de los tumores sólidos (1). El factor de crecimiento endotelial vascular (VEGF) es uno de los más potentes mitógenos de células endoteliales y desempeña un papel importante en la angiogénesis (2). Diferentes estudios clínicos han mostrado que el aumento de expresión de VEGF y el incremento de densidad en los vasos sanguíneos de los tumores están asociados con estadios avanzados de la enfermedad y con peor pronóstico en varios tipos de tumores incluido el CCR (3-5).
El gen del VEGF está localizado en el cromosoma 6p21.3 y está constituido por ocho exones que presentan splicing alternativo (6,7). Se han descrito varios polimorfismos de un único nucleótido (SNP) que afectan la expresión de este gen. Estos polimorfismos están asociados con variaciones en la expresión de la proteína VEGF y parecen estar implicados en el desarrollo de diferentes tumores (8,9). Algunos de estos polimorfismos (-2578C > A, -1154G > A, y -634G > C en la región promotora y +936C > T en la región 3'no traducida) presentan asociación con las variaciones en la síntesis de proteína VEGF (7,10). Los genotipos -2578C/A, -1154G/G y -634G/C parecen estar asociados a una mayor expresión de VEGF (11,12), mientras que el alelo +936T se correlaciona con una menor expresión de VEGF (10).
El objetivo de este estudio es analizar tres polimorfismos del gen VEGF: -2578C/A, -1154G/A y +936C/T y evaluar estos SNP con las variables clinicopatológicas y su posible implicación pronóstica en una población de pacientes intervenidos de CCR.
Pacientes y métodos
Población de estudio
La población de nuestro estudio comprende un total de 177 pacientes consecutivos intervenidos quirúrgicamente por CCR primario en el Hospital Clínico San Carlos en Madrid (España) entre marzo de 1995 y enero de 2000. Se trata de un estudio de cohortes prospectivo. Todos los pacientes fueron intervenidos por un mismo cirujano, realizándose una cirugía radical oncológica en función de la localización del tumor. La cirugía se definió como curativa cuando tras la resección no había evidencia de tumor macroscópico residual, no existían metástasis a distancia y en el estudio anatomopatológico no se objetivó ningún margen de resección invadido. Según este criterio, en 145 pacientes (81,9%) se efectuó una resección curativa y en 32 pacientes (18,1%) se resecó el tumor primario con intención paliativa. Fueron excluidos de nuestro estudio los casos de carcinomas metacrónicos, las poliposis familiares, los pacientes con criterios de HPNCC y la enfermedad inflamatoria intestinal. Ninguno de los pacientes había recibido tratamiento neoadyuvante. Se obtuvo el consentimiento informado de los pacientes antes del inicio del estudio. El proyecto fue valorado favorablemente por el comité de ética e investigación clínica de este hospital. El seguimiento clínico de los pacientes se realizó según el protocolo diseñado en nuestro servicio (13). Las variables consideradas a estudio fueron sexo, edad estadio de Dukes, localización del tumor, grado de diferenciación, tipo histológico, quimioterapia adyuvante y SNP del gen VEGF. Los tumores fueron estadificados según la clasificación de Dukes. Se consideró que los tumores estaban localizados en colon proximal cuando eran de colon ascendente y transverso y en el colon distal cuando se localizaban en el colon descendente y sigma. Los pacientes con estadios B y C recibieron quimioterapia adyuvante con 5-fluoracilo (5-FU) y leucovorín, lo que representa el 75% de la población estudiada. En estadio D las pautas de quimioterapia fueron diferentes, según protocolo del Servicio de Oncología.
Las muestras de tejido tumoral y no tumoral fueron obtenidas inmediatamente después de la retirada de la pieza y se introdujeron en nitrógeno líquido para su posterior conservación a -80 oC. El estudio anátomo-patológico lo realizaron 2 patólogos de forma independiente. Se comprobó que todas las muestras tumorales tenían más del 80% de células tumorales.
Para el análisis genético, el ADN fue extraído de las muestras de tejido no tumoral usando DNasy® Blood & Tissue Kit (Qiagen) de acuerdo con las instrucciones del fabricante.
Genotipado de los polimorfismos del VEGF
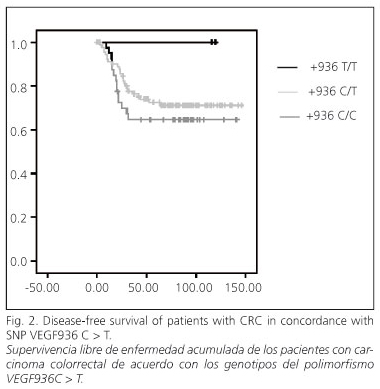
Se determinaron los polimorfismos C-2578A, G-1154A y C936T usando sondas TaqMan® MGB en la reacción en cadena de la polimerasa. Las secuencias de los cebadores sentido y antisentido así como las 2 sondas de cada uno de los tres polimorfismos aparecen reflejadas en la tabla I (14). Los genotipos fueron evaluados por PCR cuantitativa a tiempo real en una reacción multiplex usando un Smart Cycler (CEPHEID, Sunnyvale, USA).
Análisis estadístico
Las variables cualitativas fueron facilitadas con su correspondiente distribución de frecuencias. Las variables cuantitativas fueron expresadas como su media, desviación estándar (DE) y rango. Todas las variables son cualitativas exceptuando la edad y los tiempos. La variable edad fue recodificada en 2 grupos según la mediana (71 años). Se evaluó la asociación entre las variables cualitativas con el test de c2 y, en el caso de que más de un 25% de los esperados fuera menor de 5, por la prueba exacta de Fisher.
Se incluye como definición de evento en la supervivencia global (SG) los fallecimientos producidos como consecuencia del tumor y quedan excluidos los pacientes vivos y fallecidos por otra causa. La SG fue calculada como el tiempo transcurrido entre la fecha de la cirugía y la del fallecimiento o última revisión. En la supervivencia libre de enfermedad (SLE) se define el evento como el diagnóstico de una recidiva locorregional o a distancia en pacientes previamente libres de enfermedad, es decir, todos a los que se realiza cirugía curativa. La SLE fue calculada como el tiempo transcurrido entre la fecha de la cirugía y la de diagnóstico de la primera recidiva. Se estimaron las funciones de SG y SLE por el método de Kaplan-Meier, y se compararon las funciones de supervivencia de los distintos grupos mediante el test exacto de Breslow, debido a que la capacidad de inferencia del estudio está en el tiempo mediano de seguimiento. Se ajustó un modelo de regresión de riesgos proporcionales de Cox. Se presenta la hazard ratio (HR) y su intervalo de confianza (IC) del 95%. Las variables introducidas en el modelo, a partir de los criterios biológicos, fueron las siguientes: sexo, edad, estadio de Dukes, localización del tumor, grado de diferenciación, tipo histológico, quimioterapia adyuvante y SNP del gen VEGF. Se evaluó la asociación de proporcionalidad de riesgos. Se estudió la presencia de interacciones. En todos los contrastes de hipótesis se rechazó la hipótesis nula con un error de tipo I < 0,05.
El paquete informático utilizado para el análisis fue SSPS v.o 11.5 software (SPSS.Inc Chicago, ILL III).
Resultados
De los 177 pacientes del estudio, el 53,7% fueron varones y el 46,3% mujeres. La edad media fue de 71 años (mediana, 72 meses; rango, 35-94 meses). Las variables clinicopatológicas se muestran en la tabla II. El 25,7% de los tumores estaban localizados en el colon proximal, el 29,5% en el colon distal y el 44,8% en el recto. El 8,7% fueron adenocarcinomas mucinosos. En 21 pacientes no se pudo determinar el grado de diferenciación.
La distribución de los genotipos de cada uno de los polimorfismos analizados y sus frecuencias alélicas se muestran en la tabla III. En un paciente no pudo determinarse el genotipo del SNP -1154G > A. El 27,7% de los pacientes presentaban el genotipo A/A en el polimorfismo VEGF -2578C > A, el 22,2% el genotipo A/A en SNP -1154G > A y el 1,1% fueron T/T en el análisis del polimorfismo 936C > T. El 36,7% de los pacientes presentaron el genotipo A/C en el polimorfismo VEGF-2578C > A, el 33,0% el genotipo G/A en SNP -1154G > A y el 25,4% fueron C/T en el análisis del polimorfismo 936C > T.
En el estudio de la relación de los tres polimorfismos con las variables clinicopatológicas sólo en el polimorfismo 936C > T existía relación estadísticamente significativa del genotipo T/T con el sexo y con la localización; no había asociación con el resto de las variables clinicopatológicas (Tabla II). Los 2 pacientes que presentaron el genotipo T/T en este SNP fueron mujeres. Las mujeres presentaron mayor porcentaje del genotipo T/T (2,4%) y menor porcentaje del genotipo C/T (18,3%) que los varones (T/T en el 0% y C/T en el 31,6%) (p = 0,03). El 10,7% de los pacientes con este polimorfismo en heterocigosis (C/T) tenían como localización el tumor en colon proximal, el 35,2% en colon distal y el 27,6% en recto (p = 0,03).
Aunque la relación del genotipo T/T 936C > T con el estadio tumoral no es estadísticamente significativa, sí se observa mayor porcentaje de este genotipo en los tumores localizados (estadios A+B) (p = 0,08).
Se analizaron de forma conjunta los genotipos C/T y T/T (alelo T) del polimorfismo 936C > T (Tabla IV) y se observó que la presencia del alelo T en este polimorfismo se relacionaba significativamente con el estadio de Dukes y la localización. En los tumores localizados el 27,4% de los tumores presentaron el alelo T en su genotipo mientras que el 72,6% no portaba este alelo. El 40% de los tumores en estadio C tenían el alelo T frente al 60% que no presentaban este alelo. En el estadio D sólo el 11,9% de los pacientes presentaba el alelo T (p = 0,01). Con respecto a la localización, en los tumores localizados en colon proximal se observó que el 12,8% presentaban el alelo T y en el 87,2% no existía presencia de este alelo en su genotipo (p = 0,03).
La mediana del tiempo de seguimiento de nuestro estudio fue de 72 meses (6 años), con un intervalo intercuartílico entre 35 y 94 meses. En nuestra población de pacientes la SG a los 6 años fue del 67%. Todos los análisis de supervivencia quedan referidos a nuestra mediana de seguimiento. Durante el seguimiento fallecieron 90 pacientes, 70 de ellos como consecuencia de la neoplasia. En un paciente no se pudo determinar su evolución.
En el análisis univariante, no se observan diferencias significativas en la SG de los pacientes con ninguno de los tres polimorfismos estudiados. Sin embargo, destaca en el análisis del polimorfismo +936C > T que los pacientes con el genotipo +936T/T presentaban una SG del 100%, con +936C/T del 59,8% y con +936C/C del 55,5% aunque no es estadísticamente significativa ya que sólo 2 pacientes presentaron este genotipo (+936T/T) (Fig. 1). La SG de acuerdo con el resto de las variables clinicopatológicas se detalla en la tabla V. Los pacientes que presentaban tumores localizados mostraban una SG del 83,1% frente a los tumores en estadio C y D con SG del 49,3 y 8,0% respectivamente (p < 0,001).
Se realizó un análisis de la SG estratificando la muestra de acuerdo con las variables clinicopatológicas significativas para predecir el riesgo de fallecimiento de los pacientes con carcinoma colorrectal; sin embargo no se obtuvo ningún valor en la SG estadísticamente significativo posiblemente debido al tamaño muestral.
En nuestra población de pacientes la SLE es del 70%. Durante el seguimiento se objetivó una recurrencia en 36 pacientes. La localización de la recurrencia fue locorregional en 11 (30,5%) pacientes y a distancia en 25 (69,5%) pacientes.
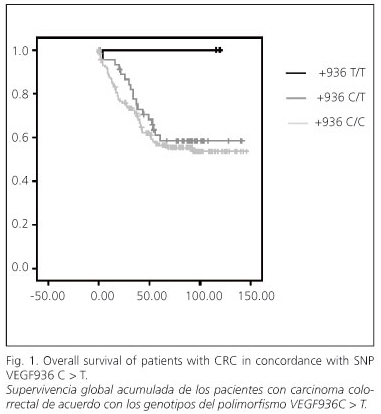
En el análisis univariante, la SLE de los pacientes según el genotipo de sus SNP no presentó variación significativa (Fig. 2). El estudio de la SLE de acuerdo con el resto de las variables clinicopatológicas se detalla en la tabla V. En las mujeres la SLE fue del 77,9% frente a los hombres con una SLE del 63,6% (p = 0,05). Según el estadio de Dukes, los pacientes que presentaron tumores en estadio de Dukes A o B mostraban una SLE del 100%, 77% en estadio C y 48,8% en estadio D (p = 0,003).
En el análisis estratificado de la SLE de la población según las variables clinicopatológicas significativas en el riesgo de recidivar no se observó ninguna diferencia.
Discusión
La angiogénesis es una secuencia que comienza con la dilatación de los vasos y el reclutamiento de los pericitos en los vasos preexistentes seguido de la proliferación de las células endoteliales, formación de nuevos vasos y reclutamiento de células perivasculares. Por tanto, la angiogénesis es una pieza fundamental en el desarrollo del tumor, la invasión y formación de metástasis. El VEGF es uno de los factores de crecimiento que interviene directamente en el control de la angiogénesis (15,16).
El gen VEGF está localizado en el cromosoma 6p12 y está constituido por ocho exones y siete intrones (6). Se trata de un gen muy polimórfico en el que se han descrito al menos 15 SNP (7,17). Algunos de los polimorfismos descritos en este gen (-2578C > A, -1154G > A, -634G > C y +936C > T) han sido asociados a variaciones en la producción de proteína (7,10). En este trabajo hemos analizado la relación de los SNP -2578C > A, -1154G > A y +936C > T del gen VEGF con las variables clinicopatológicas y la supervivencia en una población de 177 pacientes intervenidos por carcinoma colorrectal.
La distribución del polimorfismo -2578C > A en nuestro grupo de pacientes ha sido del 35,6, 36,7 y 37,7% para los genotipos CC, CA y AA, respectivamente. Esta distribución es similar a la obtenida por el grupo de Lemos y cols. (18) en el análisis de este polimorfismo en un grupo de pacientes control. Sin embargo, hemos observado diferencias en la distribución del genotipo del polimorfismo -1154G > A con respecto a los resultados obtenidos por este grupo. En nuestra población, el alelo A se encontró en mayor proporción que en la población control estudiada por Lemos (18) (22 vs. 7%). La distribución del SNP +936C > T en nuestro grupo de pacientes fue similar a la observada por otros autores en el análisis de este polimorfismo en población sana (19,20).
Diferentes estudios han demostrado que la expresión del gen VEGF se relaciona con los SNP localizados en dicho gen. Así el -2578C/A y el -1154G/G se han asociado a una mayor expresión de VEGF (11,12) mientras que la presencia del alelo T en el polimorfismo +936C > T se correlaciona con una menor expresión de la proteína (10). En un metaanálisis realizado por el grupo de Des Guetz y cols. que incluía 27 estudios se demostró que la sobreexpresión de VEGF se correlacionaba significativamente con una menor supervivencia global y un mayor riesgo de recurrencia de la enfermedad (21).
En nuestro estudio existía una relación estadísticamente significativa de la presencia del alelo T del SNP +936C > T con el estadio de Dukes. El alelo T presentaba baja frecuencia en los pacientes en estadio D; por lo tanto, la presencia de este alelo está asociado a estadios precoces de la enfermedad. En el análisis de la SG hemos observado que los pacientes con el genotipo +936T/T presentan una SG del 100%. Sin embargo, otros autores han encontrado una asociación estadísticamente significativa del genotipo +936T/T con estadios avanzados y metástasis a distancia (22) así como una peor supervivencia de aquellos pacientes que presentan este alelo T en su genotipo (23,24).
Estos hallazgos han sido extraídos del análisis de un grupo de 177 pacientes intervenidos de cáncer colorrectal por un mismo cirujano. La prevalencia del genotipo +936T/T en este grupo es del 1,1%, por lo tanto, sería necesario aumentar nuestra población de estudio para corroborar estos datos. Si se confirman estos resultados, el análisis de este polimorfismo podría constituir una ayuda en el seguimiento y terapéutica de estos pacientes.
![]() Dirección para correspondencia:
Dirección para correspondencia:
Mª Luisa Maestro de las Casas.
Servicio de Análisis Clínicos.
Hospital Clínico San Carlos.
C/ Martín Lagos, s/n. 28040 Madrid.
e-mail: mmaestro.hcsc@salud.madrid.org
Received: 25-08-09.
Accepted: 13-10-09.
Bibliografía
1. Yancopoulos GD, Davis S, Gale NW, Rudge JS, Wiegand SJ, Holash J. Vascular specific growth factors and blood vessel formation. Nature 2000; 407: 242-8. [ Links ]
2. Roy H, Bhardawai S, Yla-Herttuala S. Biology of vascular endothelial growth factors. Febs Lett 2006; 580: 2879-87. [ Links ]
3. Yamamori M, Sakaeda T, Nakamura T, Okamura N, Tamura T, Aoyama N, et al. Association of VEGF genotype with mRNA level in colorectal adenocarcinomas. Biochem Biophys Res Commun 2004; 325: 144-50. [ Links ]
4. Lu H, Shu Z-O, Cui Y, Kataoka N, Wen W, Cai Q, et al. Association of genetic polymorphims in the VEGF gene with breast cancer survival. Cancer Res 2005; 65: 5015-19. [ Links ]
5. Tzanakis N, Gazouli M, Rallis G, Giannopoulos G, Papaconstantinou I, Theodoropoulos G, et al. Vascular endothelial growth factor polymorphisms in gastric cancer development, prognosis and survival. J Surg Oncol 2006; 94: 624-30. [ Links ]
6. Vincenti V, Cassano C, Rocchi M, Persico G. Assignment of the vascular endothelial growth factor gene to human chromosome 6p21.3. Circulation 1996; 93: 1493-5. [ Links ]
7. Watson CJ, Webb NJ, Bottomley MJ, Brenchley PE. Identification of polymorphisms within the vascular endothelial growth factor (VEGF) gene: correlation with variation in VEGF protein production. Cytokine 2000; 12: 1232-5. [ Links ]
8. Lin CC, Wu HC, Tsai FJ, Chen HY, Chen WC. Vascular endothelial growth factor gene-460 C/T polymorphism is a biomarker for prostate cancer. Urology 2003; 62: 374-7. [ Links ]
9. Ku KT, Wan L, Peng HC, Tsai MH, Tsai CH, Tasai FJ. Vascular endothelial growth factor gene-460 C/T polymorphism is a biomarker for oral cancer. Oral Oncol 2005; 41: 497-502. [ Links ]
10. Renner W, Kotschan S, Hoffman C, Obermayer-Pietsch B, Pilger E. A common 936 C/T mutation in the gene for vascular endothelial growth factor is associated with vascular endothelial factor plasma levels. J Vasc Res 2000; 37: 443-8. [ Links ]
11. Koukourakis MI, Papazoglou D, Giatromanolaki A, Bougioukas G, Maltezos E, Sivridis E. VEGF gene sequence variation defines VEGF gene expression status and angiogenic activity in non-small cell lung cancer. Lung Cancer 2004; 46: 293-8. [ Links ]
12. Yang B, Cross DF, Ollerenshaw M, Millward BA, Demaine AG. Polymorphisms of the vascular endothelial growth factor and susceptibility to diabetic microvascular complications in patitients with type 1 diabetes mellitus. J Diabetes Complications 2003; 17: 1-6. [ Links ]
13. Cerdán J. Seguimiento de los pacientes intervenidos de cáncer colorrectal. Rev Cancer 1997; 11: 32-41. [ Links ]
14. Schneider BP, Radovich M, Sledge GW, Robarge JD, Li L, Storniolo AM, et al. Association of polymorphisms of angiogenesis genes with breast cancer. Breast Cancer Res Treat 2008; 111(1): 157-63. [ Links ]
15. Ferrara N, Gerber HP, LeCouter J. The biology of VEGF and its receptors. Nat Med 2003; 9: 669-76. [ Links ]
16. Flolkman J. Angiogenesis in cancer, vascular, rheumatoid and ther disease. Nat Med 1995; 1: 27-31. [ Links ]
17. Brogan IJ, Khan N, Isaac K, Hutchison JA, Pravica V, Hutchinson IV. Novel polymorphisms in the promoter and 50 UTR regions of the human vascular endothelial growth factor gene. Hum Immunol 1999; 60: 1245-9. [ Links ]
18. Lemos FB, Mol WM. The beneficial effects of recipient derived vascular endothelial growth factor on graft survival alter kidney transplantation. Transplantation 2005; 79(9): 1221-5. [ Links ]
19. Krippl P, Langsenlehner U, Renner W, Yazdani-Biuki B, Wolf G, Wascher TC, et al. A common 936 C/T gene polymorphism of vascular endothelial growth factor is associated with decreased breast cancer risk. Int J Cancer 2003; 106: 468-71. [ Links ]
20. Wu G, Hasenberg T, Magdeburg R, Bönninghoff R, Sturm J, Keese M. Association between EGF, TGF-B1, VEGF gene polymorphism and colorectal cancer. World J Surg 2009; 33: 124-9. [ Links ]
21. Des Guetz G, Uzzan B, Nicolas P, Cucherat M, Morere JF, Benamouzig R, et al. Microvessel density and VEGF expression are prognostic factors in colorrectal cancer. Meta-analysis of the literature. Br J Cancer 2006; 91(12): 1823-32. [ Links ]
22. Chae YS, Kim JG, Sohn SK, Cho YY, Ahn BM, Moon JH, et al. Association of vascular endothelial growth factor gene polymorphisms wit susceptibility and clinicopathologic characteristics of colorectal cancer. J Korean Med Sci 2008; 23: 421-7. [ Links ]
23. Kim JG, Chae YS, Sohn SK, Cho YY, Moon JH, Park JY, et al. Vascular endothelial growth factor gene polymorphisms associated with prognosis for patients with colorectal cancer. Clin Cancer Res 2008; 14(1): 62-6. [ Links ]
24. Dassoulas K, Gazouli M, Rizos S, Theodoropoulos G, Christoni Z, Nikiteas N, et al. Common polymorphisms in the vascular endothelial growth factor gene and colorectal cancer development, prognosis and survival. Molecular Carcinogenesis 2009; 48: 563-9. [ Links ]











 texto en
texto en