Mi SciELO
Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO -
 Accesos
Accesos
Links relacionados
-
 Citado por Google
Citado por Google -
 Similares en
SciELO
Similares en
SciELO -
 Similares en Google
Similares en Google
Compartir
Anales del Sistema Sanitario de Navarra
versión impresa ISSN 1137-6627
Anales Sis San Navarra vol.30 supl.2 Pamplona 2007
Microbiología de la tuberculosis
Microbiology of tuberculosis
I. Dorronsoro1, L. Torroba2
1. Servicio de Microbiología. Hospital de Navarra. Pamplona.
2. Servicio de Microbiología. Hospital Virgen del Camino. Pamplona.
Dirección para correspondencia
RESUMEN
Durante un siglo, el diagnóstico microbiológico de la tuberculosis, basado en la baciloscopia y en el aislamiento e identificación de Mycobacterium tuberculosis en los cultivos, ha sido poco sensible y lento lo que obligaba en ocasiones a iniciar de forma empírica tratamiento con tuberculostáticos.
La incorporación rutinaria de medios de cultivo líquidos y técnicas de genética molecular, en la última década del siglo XX, ha supuesto un avance importante al aumentar claramente la sensibilidad, precisión y rapidez en el diagnóstico.
La actual eclosión de las técnicas moleculares está permitiendo un mejor conocimiento de la epidemiología de la enfermedad, de los factores de virulencia y de los mecanismos de resistencia lo que dará lugar, en un futuro inmediato, a nuevas estrategias de prevención y tratamiento de la enfermedad.
Palabras clave. Mycobacterium tuberculosis. Diagnóstico microbiológico. Medios líquidos. Genética molecular.
ABSTRACT
For a century, the diagnosis of tuberculosis, based on bacilloscopy and the isolation and identification of Mycobacterium tuberculosis in cultures, has been slow and not very sensitive. This has made it necessary on occasions to initiate treatment with tuberculostatics in an empirical way.
The routine incorporation of liquid mediums and molecular genetic techniques in the final decade of the XX century brought an important advance by clearly increasing the sensitivity, precision and rapidity of diagnosis.
The present blossoming of molecular techniques is making possible a better understanding of the diseases epidemiology, the factors of virulence and the mechanisms of resistance, which in the near future will give rise to new strategies of prevention and for treating the disease.
Key words. Mycobacterium tuberculosis. Microbiological diagnosis. Liquid mediums. Molecular genetics.
Introducción
En 1882, Robert Koch describió el agente etiológico de la tuberculosis (TB) y lo denominó Bacterium tuberculosis1. El nombre inicial fue sustituido por el de Mycobacterium tuberculosis en 1896 por Lehmann y Neumann2. El término Mycobacterium significa hongo-bacteria, y esta denominación se debe al aspecto de los cultivos, que en ciertos aspectos recuerdan a los de los hongos.El descubrimiento de M. tuberculosis causó y sigue causando admiración dadas las características del microorganismo. M. tuberculosis es una bacteria que requiere técnicas especiales de tinción y medios de cultivo distintos a los empleados habitualmente en bacteriología. Además, para poder aislarla, y debido a su lento crecimiento, hay que realizar una decontaminación previa de la mayoría de las muestras, con el fin de destruir la flora acompañante que crece más rápidamente.
Con la llegada de nuevas técnicas de diagnóstico están siendo descritas nuevas especies de Mycobacterium no tuberculosis (MNT), algunas de las cuales están implicadas en la patología humana. Conviene disponer de métodos de identificación rápidos y eficaces pero, debido a la complejidad de las mismas, el nivel del servicio y la elección de los métodos deben ir en función de la población atendida y de los medios disponibles.
Género mycobacterium
El orden de los Actinomycetales incluye la familia Mycobacteriaceae, Actinomycetaceae, Streptomycetaceae y Nocardiaceae. La familia Mycobacteriaceae contiene un solo género, el género Mycobacterium, del que en sus orígenes sólo se conocían dos especies: El bacilo de la lepra o Mycobacterium leprae (A. Hansen 1874) y el bacilo tuberculoso o M. tuberculosis.
Hoy en día, dentro del género Mycobacterium se han descrito más de 120 especies de micobacterias diferentes. Se caracterizan por ser bacterias ácido-alcohol resistentes (BAAR) debido al alto contenido en lípidos que tienen en su pared celular. Este hecho impide que penetren los colorantes habituales de anilina, por lo que no se pueden ver en la tinción de Gram, y hace que para poder visualizarlas sean necesarios colorantes especiales (arilmetanos), pero que una vez teñidas no se decoloran con una mezcla de alcohol y ácido.
Las micobacterias son capaces de sobrevivir durante semanas o meses sobre objetos inanimados, siempre que estén protegidas de la luz solar, y son más resistentes a los ácidos, álcalis y desinfectantes que el resto de las bacterias no formadoras de esporas. Resisten la desecación y la congelación, pero la luz ultravioleta y el calor (>65º C durante 30 minutos) las inactiva3.
Mycobacterium tuberculosis complex
Engloba a un grupo de micobacterias que presentan >95% de homología en su DNA y que se designa como complejo M. tuberculosis (o MTC). Está compuesto, además de M. tuberculosis, por Mycobacterium bovis, M. bovis BCG (una cepa variante de laboratorio utilizada en vacunación y en instilaciones vesicales en pacientes con neoplasia de vejiga), Mycobacterium africanum (principal causante de la TB en África tropical), Mycobacterium microti (causante de la TB en roedores, llamas y otros mamíferos) y Mycobacterium canetii. Habitualmente se utiliza el termino de M. tuberculosis o bacilo tuberculoso como sinónimo de todas ellas, ya que se identifican, en la mayoría de los laboratorios, mediante la hibridación con una sonda de DNA, común para todo el complejo, y porque el aislamiento de cualquiera de ellas en muestras clínicas establece, por sí mismo, el diagnóstico de TB en el paciente. Sin embargo, entre ellas existen características diferenciales importantes para el manejo de los pacientes (p. e., M. bovis es resistente a pirazinamida) y por su interés epidemiológico.
Mycobacterium no tuberculosis (MNT)
Hoy en día, el 10-30 % de las micobacterias aisladas en laboratorios clínicos corresponden al grupo de las micobacterias no pertenecientes al complejo M. tuberculosis. Este grupo heterogéneo de micobacterias ha recibido históricamente otros nombres como micobacterias atípicas, Mycobacterium other than tuberculosis (MOTT), micobacterias ambientales o micobacterias oportunistas. Inicialmente, la nomenclatura de estas especies se realizó atendiendo a su poder patógeno para las distintas especies animales como Mycobacterium simiae o Mycobacterium avium. Posteriormente se fueron aislando otras micobacterias a partir de muestras ambientales (aguas de lagos o ríos, cañerías de los hospitales, suelo, polvo, etc.). Constituyen un grupo muy numeroso, heterogéneo y difícil de sistematizar. Algunas especies son muy ubicuas y poco patógenas; otras son menos ubicuas y su aislamiento tiene un alto valor predictivo de enfermedad; otras no tienen significado clínico conocido y cada vez es más frecuente la descripción de nuevas especies no conocidas previamente. Durante muchos años (primeros 2/3 del siglo XX), apenas se les dio importancia, no obstante, ya en 1954, Timpe y Runyon realizaron un primer intento de clasificación de las MNT aisladas de pacientes, basada en sus características de crecimiento y pigmentación (Tabla 1). Tras el declive progresivo de la TB en los países desarrollados y la irrupción del SIDA, se fue acumulando más experiencia sobre casos clínicos debidos a MNT. En cualquier caso, y a diferencia de MTC, el aislamiento de MNT en muestras de pacientes no es criterio suficiente para establecer el diagnóstico de enfermedad por micobacterias y son poco o nada contagiosas entre las personas.
Debido a la frecuencia con que producen enfermedad (micobacteriosis) o bien porque se aíslan con relativa frecuencia en los laboratorios clínicos vamos a reseñar algunas de ellas:
Mycobacterium avium
Se ha aislado del agua, plantas, tierra y otras fuentes ambientales. Tienen baja patogenicidad y conocer el verdadero significado clínico de su aislamiento a partir de muestras respiratorias suele ser muy difícil. Cuando se aíslan repetidamente ante cuadros clínicos similares a TB y no se encuentran otros patógenos puede ser que tengan significación clínica. Entre 1983 (aparición del SIDA) y 1997 (eclosión de la terapia antiretroviral intensiva) fue la causa mas frecuente de infección por micobacterias en los Estados Unidos. Solía afectar a pacientes con recuentos de CD4 inferiores a 50/mm3, dando lugar a un cuadro diseminado (con aislamiento en hemocultivos) que podía afectar a cualquier órgano. Cuando fue descrito este cuadro, la TB en EEUU se hallaba muy controlada y la infección por M. avium fue una de las formas más frecuentes de presentación del SIDA5,6. En otros países como es el caso de España, la incidencia de TB al descubrirse el SIDA era alta y la principal infección por micobacterias en los pacientes coinfectados con el VIH, siempre ha sido la tuberculosis, siendo la infección por M. avium mucho menos frecuente7. La morfología del microorganismo en las tinciones es cocobacilar y sus colonias en los medios de cultivo son muy heterogéneas.
Mycobacterium kansasii
Es una de las especies que se aísla con más frecuencia en casos de micobacteriosis. Produce cuadros clínicos semejantes a la tuberculosis y su aislamiento a partir de muestras respiratorias casi siempre suele tener significado clínico. Pese a ello, antes de atribuirle un significado clínico definitivo se debe procurar su aislamiento repetido, al igual que ocurre con todas las MNT8. Su morfología en la baciloscopia suele ser característica.
Mycobacterium gordonae
Se aísla con frecuencia y no suele tener significación clínica. Hay muy pocos casos, aproximadamente 20, documentados de enfermedad producida por M. gordonae.
En cuanto al resto de micobacterias, hay algunas a las que se atribuyen cuadros clínicos específicos, como las infecciones de piel debidas a M. marinum y M. ulcerans (causante este último de la úlcera de Buruli), y otras que precisan enriquecimientos especiales de los medios de cultivo para poder crecer (Mycobacterium haemophilum y Mycobacterium genavense).
Diagnóstico microbiológico
El diagnóstico microbiológico de la tuberculosis exige la detección, aislamiento e identificación de M. tuberculosis, así como la determinación de su sensibilidad frente a las drogas tuberculostáticas. La detección precoz del individuo bacilífero es uno de los pilares básicos de una buena organización en la lucha contra la tuberculosis.
La sensibilidad y especificidad de los distintos métodos de detección es variable y depende mucho de la experiencia e idiosincrasia de cada laboratorio en particular y de cómo los adapte a su rutina de trabajo. En general, el examen microscópico directo de las muestras clínicas, mediante técnicas específicas de tinción (baciloscopia), es la técnica menos sensible pero la más rápida. Los cultivos son las más sensibles y más lentas, mientras que las técnicas moleculares basadas en amplificaciones genómicas tienen una sensibilidad menor que los cultivos pero son más rápidas.
La sensibilidad de la baciloscopia aumenta cuando las muestras se concentran. El cultivo aumenta la sensibilidad de la baciloscopia en un 30-60%, dependiendo del laboratorio, del tipo de muestra, y de los medios utilizados. La utilización de medios líquidos ha mejorado notablemente los resultados del cultivo por lo que en la actualidad, resultan imprescindibles. En el laboratorio del Hospital de Navarra se empezó en 1996, a utilizar un medio líquido (BACTEC 460TB, Becton Dickinson), junto con el medio sólido clásico de Löwenstein-Jensen (L-J). Posteriormente, se observó que el 18,6% de los aislamientos de M. tuberculosis se realizaron exclusivamente en el medio líquido y el tiempo medio de diagnóstico se acortó a 14,2 días, frente a 22,7 días que tardaban en aparecer las colonias en L-J.
Se han desarrollado una variedad de pruebas inmunológicas, que ponen de manifiesto la respuesta específica del huésped frente al agente infeccioso. La prueba de la tuberculina fue la primera empleada y ha sido la más utilizada, aunque no permite diferenciar la infección de la enfermedad. Se han descrito una variedad de pruebas in vitro (QuantiFERON-TB Gold and T-SPOT.TB) que miden la producción de interferon-gamma frente a antígenos específicos de micobacterias para ver la respuesta inmune y de las que se ha tratado en el anterior capítulo de este tratado9.
Organización de los laboratorios
La American Thoracic Society recomienda que el diagnóstico microbiológico de la tuberculosis se lleve a cabo, únicamente, en laboratorios con las adecuadas medidas de seguridad y cuyo volumen de trabajo y competencia en el área de las micobacterias esté asegurada10.
En 1993, ante el resurgir de la tuberculosis en Estados Unidos, los Centers for Disease Control and Prevention (CDC) de Atlanta, dieron las siguientes recomendaciones para los laboratorios de micobacterias:
Proporcionar a los laboratorios los medios para que puedan informar de los resultados de los exámenes directos en 24 horas.
Exigir que los laboratorios utilicen técnicas de tinción fluorescentes, más sensibles que las clásicas de Ziehl-Neelsen.
Utilizar medios líquidos en el cultivo primario, conjuntamente con el clásico de L-J.
Utilizar medios rápidos de identificación como sondas.
Realizar los estudios de sensibilidad en medio BACTEC o similares, que proporcionen resultados fiables y rápidos.
La finalidad de estas recomendaciones es proporcionar al clínico información rápida que le sirva para la toma de decisiones, de modo que: los exámenes directos no se demoren más de 24 h, el aislamiento e identificación de M. tuberculosis pueda realizarse en 10-14 días y los resultados del estudio de sensibilidad puedan estar disponibles en 15-30 días11. La consecución de estos objetivos es hoy factible en muchos laboratorios.
Recogida y transporte de muestras
Un concepto clásico en el diagnóstico de la tuberculosis es que la eliminación de bacilos, en muchos casos puede ser intermitente. Este concepto lo avala el hecho de que en ocasiones, entre varias muestras procesadas, sólamente una resulta positiva. Si bien es verdad que el estudio de varias muestras incrementa las posibilidades de diagnóstico, en la actualidad, con las mejoras introducidas y la necesidad de rentabilizar los procedimientos de diagnóstico, hay laboratorios que han reducido de 3 a 2 el número de muestras respiratorias recomendadas para un procesamiento rutinario12,13. La mejor muestra es la primera expectoración de la mañana. Las muestras una vez obtenidas, bien cerrado el frasco y bien identificadas, se transportan al laboratorio lo mas rápidamente posible. Si va a transcurrir más de una hora entre la expectoración y la recepción en el laboratorio deberá permanecer en frío (5-8ºC) y no a temperatura ambiente. Se desaconseja la práctica de recomendar al paciente que guarde 3 esputos y los lleve a la vez al laboratorio ya que es muy probable que se contaminen los cultivos y no sean útiles ni para descartar ni para confirmar el diagnóstico. Se deben extremar las medidas en la identificación de las muestras para que no se produzcan errores en el diagnóstico microbiológico.
Técnicas de tinción o baciloscopias
Tienen por objeto, poder visualizar en el microscopio la presencia o no de BAAR en las muestras. Actualmente siguen constituyendo la forma más rápida y económica para diagnosticar la TB. No obstante, dado que su sensibilidad (40-70% en muestras respiratorias) y su especificidad no son absolutas, es necesario realizar siempre cultivos para un diagnóstico de certeza. Existen multitud de variantes descritas de técnicas para las baciloscopias; incluso las hay para observadores daltónicos14. No obstante las dos más utilizadas son:
La tinción de Ziehl-Neelsen. Utiliza fucsina y fenol junto con el calentamiento de las preparaciones. Las micobacterias se tiñen de rojo, colorante que perdura pese a la posterior decoloración con una mezcla de alcohol-clorhídrico, sobre un fondo azul o verde, según se utilice como colorante de contraste el azul de metileno o el verde malaquita. Exige la observación con el objetivo de inmersión (1.000 aumentos), por lo que, debido a que en muchas preparaciones la presencia de bacilos puede ser escasa, es necesario un mínimo de 10 minutos de observación antes de valorar el examen como negativo.
Las tinciones con fluorocromos. Emplean como primer colorante la auramina-rodamina. Se tiñen en frío y, como en el caso anterior, tampoco se decolora con la mezcla de alcohol-clorhídrico. Al observarlos en un microscopio de fluorescencia, las micobacterias emiten una luz fluorescente. Esta luz emitida puede ser detectada rápidamente. Las preparaciones se observan con un objetivo de menor aumento, con lo que la superficie visualizada es mayor, lo cual hace que la tinción resulte más sensible y requiera menos tiempo de observación (1-2 minutos). Los inconvenientes de esta técnica son la dificultad del enfoque, que requiere un microscopio de fluorescencia, que algunas MNT de crecimiento rápido puede que no se tiñan, que puede a la larga dañar la vista del observador y, sobre todo, que es necesario personal con suficiente experiencia para visualizarlas. La decisión de utilizar una u otra en cada laboratorio está en función del número de baciloscopias que se realizan, la dotación de personal (su número y su experiencia) y del material disponible. En una jornada laboral un solo observador es prácticamente imposible que pueda ver más de 15-20 tinciones de Ziehl con garantías, mientras que podría ver 50-60 tinciones fluorescentes.
Cuantificación de las baciloscopias
El número de bacilos ácido-alcohol resistentes presentes en una muestra es un índice de la severidad de las lesiones y del grado de infectividad, por lo que la cuantificación de las muestras positivas es una práctica habitual pero hay que tener en cuenta si la tinción se ha realizado de la muestra directa o tras el proceso de HDC. En muchos laboratorios, como en el nuestro, no se realiza rutinariamente una baciloscopia a la muestra directamente sino que se hace siempre tras el proceso de decontaminación ya que se consigue mayor sensibilidad. Cuando se utiliza un fluorocromo3 y si únicamente se observan hasta 9 bacilos en toda la preparación, se informa como Dudosa. Entre 1-9 bacilos en 10 campos, se informa: Positiva 1+. Entre 1-9 bacilos por campo, se informa: Positiva 2+. Entre 10-90 bacilos por campo, se informa: Positiva 3+; y si se observan mas de 90 bacilos por campo, se informa: Positiva 4+.
Morfología de los BAAR
Habitualmente la morfología de los BAAR en las tinciones no permite diferenciar si se trata de una micobacteria u otra. No obstante un observador experimentado puede sospechar que no se trata de MTC cuando no aprecia su forma alargada y su dishomogeneidad en la coloración. Hay que tener en cuenta que la morfología del bacilo se altera durante el tratamiento tuberculostático. M. kansasii puede observarse como muy largo y con morfología acebrada lo que, aunque no definitivo, es muy característico. M avium se ve como bacilos muy cortos y con tinción uniforme. Mycobacterium fortuitum y Mycobacterium chelonae pueden ser muy polimorfos.
Causas de falsos positivos
Existen una serie de circunstancias técnicas que pueden dar lugar, en personal no experimentado, a errores al informar como BAAR acúmulos de pigmento debidos: a la presencia de restos de comida, aceites, cera o fibras en la muestra, a la reutilización de portas con fisuras en las que se acantona el colorante o a la no filtración de los reactivos. Otras veces se ven verdaderos BAAR pero puede tratarse de MNT en vez de MTC y ser saprofitos o colonizantes del paciente. También se dan casos de falsos positivos en los que los BAAR visualizados no pertenecen al paciente; así, si las cañerías del hospital están colonizadas con MNT y utilizamos esta agua para preparar la solución decontaminante luego podremos visualizarlas en la baciloscopia, o bien cuando transferimos BAAR de un porta a otro con el aceite contaminado utilizado con el objetivo de inmersión, en la microscopía óptica.
Causas de falsos negativos
Pueden encontrarse falsos negativos por realizar un frotis demasiado grueso o demasiado fino, por desprendimiento del frotis si calentamos excesivamente el porta. La falta de tiempo, la impaciencia o el cansancio del observador es la más importante. En el caso de tinción con fluorocromos la exposición excesiva a la luz UV pude hacer perder la capacidad de emitir fluorescencia. Hay que tener en cuenta que al hacer una extensión del material en el porta para la realización de la baciloscopia se utilizan 0,01 ml. Cuando en 1 ml de esputo hay un millón de BAAR la baciloscopia siempre suele ser positiva mientras que si hay 10.000 BAAR sólo es positiva en el 60%. La sensibilidad claramente disminuye cuando el nº de BAAR/ml de esputo es menor.
Persistencia de baciloscopias positivas en pacientes recibiendo tratamiento adecuado
A los 30 días de tratamiento el nº de BAAR visualizados en la tinción debe ser claramente inferior al de la baciloscopia inicial. Hasta el 50% permanecen con baciloscopias positivas al final del segundo mes y menos del 20% son positivos al final del tercer mes. Esta persistencia de baciloscopias positivas no debe hacer pensar en fracaso del tratamiento siempre que los cultivos sean negativos. Estos deben ser negativos a partir de los 30 días de tratamiento14.
Técnicas de cultivo
Los cultivos siguen siendo los métodos más sensibles para la detección de MTC en pacientes que no están recibiendo tuberculostáticos. Pueden detectar entre 10 y 100 bacilos/ml de esputo. A pesar del gran desarrollo y revolución que han supuesto, desde 1990, las técnicas moleculares, sobre todo las basadas en amplificación de ácidos nucleicos (AAN), ninguna de ellas, a fecha actual, ha conseguido en estudios clínicos ser tan sensible como los cultivos. Sin embargo, precisaremos de medios de cultivo específicos ya que con la inoculación directa de la muestra en los medios de cultivo habituales no crecen las micobacterias. El microbiólogo por tanto debe ser informado de si a una muestra respiratoria se le desea buscar una micobacteria o no.
La mayoría de las muestras respiratorias (excepto el líquido pleural obtenido por toracocentesis) contienen otros microorganismos que crecen rápidamente y que es preciso eliminar para que puedan desarrollarse las micobacterias. Para ello se realizan unos procedimientos de decontaminación de la muestra que a su vez la homogeneizan y la concentran (DHC). Estos procedimientos empleados para destruir las bacterias contaminantes deben preservar a las micobacterias, sin embargo, no es posible inactivar los contaminantes sin dañar a las micobacterias. Se acepta que el procedimiento es adecuado si se contaminan (y por tanto no son útiles para establecer el diagnóstico) un 5% de los cultivos realizados. Si el porcentaje de cultivos contaminados es inferior se asume que la decontaminación es excesiva y puede hacer que, si la cantidad de micobacterias presentes en la muestra es pequeña, éstas no sean viables y no crezcan en los cultivos.
Todo el procesamiento, debe llevarse a cabo en cabinas de seguridad biológica y por personal bien entrenado. Deben extremarse las medidas y los controles para evitar tanto la contaminación del personal, como la de otras muestras que se procesan al mismo tiempo15.
Existen diversos métodos descritos para decontaminar las muestras. Los más utilizados han sido los del laurilsulfato sódico (descrito por Tacquet y Tison) y el de la N-acetil-cisteína-hidróxido sódico (NALC-NaOH). En Europa se utilizaba más el del lauril y en USA el NALC-NaOH. El método del laurilsulfato sódico continúa manteniendo su actividad antibacteriana durante todo el periodo de incubación (y no sólo durante la inoculación) cuando se utilizan medios de cultivo líquidos o con base de agar, lo cual puede disminuir su rendimiento. Es muy buena opción cuando se utilizan medios en base de huevo ya que la lecitina inactiva la acción antibacteriana14. La incorporación rutinaria en los últimos 10 años de los medios líquidos ha hecho que la mayor parte de los laboratorios utilicemos el NALC-NaOH siguiendo las recomendaciones de los proveedores y de los CDC16.
Estos métodos de DHC son muy laboriosos y tediosos para los trabajadores durando varias horas y en ellos hay una serie de variables que pueden incidir en que luego podamos recuperar o no el MTC. La concentración del decontaminante, el tiempo que está en contacto con la muestra, las revoluciones y la temperatura de la centrífuga son las más importantes.
Una vez que las muestras se han decontaminado y concentrado, se procede a la realización de las extensiones para tinción y a la inoculación de los medios de cultivo. Cada vez es mayor el número de laboratorios que además hacen técnicas de amplificación de ácidos nucleicos (AAN), bien sea de forma inmediata (en el mismo día) o diferida (1-3 veces por semana).
La incubación de los medios de cultivo se realiza a 35-37ºC que es la temperatura óptima para el desarrollo de la mayoría de las micobacterias, con excepción de las muestras de piel que deben incubarse además a 28ºC ya que muchas micobacterias que producen infecciones de la piel y tejido subcutáneo se aíslan a esta temperatura. Los cultivos se mantienen un mínimo de 7 semanas antes de descartarlos como negativos.
Los medios de cultivo pueden ser líquidos o sólidos. Éstos a su vez se diferencian en que la base sea agar (las diferentes variantes del agar Middlebrook son las más utilizadas) o de huevo (el medio L-J y el medio Coletsos son los más utilizados).
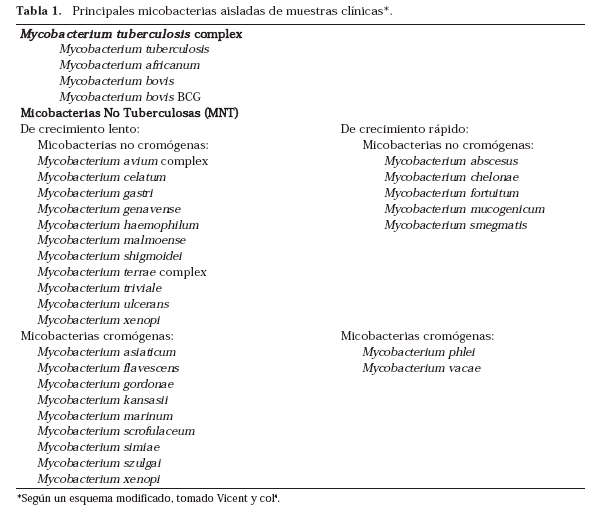
La detección del crecimiento depende del tipo de medios empleados. Con los medios sólidos en base de huevo la detección se realiza mediante la observación macroscópica 1 vez por semana. Si utilizamos medios en base de agar habrá que visualizarlos con el microscopio (100 aumentos) 2-3 veces por semana (Figs. 1 y 2). Los medios en base de agar cada vez son menos utilizados de forma rutinaria ya que consumen mucho tiempo de personal experimentado para poder leerlos y a que la fundamental ventaja que antaño tenían, que era la detección más precoz del crecimiento, es menor desde la incorporación de los medios líquidos.
Los medios líquidos son más sensibles que los sólidos y además detectan el crecimiento más precozmente. Antiguamente sólo se utilizaban en muestras muy concretas como líquidos cefalorraquídeos. A principios de la década de los 80 salió al mercado un sistema denominado BACTEC 460, hoy en día comercializado por Becton Dickinson (BD), que consiste en unos frascos con medio líquido Middlebrook 7H9 modificado (denominado Middlebrook 7H12) en que el ácido palmítico del medio está marcado con un isótopo del carbono. Los viales son leídos 2-3 veces por semana las dos primeras semanas y 1 vez por semana las cinco restantes en un aparato que pincha el frasco y detecta si hay o no crecimiento bacteriano en función de la radiactividad medida. Constituye el patrón de referencia con el que se comparan todos los medios antes de ser aprobados para su uso diagnóstico. Sólo algunos hospitales dotados de infraestructura para trabajar con material radiactivo lo introdujeron en su rutina de trabajo.
A partir de 1995 empiezan a salir al mercado medios líquidos que obvian el problema de la radiactividad y del tener que pinchar el frasco (con la teórica posibilidad de contaminar un frasco con bacterias del frasco anterior). Estos medios se introducen en unos aparatos que incuban los frascos y a su vez realizan una monitorización continua de la actividad metabólica que hay en su interior para ver si hay crecimiento o no. Los más utilizados son el MGIT (Mycobacterial Growth Indicator Tube, BD), el ESP Culture System II (Trek Diagnostic Systems) y el MB/BacT ALERT 3D (BioMerieux). MGIT utiliza un sensor de fluorescencia, ESP de presión y MB/BacT de colorimetría para detectar el crecimiento. Estos sistemas se han introducido en la mayor parte de los laboratorios y han constituido un gran avance.
Técnicas de identificación
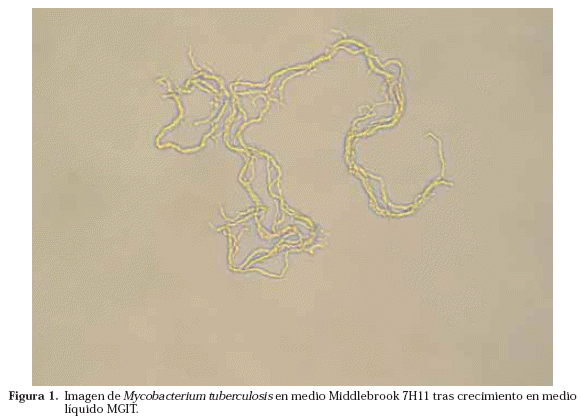
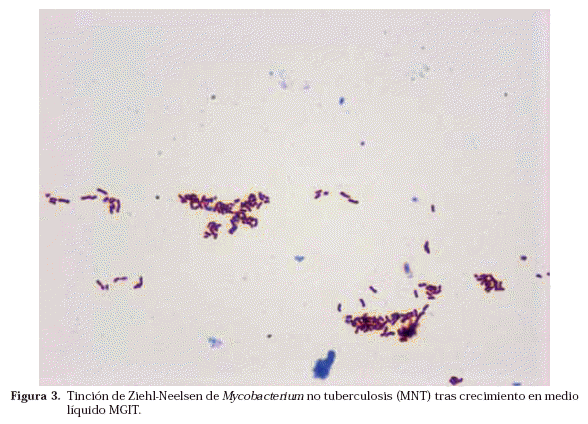
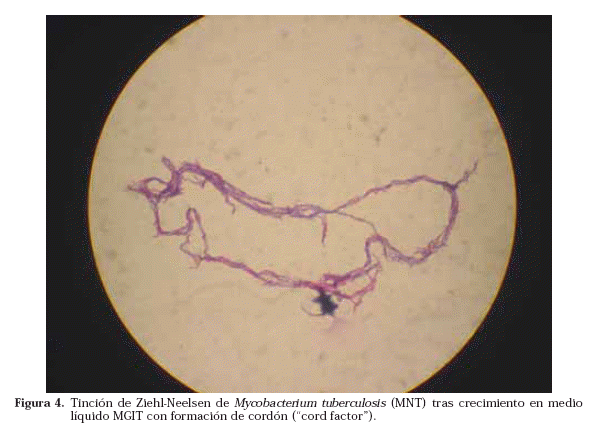
Una vez que se ha detectado crecimiento en alguno de los medios, se realiza la tinción de Ziehl-Neelsen para ver si lo que ha crecido son BAAR o no (Fig. 3). Caso de ser BAAR, su morfología nos puede orientar, en algunas ocasiones, hacia una especie u otra como en el caso de M. avium o de M. kansasii. Además la visualización de BAAR formando cordones (en medios líquidos) es muy característica del crecimiento de M. tuberculosis, aunque no patognomónica (Fig. 4)17.
Lo prioritario para el manejo de los pacientes es saber si se trata de MTC o de MNT. Para ello, hoy en día, la mayoría de los laboratorios realizan la identificación de MTC mediante sondas genéticas debido a su rapidez, precisión y simplicidad.
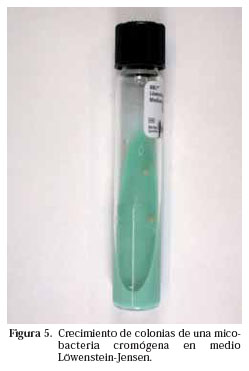
Tradicionalmente la identificación se realizaba en base a una serie de características fenotípicas. La velocidad de crecimiento en los cultivos y la temperatura óptima para ello, la morfología de la colonia, su pigmentación y su fotorreactividad orientaban preliminarmente hacia unas micobacterias u otras y posteriormente se realizaban pruebas bioquímicas a partir de la cepa crecida. Se considera que una micobacteria es de crecimiento lento cuando al hacer un subcultivo de una cepa, en medios de cultivo como el L-J, tardan más de 5-7 días en apreciarse las colonias macroscópicamente. Es cromógena cuando la colonia presenta pigmentación (Fig. 5), no cromógena cuando no la presenta y fotocromógena cuando pigmenta tras exposición a la luz. M. tuberculosis es una micobacteria no cromógena (Fig. 6) y de crecimiento lento. Sin embargo, cuando la baciloscopia es muy positiva, crece en los cultivos en medios líquidos en menos de una semana. Algunas de las pruebas bioquímicas utilizadas en la identificación de las micobacterias son el test de acumulación de niacina, la reducción de los nitratos, la hidrólisis del Tween, la catalasa, la captación de hierro, la pirazinamidasa, la detección de actividad ureasa, arylsulfatasa, etc.
Estos métodos clásicos, basados en pruebas bioquímicas, son muy lentos, laboriosos y peligrosos para el trabajador (además del riesgo de contagio, ya que se requiere trabajar con inóculos fuertes de micobacterias vivas, hay pruebas bioquímicas como la de la niacina, que suele ser positiva en M. tuberculosis en la que se formaban compuestos de cianuro).
Hoy en día, la mayoría de los laboratorios realizan la identificación de MTC mediante sondas genéticas. A principios de la década de los 90 se comercializaron sondas muy sensibles y específicas que permiten la identificación en 2 horas (AccuProbe, Gen-Probe Inc, San Diego, CA). Se trata de una prueba de hibridación en la que la sonda está compuesta por DNA de cadena única, complementario del RNA ribosomal de la micobacteria problema, y al que se ha unido un marcador quimioluminiscente. El RNA de la bacteria se libera previamente mediante su rotura con ultrasonidos. La sonda de DNA marcado combina con el RNA de la bacteria problema dando un híbrido estable DNA:RNA. La señal emitida por el híbrido marcado DNA:RNA se mide en un luminómetro, dando un resultado positivo o negativo, según alcance o no un umbral establecido. Tiene la limitación de que no permite la diferenciación entre las diferentes variantes del complejo MTC. La diferenciación entre ellas se hará mediante pruebas bioquímicas clásicas como la producción de nitritos y la sensibilidad a pirazinamida o mediante estudios moleculares. También existen y se utilizan sondas para la identificación del grupo Mycobacterium avium-intracellulare, M. avium, M. intracellulare, M. gordonae y M. kansasii que son las especies que se aíslan con más frecuencia.
Para la identificación de especies distintas de éstas, se recurre a distintos procedimientos. Los métodos clásicos de identificación están siendo sustituidos por los cromatográficos o genético-moleculares18.
Identificación cromatográfica mediante análisis de los ácidos micólicos
Los ácidos micólicos son ácidos grasos de alto peso molecular que forman parte de la pared celular de las micobacterias y otros géneros relacionados (Nocardia, Corynebacterium, Rhodococcus, Tsukamurella, etc.) El análisis de los ésteres de estos ácidos con diferentes técnicas cromatográficas permite realizar una identificación rápida, precisa y reproducible. La cromatografía líquida de alta eficiencia (HPLC) ha sido la que más se ha desarrollado y estandarizado. No obstante se requiere partir de abundante masa de bacterias para poder realizarlas lo cual implica que se necesita más tiempo hasta conseguir que el cultivo alcance la suficiente masa. Son técnicas complejas, que requieren una infraestructura costosa, un gran entrenamiento del personal por lo que son pocos los centros con dotación y experiencia suficiente para realizarlas.
Identificación mediante estudios genético-moleculares
El genoma completo de una cepa de MTC se ha secuenciado y contiene 4,4 millones de pares de bases, 4.000 genes que codifican proteínas y 50 que codifican RNA4. Las micobacterias contienen regiones bien conservadas de su DNA específicas de género y regiones hipervariables específicas de especie. Estas últimas son las que se utilizan como dianas, siendo las mejor estudiadas: el gen hsp65, que codifica la proteína de 65 KDa (heat shock), regiones genómicas de la subunidad ribosómica 16S, la región intergénica 16S-23S y los elementos de inserción. El grupo de técnicas que mayor interés y desarrollo tienen hoy en día se basan en la AAN de algunas de las secuencias citadas. Algunas de las más utilizadas son:
PCR-RFLP (PRA)
Consiste en realizar una PCR (reacción en cadena de la polimerasa), que es una de las técnicas de AAN más utilizada, del gen hsp65 seguida de un posterior análisis del polimorfismo de la longitud de los fragmentos de restricción (RFLP) tras la aplicación de 2 enzimas (BstEII y HaeIII) sobre lo amplificado. Fue descrita en 1992 y, en 1993 el asturiano Telenti19,20 la simplificó y modificó habiendo sido utilizada por multitud de centros (en la página web http://app.chuv.ch/prasite/index.html se pueden consultar los patrones de los polimorfismos encontrados). Consigue una identificación rápida (en una jornada laboral), precisa y económica de múltiples cepas. Tiene muchísima capacidad discriminatoria, siendo capaz de diferenciar unas 36 especies diferentes y las subespecies de los diferentes complejos micobacterianos, pero no discrimina las diferentes variantes del MTC. No necesita mucha masa de micobacterias y el equipamiento necesario no es excesivamente caro. Aunque bastante estandarizado tiene el inconveniente de que no es un método demasiado sencillo de realizar y que no está comercializado ni aprobado por la FDA.
Secuenciación del DNA
La secuenciación completa del 16S rDNA (1.500 pares de bases) o del hsp65 (400 pares de bases) está fuera del alcance de la mayoría de los laboratorios. La identificación dentro del 16S rDNA de secuencias específicas de especie, permite el diseño de protocolos sencillos de PCR seguidos de secuenciación de lo amplificado. Aunque la mayoría de los laboratorios no dispone de secuenciadores automatizados se pueden enviar los amplicones obtenidos a centros de referencia para que los secuencien. Esta técnica permite la identificación de muchas micobacterias en las que no es posible hacerlo por métodos fenotípicos y constituye el patrón de referencia de las identificaciones genotípicas. El rápido progreso de la tecnología con la aparición de nuevos secuenciadores capilares de microchips de secuenciación, es posible que facilite la difusión en el futuro de estas técnicas.
Hibridación en fase sólida
Se basa en la disponibilidad de sondas cortas de DNA específicas de especie, presentadas en formatos convencionales (placas con micropocillos, tiras de nitrocelulosa, etc.) o novedosos (chips o arrays de DNA). Se aplica el producto amplificado frente a diversas sondas en una sola prueba. Existen 2 productos comerciales con tiras de nitrocelulosa (INNO-LIPA y GenoType). Ambos se basan en la amplificación de una secuencia genética concreta (espacio intergénico 16S-23S y el 23S rDNA respectivamente) y su posterior hibridación. Los resultados se obtienen en 5-6 horas, son de fácil lectura e interpretación y además permiten detectar posibles coinfecciones. La sensibilidad y especificidad de ambas es muy buena. En la tira de INNO-LiPA hay 16 sondas que permiten la detección de MTC, M. kansasii (subtipos I, II y III y Mycobacterium gastri), Mycobacterium xenopi, M. gordonae, M. genavense, M. simiae, M. marinum / ulcerans, Mycobacterium celatum, M. avium complex, M. avium, M. intracellulare (grupo I y II), Mycobacterium scrofulaceum, Mycobacterium malmoense, M. haemophilum, M. chelonae complex (grupo I y II), M. fortuitum complex y Mycobacterium smegmatis. GenoType incluye diversas tiras con diferentes niveles de identificación. Una primera para 14 especies: MTC, M. kansasii, M. xenopi, Mycobacterium interjectum, Mycobacterium peregrinum, M. marinum / ulcerans, Mycobacterium abscessus, M. avium, M. intracellulare, Mycobacterium scrofulaceum, M. chelonae, M. gordonae, M. fortuitum y M. malmoense. Una segunda tira dispone de otras 16 identificaciones posibles y una tercera permite identificar las diferentes variantes de MTC.
Los chips (arrays) de DNA son los formatos de hibridación con más futuro (actualmente no esta generalizado su uso) por su flexibilidad tanto para la identificación como para la detección de mecanismos genéticos de resistencia a tuberculostáticos.
Cada método de aproximación taxonómica tiene su utilidad y ninguno por sí solo puede dar un resultado seguro al 100%. Las características fenotípicas clásicas son útiles inicialmente para decidir la cronología de los medios de identificación a utilizar.
Dentro del complejo M. tuberculosis, la diferenciación de las distintas variantes puede realizarse mediante pruebas bioquímicas clásicas como la producción de nitritos y la sensibilidad a pirazinamida o mediante estudios moleculares, como RFLP con IS6110 u otros.
Siempre hay que intentar la identificación de toda micobacteria, aislada de muestras clínicas, a nivel de especie aunque no se sepa su valor como patógeno.
Pruebas directas de detección de ácidos nucleicos de MTC EN muestras clínicas
Tienen por objeto detectar e identificar sobre la muestra (y no sobre los cultivos) la presencia de micobacterias del complejo tuberculosis. En teoría serían capaces de detectar la presencia de una sola micobacteria en la muestra, pero en la práctica ningún estudio ha demostrado que sean tan sensibles como los medios de cultivo líquido. Sí que son más sensibles que las baciloscopias y más rápidos que los cultivos. Habitualmente no se realizan sobre la muestra directa sino sobre el producto obtenido tras el proceso de DHC. En 24-48 horas desde la entrada de la muestra al laboratorio se puede detectar si hay o no MTC en la muestra. Existen varios en el mercado21-23, que a continuación se citan.
Amplified Mycobacterium tuberculosis direct (AMTD)
Fue el primero aprobado por la FDA (se comercializó por Gen Probe en 1993) y es el más ampliamente evaluado y utilizado. Es una amplificación mediada-transcripción realizada a temperatura constante (42ºC). La diana es una secuencia específica del 16S rRNA de la cual hay unas 4000 copias por cada bacteria de MTC, lo cual es una ventaja ya que en el resto de las técnicas la secuencia diana es menos frecuente. Un porcentaje importante de los laboratorios lo han incorporado a su rutina diagnóstica, si bien el coste en reactivos de cada determinación (a modo orientativo unos 36 vs 8 que puede costar un medio líquido y 1 que cuesta un tubo de L-J) ha dificultado su implantación. Los resultados se obtienen en 3-5 horas.
AMPLICOR M. tuberculosis PCR tests
Basado en una PCR es similar al anterior, pero ligeramente menos sensible y específico y además la técnica es algo más larga (6-8 horas). Ha sido comercializada por Roche y también aprobada por la FDA.
Otras menos utilizadas son el SDA (Strand Displacement Amplification, comercializado por BD como BDProbeTec ET) y la LCR (Ligase Chaín Reaction, comercializada por Abbott).
PCR en tiempo real
Como norma general, esta novedosa tecnología permite resultados más rápidos, específicos y es más facil de realizar que las técnicas de AAN anteriormente citadas. En el momento actual y en lo que se refiere a su aplicación al diagnóstico de MTC todavía no hay suficientes evaluaciones concluyentes sobre su utilidad pero es de esperar que aparezcan en un futuro inmediato. El hecho de que estas técnicas requieran inicialmente la extracción de los ácidos nucleicos, que en el caso de las micobacterias es especialmente difícil por las características de su pared celular, y que su manejo en el laboratorio presenta la dificultad del riesgo de contagio, pueden haber ralentizado el desarrollo de la PCR en tiempo real.
Técnicas moleculares para tipificación de MTC. Interés epidemiológico
Durante mucho tiempo, para el estudio de brotes se han utilizado marcadores fenotípicos que no tenían suficiente capacidad para discriminar las posibles diferencias que pudieran existir entre una cepa y otra. El desarrollo, a partir de 1991, de diversos métodos moleculares de tipificación ha permitido comparar las huellas genéticas de los distintos aislamientos y, por tanto, establecer diferencias entre diversas cepas. Al diferenciar las cepas aisladas, ha sido posible obtener nueva información sobre la epidemiología de la enfermedad lo que ha cambiado muchos conceptos que se habían aceptado durante décadas. Así, se cree actualmente que la transmisión reciente de MTC puede ser un factor más importante de lo que se suponía, oscilando, en nuestro entorno, entre el 28% de Segovia y el 58% de Gran Canaria. Se ha podido demostrar la conexión epidemiológica en brotes de tuberculosis multirresistente en España. Hay datos de epidemiología molecular, referentes a la cepa Beijinj que permite suponer que hay cepas más virulentas o bien más transmisibles que otras, cosa que antes era impensable24. También ha permitido documentar más contaminaciones cruzadas en los laboratorios de las que se sospechaban. Las técnicas más conocidas se recogen a continuación.
IS6110 RFLP
La IS6110 es una secuencia de inserción de 1.335 pares de bases que se repite entre 0 y 25 veces en diferentes lugares del cromosoma de cada cepa de MTC. El análisis de los polimorfismos de la longitud de los fragmentos de restricción (RFLP) utilizando como secuencia diana la IS6110 es el método más utilizado y el que se considera de referencia. Se realiza siguiendo un protocolo estandarizado y es una técnica muy reproducible y útil para distinguir entre los aislamientos relacionados epidemiológicamente de los no relacionados, de modo que se ha utilizado como indicador de transmisión reciente. Presenta algunas limitaciones, ya que no puede utilizarse si la micobacteria tiene menos de 6 copias de este fragmento en su cromosoma, en cuyo caso es preciso la utilización de otras técnicas. Además, requiere grandes cantidades de ADN (lo que implica la realización de subcultivos que precisan varias semanas), es técnicamente complejo y caro.
Spoligotyping
Es un método muy utilizado por su relativa simplicidad, rapidez y bajo coste, generalmente como complemento del anterior. Estudia la presencia o ausencia de 43 fragmentos de ADN, llamados espaciadores. Requiere menos ADN que el anterior y, al expresarse en forma de positivo o negativo (de cada espaciador), puede analizarse en un formato digital. Existe una base de datos internacional con más de 11.000 patrones (espoligotipos) de aislamientos obtenidos en más de 90 países11. Es muy útil para la confirmación de la identificación de las diferentes variantes dentro del MTC. Sin embargo, este método no reemplaza por completo al RFLP por su menor poder discriminante.
Número variable de repeticiones en tándem (variable number tándem repeat, VNTR)
El método se basa en la detección del número de veces que se repiten de forma adyacente varias secuencias dentro del genoma de la micobacteria. El más utilizado es el Mycobacterial Interspersed Repetitive Unit-variable-number tándem repeat analysis (MIRU-VNTR), que determina las unidades repetitivas en 12 loci tras una PCR. En cada uno de los 12 loci hay de 2 a 8 alelos, lo que da lugar a unos 20 millones de posibles combinaciones de alelos. El MIRU-VNTR es más discriminante que el spoligotyping y, similar al basado en RFLP-IS61101, se puede automatizar y técnicamente es más simple, por lo que es previsible que se imponga como método de referencia. Existe una base de datos internacional accesible por Internet para poder comparar los hallazgos.
Métodos que utilizan el genoma completo
Están prácticamente fuera del alcance de casi todos los laboratorios de investigación y son impensables, hoy en día, para los laboratorios clínicos. Inicialmente se somete el DNA completo a enzimas de restricción y luego se hace una AAN selectiva de las zonas parcialmente complementarias o/y contiguas a la zona donde actúan los enzimas de restricción. Es capaz de detectar mínimos cambios en las bases de los fragmentos amplificados.
Estudios de sensibilidad
Su finalidad es poder predecir, si la cepa aislada responderá al tratamiento tuberculostático convencional o bien si presenta algún mecanismo de resistencia que indican que una determinada droga no va a ser efectiva.
Antaño en lugares donde los índices de resistencia eran bajos, menores del 4% para la Isoniacida (INH) se desaconsejaba la realización de los estudios de sensibilidad. Una metodología inadecuada, en no pocas ocasiones, daba lugar a que se recomendase el uso de drogas más tóxicas y menos eficaces con el consiguiente perjuicio para el paciente y para la salud pública.
El incremento de cepas resistentes registrado a partir de los años 90, hace que los estudios de sensibilidad sean recomendables hoy en día frente a todas las cepas de M. tuberculosis que se aíslan inicialmente a cualquier paciente. Aunque cada vez la tecnología es más sencilla, el hecho de que cada laboratorio, en los países desarrollados, no aísle suficientes cepas para coger la experiencia necesaria ha de hacer que se plantee su realización o la derivación del estudio, hacia centros con más experiencia o/y mejor tecnología4.
El problema de la resistencia en la tuberculosis está ligada al hecho de la elevada concentración bacteriana presente en las lesiones. En la tuberculosis pulmonar, la mayor concentración bacteriana se da en las lesiones cavitadas que pueden contener hasta 107 ó 109 organismos, mientras que la concentración presente en los focos caseosos, no suele exceder los 102 ó 104. Desde 1949, era un hecho conocido, que la aparición de resistencias, se daba con mucha más frecuencia en el tratamiento de lesiones cavitadas25,26.
David27 en 1970, demostró la probabilidad de distribución de mutantes resistentes, mediante un test de fluctuación, con el que puso en evidencia, que M. tuberculosis adquiría espontáneamente una mutación que le hacía resistente a isoniacida, estreptomicina, etambutol y rifampicina con una frecuencia media de 3x10-8, 3x10-8, 1x10-7, y 2x10-10 respectivamente. De este modo, la probabilidad teórica de adquirir una mutación que confiera resistencia a dos drogas es menor a 10-15. De ahí que la base del tratamiento de la tuberculosis sea el empleo de varias drogas efectivas simultáneamente.
Los criterios para definir la resistencia a una droga en M. tuberculosis se han tomado sobre una base empírica, teniendo en cuenta que a partir de una cierta proporción en el número de mutantes resistentes, la droga no va a ser efectiva. En base a estudios clínicos y bacteriológicos, se acepta como proporción crítica la del 1%. Si se produce un crecimiento por encima de esta proporción, se valora la cepa como resistente.
Para estudiar la sensibilidad de las micobacterias, se emplea generalmente el método de las proporciones, es decir se compara el número de colonias que crecen en un medio con droga y el número de colonias que crecen en el mismo medio libre de droga. Si el número de colonias en el primero no llega al 1% del segundo, la droga se considera efectiva. Los medios que contienen huevo, tipo L-J, no son adecuados para el antibiograma. En un intento por conseguir una uniformidad en los estudios de sensibilidad el CDC y el NCCLS (National Committee for Clinical Laboratory Standards) recomiendan la utilización del medio agar de Middlebrook 7H10 o 7H1128, 29.
La realización del antibiograma en medios líquidos cada vez es más frecuente por su relativa sencillez, reproducibilidad y rapidez, ya que los resultados pueden obtenerse en 5-10 días, frente a 3 semanas que requieren los medios sólidos. En el método radiométrico BACTEC, que equivale a una versión modificada del de las proporciones, la resistencia se determina comparando el crecimiento que se detecta en un vial control sin droga y en otro igual que contiene la droga que se quiere testar. El vial que contiene droga se inocula con una concentración de bacterias 100 veces superior a la del control sin droga. Los viales se leen diariamente, registrándose su índice de crecimiento. Se determina la diferencia entre los índices que se registran en días consecutivos. Cuando el índice de crecimiento en el vial control alcanza un valor determinado, se interpretan los resultados del siguiente modo: si la diferencia entre los índices de crecimiento, es menor en el vial que contiene droga que en el control, la población es sensible; si mayor, es resistente30. Se debe hacer constar cuál es la concentración o concentraciones a la/s que se ha testado el fármaco. Es posible que cepas resistentes a INH a concentraciones bajas y sensibles a concentraciones altas respondan a INH y no se deba suspender su prescripción. Los estudios se realizan frente a los 4 tuberculostáticos principales: isoniacida, estreptomicina, rifampicina y etambutol. La pirazinamida solamente es activa en medio ácido por lo que es preciso bajar el pH del medio, con lo que muchas veces, queda comprometido el crecimiento bacteriano. El sistema BACTEC ha resuelto este problema utilizando una modificación del medio de cultivo para el estudio de esta droga.
Aunque con menos experiencia que con el BACTEC radiométrico también se han desarrollado y adaptado los antibiogramas a otros medios líquidos como el ESP, el MGIT y el MB/BacT ALERT 3D.
Los estudios de sensibilidad frente a otras drogas resultan problemáticos y no están bien estandarizados31. La utilidad de los antibiogramas de MNT para guiar el tratamiento de las micobacteriosis no está clara. Puede ser útil testar M. avium frente a macrólidos y M. kansasii frente a rifampicina
Cuando el paciente, por datos epidemiológicos conocidos, es probable que albergue una cepa de MTC resistente y la baciloscopia sea claramente positiva, puede plantearse realizar el antibiograma directamente de la muestra. Posteriormente se debe confirmar el antibiograma con la técnica estandarizada a partir del cultivo31.
Se han descrito diversos genes cuya mutación se asocia con la resistencia a drogas. Así, la resistencia a rifampicina en más del 96% de los casos se debe a una mutación en el gen rpo. En cuanto a la isoniacida, se han descrito diversos genes katG, inhA, ahpC, kasA, que en su conjunto son responsables de la resistencia a esta droga aproximadamente en un 90% de los casos; la resistencia a etambutol en un 47-65% de los casos, se debe a una mutación en el gen embB y la resistencia a estreptomicina en un 70% se debe a la mutación del gen rpsL. Se han desarrollado múltiples métodos moleculares para detectar la posible existencia de estos mecanismos de resistencia con el objeto de no tener que esperar los 5-10 días que puede tardar el antibiograma a partir de que la cepa este aislada, pero todavía no son lo suficientemente simples como para incorporarlos a la rutina diagnóstica32.
En el Hospital de Navarra, el promedio del porcentaje de cepas resistentes a isoniacida en 5 años (1998-2002) es del 7%, que es un índice relativamente alto, sin embargo, la incidencia de cepas multirresistentes es decir, resistentes al menos a isoniacida mas rifampicina, en ese mismo período de tiempo, no llegó al 2%.
Consideraciones finales
1. La baciloscopia sigue siendo el método más rápido y barato para diagnosticar la TB en nuestro medio.
2. El diagnóstico de certeza se establece con el aislamiento en cultivo e identificación de M. tuberculosis a partir de muestras clínicas.
3. La utilización sistemática de medios de cultivo líquidos ha aumentado su sensibilidad y rapidez. También ha hecho que se aíslen más micobacterias no tuberculosas cuya significación clínica es, en muchas ocasiones, difícil de establecer.
4. Los estudios de sensibilidad deben realizarse a todo primer aislamiento de M tuberculosis en centros acreditados y con suficiente experiencia.
5. El gran desarrollo de la tecnología molecular de ácidos nucleicos ha permitido en muchos laboratorios que la detección e identificación de M tuberculosis y otras micobacterias sea más rápida y precisa.
6. Las técnicas moleculares están permitiendo conocer mejor los factores de virulencia del microorganismo, sus mecanismos de resistencia y aspectos epidemiológicos de las micobacteriosis, que redundarán en un mejor manejo de la enfermedad y de los pacientes.
Bibliografia
1. Koch R.1884. Die Aetiologie der Tuberkulose. Mittheilungen aus dem Kaiserlichen Gesundheitsamte; 2: 1-88. [ Links ]
2. Collins CH, Grange JM, Yates MD. Identification of species and variants of tubercle bacilli. En: Organization and practice in tuberculosis bacteriology. Londres, Boston: Butterworths and Co Ltd, 1985; 59-66.
3. Pfyffer GE, Brown-Elliott BA, Wallace RJ. 2003. Mycobacterium: General characteristics, isolation, and staining procedures. En: Murray P.R., editor. Manual of Clinical Microbiology, 8th ed. American Society for Microbiology, Washington, DC: 532-559.
4. Vincent V, Brown-Elliott BA, Jost KC, Wallace RJ. 2003. Mycobacterium: Phenotypic and Genotypic Identification. En: Murray P.R., editor. Manual of Clinical Microbiology, 8th ed. American Society for Microbiology, Washington, DC: 560-583.
5. Hirschman SZ. Mycobacterial infection in the AIDS era: implications for infection control and employee health. Mt Sinai J Med 1990; 57: 207-208.
6. Ellner J, Goldberger M, Parenti D. Mycobacterium avium infection and AIDS: a therapeutic dilema in rapid evolution. J Infect Dis 1991; 163: 1326-1335.
7. Ocaña I, Ruiz I, Vidal R, Martín N, De Gracia J, Ribera E et al. 1988. Micobacteriosis y tuberculosis en pacientes con infección por VIH. Enf Infec Microbiol Clin 1988; 6: 291-297.
8. García JM, Palacios JJ, Sánchez AA. Infecciones respiratorias por micobacterias ambientales. Arch Bronconeumol 2005; 41: 206-219.
9. Arend SM, Thijsen SF, Leyten EM, Bouwman JJ, Franken WP et al. Comparison of two interferon-gamma assays and tuberculin skin test for tracing tuberculosis contacts. Am J Respir Crit Care Med 2007; 175: 618-627.
10. American Thoracic Society. Diagnostic standards and classification of tuberculosis. Am Rev Respir Dis 1990; 142: 725-735.
11. Tenover FC, Crawford JT, Huebner RE, Geiter LJ, Horsburgh CR Jr et al. The resurgence of tuberculosis: is your laboratory ready? J Clin Microbiol 1993; 31: 767-770.
12. Nelson SM, Deike MA, Cartwrigt CP. Value of examining multiple specimens in the diagnosis of pulmonary tuberculosis. J Clin Microbiol 1998; 36: 467-469.
13. Dorronsoro I, Martín C, Cabodevilla B, Ojer M, Ruz A. Influencia del número de muestras estudiadas en el diagnóstico de la tuberculosis. Enferm Infecc Microbiol Clin 2000; 18: 215-218.
14. Ortega A, March J. Manual de Micobacteriología. 2006 (Distribuido por BioMerieux España, www.biomerieux.es).
15. Aznar J, Safi H, Palomares JC. Falso brote de tuberculosis por contaminación en las muestras en un laboratorio de micobacteriología: confirmación por técnicas moleculares. Enferm Infecc Microbiol Clin 1997; 15: 144-146.
16. Kent PT, Kubica GP. Public health mycobacteriology, a guide for the level III laboratory. Centers for Disease Control, Division of Laboratory Training and Consultation. Atlanta, Ga.
17. Tu HZ, Chang SH, Huaug TS, Huaug WK, Liu YC, Lee SS. Microscopic morphology in smears prepared from MGIT broth medium for rapid presumptive identification of Mycobacterium tuberculosis complex, Mycobacterium avium complex and Mycobacterium kansasii. Ann Clin Lab Sci 2003; 33: 179-186.
18. Alcaide F. Nuevos métodos de identificación de micobacterias Enferm Infecc Microbiol Clin 2006; 24: 53-57.
19. Telenti A, Marchesi F, Balz M, Bally F, Böttger EC, Bodmer T. Rapid identification of mycobacteria to the species level by polymerase chain reaction and restriction enzyme analysis. J Clin Microbiol 1993; 31: 175-178.
20. Devallois A, Goh KS, Rastogi N. Rapid identification of mycobacteria to species level by PCR-Restriction Fragment Length Polimorphism Analysis of the hsp65 gene and proposition of an algorithm to differentiate 34 mycobacterial species. J Clin Microbiol 1997; 35: 2969-2973.
21. Carpentier E, Drouillard B, Dailloux M, Moinard D, Vallee E et al. Diagnosis of tuberculosis by Amplicor Mycobacterium tuberculosis test: a multicenter study. J Clin Microbiol 1995; 33: 3106-3110.
22. Clarridge JE 3rd, Shawar RM, Shinnick TM, Plikaytis BB. Large-scale use of polymerase chain reaction for detection of Mycobacterium tuberculosis in a routine mycobacteriology laboratory. J Clin Microbiol 1993; 31: 2049-2056.
23. Piersimoni C, Scarparo C, Piccoli P, Rigon A, Ruggiero G et al. Performance assessment of two commercial amplification assays for direct detection of Mycobacterium tuberculosis complex from respiratory and extrapulmonary specimens. J Clin Microbiol 2002; 40: 4138-4142.
24. García-Pachón E. Epidemiología molecular de la tuberculosis: principales hallazgos y su aplicación en España. Arch Bronconeumol 2005; 41: 618-624.
25. Canetti G. Present aspects of bacterial resistance in tuberculosis. Am Rev Respir Dis 1965; 92: 687-702.
26. Howard WL, Maresh F, Mueller EE, Yanitelli SA, Woodruff GF. The role of pulmonary vacitation in the development of bacterial resistance to streptomycin. Am Rev Tuberc 1949; 59: 391-401.
27. David HL. Probability distribution of drug-resistant mutants in unselected populations of Mycobacterium tuberculosis. Appl Microbiol 1970; 20: 810-814.
28. Inderlied CB, Pfyffer GE. 2003. Susceptibility test methods: Mycobacteria. En: Murray P.R., editor. Manual of Clinical Microbiology, 8th ed. American Society for Microbiology, Washington DC: 1149-1177.
29. National Committee for Clinical Laboratory Standards. Susceptibility testing of mycobacteria, nocardiae, and other aerobic actinomycetes. Approved Standard. Wayne, PA. NCCLS. 2003. Document nº M24-A.
30. Siddiqi SH. 1995. BACTEC 460TB SYSTEM Product and Procedure Manual. MA-0029 Becton Dickinson and Company.
31. Griffith DE, Aksamit T, Brown-Elliott BA, Catanzaro A, Daley C et al. An official ATS/IDSA statement: diagnosis, treatment, and prevention of nontuberculous mycobacterial diseases. Am J Respir Crit Care Med 2007; 175: 367-416.
32. Alcaide F, Telenti A. Molecular techniques in the diagnosis of drug-resistant tuberculosis. Ann Acad Med Singapore 1997; 26: 647-650.
 Dirección para correspondencia:
Dirección para correspondencia:
Luis Torroba Álvarez
Laboratorio de Microbiología
Hospital Virgen del Camino
C/ Irunlarrea, 4
31008 Pamplona
E mail: ltorroba@gmail.com