Meu SciELO
Serviços Personalizados
Journal
Artigo
Indicadores
-
 Citado por SciELO
Citado por SciELO -
 Acessos
Acessos
Links relacionados
-
 Citado por Google
Citado por Google -
 Similares em
SciELO
Similares em
SciELO -
 Similares em Google
Similares em Google
Compartilhar
Revista de Osteoporosis y Metabolismo Mineral
versão On-line ISSN 2173-2345versão impressa ISSN 1889-836X
Rev Osteoporos Metab Miner vol.8 no.1 Madrid Jan./Mar. 2016
Estudio del patrón de expresión de microRNAs en el hueso osteoporótico
Study of miRNAs expression patterns in osteoporotic bone
García-Giralt N.1, De-Ugarte L.1, Yoskovitz G.1, Güerri R.1,2, Grinberg D.3, Nogués X.1,2, Mellibovsky L.1,2, Balcells S.3 y Díez-Pérez S.1,2
1 IMIM (Instituto Hospital del Mar de Investigaciones Médicas) - Red Temática de Investigación Cooperativa en Envejecimiento y Fragilidad (RETICEF) - ISCIII - Barcelona (España)
2 Departamento de Medicina Interna - Parque de Salud Mar - Universidad Autónoma de Barcelona - Barcelona (España)
3 Departamento de Genética - Universidad de Barcelona - IBUB - Centro de Investigación Biomédica en Red de Enfermedades Raras (CIBERER) - ISCIII - Barcelona (España)
Trabajo becado por la FEIOMM para asistir al 36o Congreso de la ASBMR (Houston, 2014).
Este trabajo ha sido financiado por la Red Temática de Investigación Cooperativa en Envejecimiento y Fragilidad (RETICEF; RD12/0043/0022), y la ayuda FIS PI13/00116 (Instituto de Salud Carlos III, Ministerio de Ciencia e Innovación) y los fondos FEDER.
Dirección para correspondencia
RESUMEN
Objetivos: Identificar microRNAs (miRNAs) diferencialmente expresados en muestras óseas con fractura osteoporótica respecto a huesos sanos.
Material y métodos: Se extrajo RNA total a partir de hueso trabecular fresco del cuello femoral de mujeres sometidas a reemplazo de cadera, ya sea debido a fractura osteoporótica (n=6) o por artrosis en ausencia de osteoporosis (según la DMO) (n=6). Las muestras se hibridaron en un array de miRNAs y se realizaron diagramas de PCA y de mapa de calor. Para la comparación de los niveles de expresión, se fijó como significativo un umbral de cambio de >1,5 veces y un valor p<0,05 en la t de Student (corregido para múltiples pruebas).
Resultados: Tanto los análisis de PCA como el mapa de calor mostraron una agrupación de las muestras según si eran de fractura o no. Se detectaron 790 miRNAs en las muestras de hueso, 82 de los cuales estaban alterados en las muestras osteoporóticas. Tras la validación en otro panel de 6 muestras osteoporóticas y 6 no osteoporóticas mediante PCR a tiempo real de los miRNAs más significativos, y para los que existía un ensayo disponible, se confirmaron los miRNAs miR-320a y miR-22-3p.
Estos dos miRNAs se detectaron en cultivos de osteoblastos primarios, aunque no mantenían el mismo patrón de expresión que en las muestras de hueso total.
Conclusiones: Hemos demostrado que existen diferencias en la expresión de miRNAs en muestras con fractura osteoporótica, lo que abre nuevas perspectivas para la investigación y diseño de nuevas terapias.
Palabras clave: microRNA, fractura ósea, osteoporosis, osteoblastos, hueso.
SUMMARY
Objectives: To identify microRNAs (miRNAs) differentially expressed in bone samples with osteoporotic fracture compared with healthy bones.
Methods: Total RNA was extracted from fresh trabecular bone of the femoral neck of women undergoing hip replacement surgery, either because to osteoporotic fracture (n=6) or in the absence of osteoarthritis osteoporosis (based on BMD) (n=6). The samples were hybridized on an array of miRNAs and PCA diagrams and heat map were made. To compare expression levels, >1.5 times and a value p<0.05 Student's T test (corrected for multiple testing) was set as a threshold of significant change.
Results: Both PCA analysis and the heat map showed a samples grouping whether there was fracture or not. 790 were detected miRNAs in bone samples, 82 of which were altered in the osteoporotic samples. After validation in another panel of 6 samples 6 osteoporotic and non-osteoporotic by PCR real time of the most significant miRNAs, and for which there was a test available, the miRNAs, miR-320a and miR-22-3p were confirmed. These two miRNAs were detected in cultures of primary osteoblasts, although they did not maintain the same pattern of expression in total bone samples.
Conclusions: We have shown that there are differences in the expression of miRNAs in samples with osteoporotic fracture. This opens prospects for research and design of new therapies.
Key words: microRNA, bone fracture, osteoporosis, osteoblasts, bone.
Introducción
Los microRNAs (miRNAs) han sido relacionados con una variedad de procesos, tales como la proliferación, la diferenciación y la apoptosis celular. Una desregulación de cualquiera de estos procesos podría conducir a procesos patológicos, algunos de ellos tan graves como el cáncer [1].
Los microRNAs son pequeños RNAs (18-24nt), no codificantes, que regulan negativamente la expresión génica mediante la unión a las secuencias 3'-UTRs de mRNAs diana. En el tejido óseo, los miRNAs han sido descritos como factores clave para la regulación de la formación, remodelación, y la homeostasis del hueso [2,3]. Además, varios estudios han demostrado que los miRNAs están involucrados en el control de la diferenciación y de la función de las células óseas [4], y por ello la identificación de dichos miRNAs podría ser una herramienta para desarrollar terapias para promover la formación ósea o inhibir la resorción y así actuar sobre las enfermedades óseas.
En el campo de la osteoporosis hay muy pocos estudios sobre la implicación de los miRNAs en su fisiopatología. Li, et al. [5] describieron una mutación en el pre-miR-2861 que bloqueaba la expresión del miR-2861, causando osteoporosis primaria en dos adolescentes emparentados. En otro estudio, tres polimorfismos en las secuencias diana para el miR-146a y el miR-146b en el gen FGF2 se asociaron genéticamente con la densidad mineral ósea (DMO) del cuello femoral [6]. Se han realizado también varios estudios intentando identificar miRNAs con un patrón de expresión alterado asociado a la fractura osteoporótica con el fin de encontrar biomarcadores de la patología [7-9]. Cabe destacar que todos estos estudios detectaron diferentes miRNAs demostrando una vez más la complejidad de la osteoporosis de la fractura.
De todas maneras, el tipo de muestra, las condiciones de recogida y el proceso de manipulación, así como las características de la población de estudio pueden influenciar al resultado final. De hecho, se ha encontrado una variedad de perfiles de expresión de miRNAs en función del tipo de célula estudiada dentro del linaje osteoblástico (proliferación, diferenciación, mineralización), de si está sometida a algún tratamiento (hormonas, citoquinas) o de la especie de procedencia [10,11]. De esta manera, se hace difícil hacer comparaciones entre estudios, aunque cualquier aportación en el campo es un paso más para entender la fisiopatología de la fractura.
El objetivo de este estudio fue identificar miRNAs con expresión alterada en el hueso osteoporótico, utilizando una metodología experimental lo más cercana posible a las condiciones fisiológicas. Para este propósito se analizó el hueso trabecular obtenido de pacientes con una fractura reciente por osteoporosis en comparación con muestras de hueso no osteoporóticas. Se realizó un estudio de hibridación de microarray a partir de tejido óseo total fresco para detectar todos los miRNAs expresados en estas muestras.
Material y métodos
Obtención de las muestras óseas
El hueso trabecular del cuello femoral se obtuvo de mujeres post-menopáusicas sometidas a reemplazo de cadera, ya sea por fractura osteoporótica (OP) (n=6) o por artrosis (n=6). En las muestras de artrosis se determinó la ausencia de osteoporosis mediante la medición de la DMO y se consideraron como el grupo control. Para la validación de los resultados del array se obtuvieron 6 muestras adicionales de hueso trabecular del cuello femoral con fractura y 6 muestras sin osteoporosis. De estas últimas muestras también se obtuvieron osteoblastos primarios (HOb). Ningún paciente tenía antecedentes de enfermedad metabólica o endocrina, insuficiencia renal crónica, enfermedad hepática crónica, cáncer, enfermedad ósea de Paget, síndrome de mala absorción, tratamientos de terapia de reemplazo hormonal, agentes antirresortivos o anabólicos o esteroides orales, fármacos anti-epilépticos, litio, heparina o warfarina. Se obtuvieron los consentimientos informados por escrito de acuerdo con los reglamentos del Comité Ético de Investigación Clínica del Parque de Salud MAR, que aprobó el estudio.
Cultivo de osteoblastos primarios
Para el cultivo de osteoblastos procedentes de las muestras óseas de la fase de validación, se obtuvieron fragmentos pequeños de hueso trabecular y se colocaron en placas de cultivo de 140 mm. Se incubaron con medio de cultivo DMEM (Dulbecco's Modified Eaglle Medium) suplementado con FBS (Fetal Bovine Serum) al 10%, penicilina/streptomicina al 1%, fungisona al 0,4% y 100 μg/ml de ácido ascórbico. Las placas se tripsinizaron aproximadamente al cabo de tres semanas justo antes de llegar a la confluencia para la extracción de RNA.
Extracción de RNA
Para la extracción de RNA de tejido óseo total, las muestras frescas de hueso trabecular se cortaron en pequeños fragmentos, se lavaron tres veces en solución tamponada con fosfato (PBS), y se almacenaron a -80oC hasta su uso.
La extracción de RNA tanto de hueso total como de los HOb se realizó utilizando el kit miRNeasy Mini (Qiagen) siguiendo las instrucciones del fabricante. En el caso de los osteoblastos primarios también se utilizó el Rneasy MinElute Cleanup (Qiagen) para obtener la fracción enriquecida en miRNAs. La concentración del RNA purificado se analizó en un espectrofotómetro (Nanodrop, Thermo Fisher Scientific Inc).
Microarray de microRNAs de las muestras de hueso total y análisis de datos
El microarray y el análisis de datos se realizaron en la plataforma de Exiqon, Dinamarca. La calidad del RNA total se verificó mediante el Agilent 2100 Bioanalyzer y 250 ng de RNA, tanto de las muestras como el de referencia, se marcaron con señales fluorescentes HY3™ y HY5™, respectivamente, utilizando el miRCURY LNA™ microRNA Hi-Power Labeling Kit, HY3™/HY5™ (Exiqon, Dinamarca) siguiendo el procedimiento descrito por el fabricante. El RNA de las muestras, marcado con HY3™, y el de la muestra de referencia marcado con HY5™, se mezclaron por igual y se hibridaron al array de microRNA miRCURY LNA™ (Exiqon, Dinamarca), que contiene sondas de captura dirigidas a todos los miRNAs humanos, de ratón o de rata entrados en el miRBase 18.0. La hibridación se realizó siguiendo el manual de instrucciones del array utilizando una estación de hibridación Tecan HS4800™ (Tecan, Austria). Después de la hibridación, los resultados fueron escaneados y almacenados en un ambiente libre de ozono (<2,0 ppb de ozono) con el fin de evitar la extinción de los marcadores fluorescentes. EL escaneado se hizo utilizando el Sistema Agilent Microarray Scanner G2565BA (Agilent Technologies, Inc., EE.UU.) y el análisis de las imágenes se realizó utilizando el ImaGene® 9 (Análisis de Software microARN miRCURY LNA™, Exiqon, Dinamarca). Las señales fueron cuantificadas (Normexp with offset value 10, ver Ritchie, et al. [12]) y normalizadas utilizando el algoritmo de regresión global Lowess (Diagrama de dispersión suavizado localmente ponderado). Después de la normalización, se realizó el análisis de datos no supervisado y supervisado. Se realizaron diagramas de análisis de componentes principales (PCA) y de mapa de calor mostrando el agrupamiento jerárquico no supervisado. Los niveles de expresión se compararon mediante la t de Student. El umbral de significación se estableció en el logaritmo del cambio (logC) >1,5 veces y un valor de p<0,05.
Validación de los miRNAs expresados diferencialmente entre el grupo OP y el grupo control
La cuantificación de la expresión de miRNAs en las muestras de hueso total se realizó mediante PCR a tiempo real (qPCR) en la plataforma Exiqon (Dinamarca). Se transcribieron 10 ng de RNA a cDNA utilizando el kit miRCURY LNA™ Universal RT microRNA PCR, Polyadenylation and cDNA synthesis según las intrucciones de uso. El cDNA se diluyó 100 veces y se cuantificó la expresión de cada uno de los miRNAs mediante qPCR utilizando un panel personalizado de primers específicos (custom pick&mix panel, Exiqon) y la mastermix ExiLENT SYBR® Green. Los controles negativos se procesaron en paralelo a las muestras. La amplificación se realizó en un LightCycler® 480 Real-Time PCR System (Roche) en placas de 384 pocillos. Las curvas de amplificación fueron analizadas utilizando el software Roche LC para la determinación del valor Cq (por método de la segunda derivada). Las diferencias en los niveles de expresión entre los dos grupos se calculó como 2^ddCq. La eficiencia de amplificación se calculó usando algoritmos similares al software LinReg. Sólo se incluyeron en el análisis de datos los ensayos detectados con 3 Ct por debajo del control negativo y Ct <37. La normalización se realizó en base a la media de los ensayos detectados en todas las muestras, ya que está demostrado que es el mejor método para la normalización en estudios de qPCR que involucran numerosos ensayos [13]. Para el presente estudio, esto incluía 11 ensayos. La estabilidad de la media de 11 miRNAs era más alta que cualquier miRNA por si solo en el conjunto de datos medidos por el software NormFinder [14].
La fórmula utilizada para calcular los valores Cq normalizados (dCq) es:
Normalizado Cq = Media Cq - ensayo Cq (muestra)
Finalmente se realizó el control de calidad de los datos, el análisis de datos sin supervisión, y la prueba t de Student y el test de Wilcoxon para la comparación entre grupos (una p<0,05 se aceptó como significante).
Validación en osteoblastos primarios
Para la obtención de cDNA, se retrotranscribió 1 µg de RNA de cada muestra utilizando el kit miScript II RT (Qiagen).
La expresión de los miRNAs se cuantificó por qPCR, utilizando el kit MiScript Syber Green PCR (Qiagen) utilizando la secuencia del miRNA maduro como primer. El cDNA se diluyó a una razón de 1/5 y se utilizó 0,5 µl de muestra por pocillo, siguiendo el protocolo descrito por el proveedor. La amplificación se realizó en el sistema "QuantStudio 12K Flex Real-Time PCR" en placas de 384 pocillos, y se analizaron los datos con el software "expression suite". La expresión se analizó mediante cuantificación relativa RQ utilizando el método de la segunda derivada (ddCt). Se utilizó el snRNA U6 como control endógeno para normalizar las muestras. Cada experimento se hizo por triplicado. Para comparar las diferencias estadísticas entre los grupos con osteoporosis y sin osteoporosis se utilizó el test estadístico no paramétrico U de Mann-Whitney, utilizando el programa SPSS versión 12.0 para Windows.
Análisis bioinformatico de los miRNAs validados
Para el estudio de los genes diana de los miRNAs expresados diferencialmente se utilizó los siguientes programas: PicTar (http://pictar.mdc-berlin.de), TargetScan Human (http://www.targetscan.org), miRDB (http://mirdb.org), Miranda (http://www.microrna.org), DIANA-TarBase (http://diana.imis.athena-innovation.gr) y miRTarBase (http://mirtarbase.mbc.nctu.edu.tw). Se utilizó la herramienta DIANA-mirPath basada en la web computacional (11) para identificar las vías moleculares potencialmente alteradas y la intersección de los miRNAs expresados diferencialmente en el hueso fracturado. La información sobre la función proteica se obtuvo de la base de datos UniProtKB (http://www.uniprot.org).
Resultados
Descripción de las pacientes del estudio
Las características antropométricas, tanto de las pacientes cuyas muestras óseas se utilizaron para el array de expresión, como de las pacientes para llevar a cabo las validaciones de los resultados del array, se muestran en la tabla 1. No había diferencias en la edad e índice de masa corporal entre los dos grupos de pacientes (prueba de Mann-Whitney).
Análisis no supervisado del array de expresión
Cada muestra de hueso trabecular de cada una de las pacientes del estudio se analizó de forma individual en el microarray de miRNAs, y se realizó un análisis no supervisado de los resultados basado en el perfil de expresión con el fin de identificar los patrones de variación relacionados con factores biológicos o técnicos. Se realizó un análisis de componentes principales (PCA) incluyendo los 50 miRNAs con mayor variación de la expresión entre muestras para obtener una visión general de la agrupación de las muestras según su varianza (Figura 1). La agrupación de muestras no osteoporóticas (grupo control) mostró un perfil mucho más homogéneo que las muestras osteoporóticas. La muestra O-500, obtenida de una paciente con osteoporosis, se consideró un valor atípico y se excluyó de los análisis. El diagrama de mapa de calor corrobora los resultados del PCA con una clara agrupación de las muestras control y una agrupación más dispersa de las muestras osteoporóticas (Figura 2).
Comparación de la expresión de microRNAs entre OP y controles en las muestras de hueso total
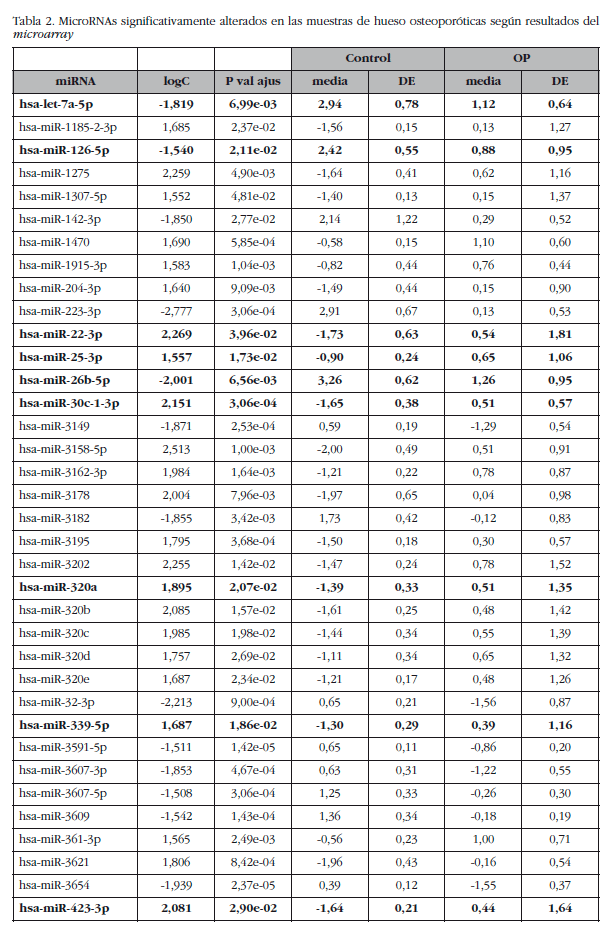
La media de los niveles de expresión de los miRNAs se comparó entre los grupos con y sin osteoporosis, con exclusión de la muestra O-500. Este análisis identificó un subgrupo de 82 miRNAs (sobre los 1.932 miRNAs analizados) cuyo valor absoluto del logaritmo de la ratio era mayor que 1,5 y con un p valor ajustado inferior a 0,05. Siete de estos miRNAs correspondieron a RNAs pequeños nucleolares con motivos C/D (SNORDs), tres a virus y uno era una secuencia miRPlus™ propiedad de Exiqon que no está anotada en la base de datos miRBase, y que fueron excluidos de la fase de validación.
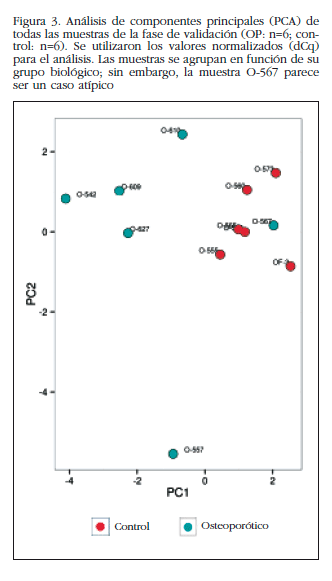
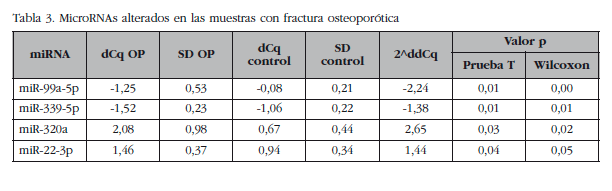
De los restantes (Tabla 2), se escogieron 15 hsa-miRNAs con los mejores valores de significación y para los que Exiqon disponía de sondas para su validación por qPCR: let-7a-5p, miR-126-5p, miR-30c-1-3p, miR-22-3p, miR-25-3p, miR-26b-5p, miR-339-5p, miR-423-3p, miR-320a, miR-483-5p, miR-491-3p, miR-574-5p, miR-631, miR-99a-5p y miR-99b-5p. El diagrama PCA de los resultados de la qPCR individuales mostró una agrupación de las muestras muy similar a los resultados del array de expresión, lo que corrobora la diferente fuente biológica de los dos grupos de muestras (Figura 3). Una muestra osteoporótica (O-567) se localizó en el grupo control y fue excluida del análisis. Después del análisis estadístico, cuatro miRNAs; miR-320a, miR-99a-5p, miR-339-5p y miR-22-3p mostraron diferencias significativas entre los grupos con osteoporosis y control (Tabla 3). Sin embargo, los miRNAs miR-99a-5p y miR-339-5p se encontraron sobreexpresados en las muestras OP en los resultados del array de expresión, mientras que estaban subexpresados en las muestras OP de la fase de validación. Estos resultados contradictorios sugieren un papel de estos miRNAs no relacionado con la patología osteoporótica y fueron descartados para el estudio in silico.
Comparación de la expresión de microRNAs entre OP y controles en los cultivos de HOb
De cada una de las muestras de hueso total se obtuvieron osteoblastos primarios en los cuales se analizó mediante qPCR la expresión de los miRNAs que previamente se habían validado en las muestras de tejido óseo. Los miRNAs miR-99a-5p y miR-339-5p no se detectaron en las células osteoblásticas, mientras que los miRNAs miR-320a y miR-22-3p sí que se expresaban en los HObs, aunque ninguno de los dos mostró diferencias significativas entre los dos grupos biológicos.
Predicción de genes diana y análisis de las vías de señalización de los miRNAs validados
Un análisis bioinformático exhaustivo mediante seis programas diferentes nos permitió predecir posibles genes diana para los miRNAs validados. El miR-320a puede regular genes involucrados en la proliferación celular (KRAS, PDGFD), la inhibición de la apoptosis (MCL1), transducción de señal (MAPK1, SOS2, PTEN), regulación de la expresión génica (RUNX2, PPARGC1A, SP1, CAMTA1, ESRRG), receptores de factores de crecimiento, hormonas y citoquinas (NPR1, BMPR1A, AR, IGF1R, ESR1), etc. Además, como dianas validadas se encuentran los genes CTNNB1, TFRC y POLR3D. Este miRNA participa en 44 de las vías de la enciclopedia de genes y genomas de Kioto (KEGG) según predicciones del programa DIANA-mirPath, donde la vía del cáncer de próstata es la más significativa (1,105e-12), seguida de la vía del cáncer endometrial (2,261e-10), la vía de señalización de mTOR (7,132e-08), y la vía de señalización de PI3K-Akt (3,914e-07).
El miR-22-3p tiene muchos genes diana validados, algunos de ellos muy importantes en la regulación del metabolismo óseo. Entre ellos encontramos el ESR1, el PRKACA (necesario para la diferenciación adipogénica y la inhibición de la diferenciación osteoblástica), HDAC4, SP1, BMP7 y CDK6 (antagonista de la señalización BMP2). La principal vía de señalización KEGG de este miRNA es la endocitosis (4,21e-05) aunque también actúa en vías tan importantes como la señalización P53 (p=0,003) y MAPK (p=0,003).
El software Diana tools utilizando los genes validados (según la base de datos Tarbase) muestra la vía de la endocitosis como la vía de intersección de los dos miRNAs, ya que comparten el gen diana TFRC (receptor de la transferrina). Por otro lado, si evaluamos la intersección teniendo en cuenta los genes diana predichos según la base de datos MicroT_CDS, las vías de señalización más significativas son la del cáncer de próstata y la de mTOR donde los dos miRNAs comparten los genes diana AKT3, PTEN y IGFR1.
Discusión
El presente estudio se centra en la identificación de miRNAs con expresión alterada en el hueso osteoporótico. Para ello se recogió hueso trabecular fresco de pacientes con o sin fractura osteoporótica, con parámetros antropométricos homogéneos, tales como la edad, índice de masa corporal, y género. Se excluyeron del estudio pacientes con trastornos que afectan la remodelación ósea. Las muestras procedentes de fractura osteoporótica mostraron una clara afectación en el patrón de expresión de miRNAs, demostrando que la regulación epigenética está alterada en el hueso patológico. Estos resultados pueden ayudar a una mejor comprensión de la biología del hueso que ha sufrido una fractura osteoporótica. Además, nos permitirían identificar moléculas susceptibles de ser utilizadas como dianas terapéuticas.
El análisis no supervisado de los resultados del array de expresión de miRNAs a partir de muestras óseas totales mostró que las muestras no osteoporóticas (control) se agrupaban generando un clúster biológico. Por otro lado, el hueso osteoporótico claramente difería de las muestras control, mostrando una distribución más dispersa, sugiriendo que la fisiopatología de la enfermedad osteoporótica y, en última instancia, la fractura del hueso, tienen una etiología heterogénea. El hecho de trabajar con muestras humanas frescas genera mayor variabilidad que trabajar con líneas celulares establecidas o modelos animales, haciendo difícil ver diferencias significativas entre grupos, pero ofrece un escenario más cercano a la situación fisiopatológica. En este sentido, nuestro estudio se ha basado en muestras de huesos humanos obtenidos con una mínima manipulación de laboratorio, lo que nos permite acercarnos a una situación lo más similar a las condiciones in vivo.
En el análisis de la expresión diferencial entre las muestras osteoporóticas y control, se encontraron 82 miRNAs que alcanzaron niveles de significación con un valor absoluto del logaritmo de la ratio mayor que 1,5 veces. De estos, 15 se testaron por qPCR en un nuevo conjunto de muestras, y los miRNAs miR-99a-5p, miR-339-5p, miR-320a y miR-22-3p fueron finalmente validados. Estos miRNAs se evaluaron en osteoblastos primarios (HOb) en cultivo, comparando células procedentes de muestras osteoporóticas versus células procedentes de muestras no osteoporóticas. Los miRNAs miR-99a-5p y miR-339-5p no se detectaron en los HOb, sugiriendo otro tipo celular como la fuente de estos dos miRNAs. Además, estos miRNAs se encontraron sobreexpresados en las muestras osteoporóticas del array mientras que estaban subexpresados en las muestras osteoporóticas de la fase de validación, descartando una relación de estos miRNAs con la osteoporosis. Los miRNAs, miR-320a y miR-22-3p sí se expresaban en los HOb, aunque no se observaron diferencias en sus niveles de expresión entre grupos de muestras. Esto podría ser debido a las condiciones artificiales de los cultivos celulares in vitro que afectarían la expresión de los microRNAs, sobre todo aquéllos implicados en la regulación de funciones celulares tan importantes como la proliferación y diferenciación.
Esto nos demuestra una vez más que, aunque el uso de cultivos de células, tanto de líneas establecidas como cultivos primarios, pueden ayudar a entender ciertos procesos celulares, los resultados obtenidos pueden diferir de las condiciones reales que se darían en el tejido original dentro del ambiente fisiológico.
Además, el uso del tejido fresco no manipulado para obtener datos más próximos a las condiciones fisiológicas también puede dar resultados dispares tal y como se demuestra en los estudios realizados por Seeliger, et al. [7] y Garmilla-Ezquerra, et al. [8]. Estos estudios también realizaron un microarray de expresión a partir de tejido óseo total comparando muestras osteoporóticas versus no osteoporóticas de manera muy similar al nuestro, pero con hallazgos diferentes. Cabe destacar que estos dos estudios anteriores tienen varias características que pueden explicar estas discrepancias. Entre ellas se encuentran los diversos arrays comerciales utilizados y el tamaño muestral. Además, estos estudios comparaban grupos biológicos con características antropométricas o clínicas no homogéneas, como la edad, género, índice de masa corporal y trastornos endocrinos (por ejemplo: diabetes mellitus), que son esenciales para la regulación del metabolismo óseo. Por lo tanto, los miRNAs identificados en estos estudios podrían estar también relacionados con otros parámetros externos, mientras que, en nuestro estudio, la ausencia de enfermedades concomitantes, así como las características similares entre los dos grupos, nos permite acercarnos de una manera más fiable a lo que sucede en la fractura osteoporótica.
Por esto, un esfuerzo importante realizado en nuestro trabajo es el control extremadamente cuidadoso de las potenciales características de confusión entre casos y controles en cuanto a edad, sexo, índice de masa corporal y las enfermedades metabólicas relacionadas con el envejecimiento. Estos estrictos criterios de inclusión restringen mucho nuestro tamaño de muestra en comparación con trabajos similares, donde las muestras son de pacientes con características más heterogéneas. Otra limitación de nuestro estudio es que las muestras no osteoporóticas utilizadas como controles procedían de pacientes con artrosis y, por lo tanto, no se puede descartar la presencia de otras anomalías óseas. Debido a razones éticas obvias no se permite la recogida de hueso a partir de individuos sanos. Sin embargo, en un intento de minimizar este potencial inconveniente, la muestra se obtuvo de hueso ubicado lo más distante posible de la lesión artrósica.
El miR-320a se encuentra conservado en humanos, ratón, rata y vaca. La secuencia de este miRNA está localizada dentro del promotor basal del gen POLR3D, que a su vez es uno de sus genes diana, silenciando así su expresión [15]. Además, se ha observado que el miR-320a está implicado en la regulación de la función osteoblástica, ya que tiene como genes diana el CTNNB1 (que codifica para la B-catenina) [16] y el RUNX2 [17].
Por otro lado, el miR-22-3p es un miRNA sérico que se ha asociado previamente a la fractura osteoporótica [9], así como ha sido implicado en la diferenciación osteogénica [18].
Los resultados de nuestro estudio, junto con los otros estudios, ofrecen un avance importante para la comprensión de la biología del hueso y de la implicación de los miRNA en la patología de la osteoporosis.
Conclusiones
Hemos identificado dos miRNAs que se sobreexpresan en muestras de hueso trabecular de pacientes con osteoporosis. La expresión de ambos miRNAs se ha detectado en osteoblastos primarios, aunque no se ha observado esta sobreexpresión en los cultivos procedentes de muestras osteoporóticas. No se conoce si la alteración de estos miRNAs es causa o efecto de la enfermedad y su relación con la fractura osteoporótica. No obstante, estos miRNAs podrían ofrecer un prometedor potencial para el diseño de nuevos fármacos para la osteoporosis.
Conflicto de intereses
Los autores declaran no tener ningún conflicto de intereses en relación a este trabajo.
Bibliografía
1. Zhang B, Pan X, Cobb GP, Anderson TA. microRNAs as oncogenes and tumor suppressors. Dev Biol 2007;302:1-12. [ Links ]
2. Taipaleenmaki H, Bjerre Hokland L, Chen L, Kauppinen S, Kassem M. Mechanisms in endocrinology: micro-RNAs: targets for enhancing osteoblast differentiation and bone formation. Eur J Endocrinol 2012;166:359-71. [ Links ]
3. Lian JB, Stein GS, van Wijnen AJ, Stein JL, Hassan MQ, Gaur T, et al. MicroRNA control of bone formation and homeostasis. Nat Rev Endocrinol 2012;8:212-27. [ Links ]
4. van Wijnen AJ, van de Peppel J, van Leeuwen JP, Lian JB, Stein GS, Westendorf JJ, et al. MicroRNA Functions in Osteogenesis and Dysfunctions in Osteoporosis. Curr Osteoporos Rep 2013;11:72-82. [ Links ]
5. Li H, Xie H, Liu W, Hu R, Huang B, Tan YF, et al. A novel microRNA targeting HDAC5 regulates osteoblast differentiation in mice and contributes to primary osteoporosis in humans. J Clin Invest 2009;119:3666-77. [ Links ]
6. Lei SF, Papasian CJ, Deng HW. Polymorphisms in predicted miRNA binding sites and osteoporosis. J Bone Miner Res 2011;26:72-8. [ Links ]
7. Seeliger C, Karpinski K, Haug A, Vester H, Schmitt A, Bauer J, et al. Five Freely Circulating miRNAs and Bone Tissue miRNAs are Associated with Osteoporotic Fractures. J Bone Miner Res 2014;29:1718-28. [ Links ]
8. Garmilla-Ezquerra P, Sanudo C, Delgado-Calle J, Perez-Nunez MI, Sumillera M, Riancho JA. Analysis of the Bone MicroRNome in Osteoporotic Fractures. Calcif Tissue Int 2015;96:30-7. [ Links ]
9. Weilner S, Skalicky S, Salzer B, Keider V, Wagner M, Hildner F, et al. Differentially circulating miRNAs after recent osteoporotic fractures can influence osteogenic differentiation. Bone 2015;79:43-51. [ Links ]
10. Guo L, Zhao RC, Wu Y. The role of microRNAs in self-renewal and differentiation of mesenchymal stem cells. Exp Hematol 2011;39:608-16. [ Links ]
11. Kapinas K, Delany AM. MicroRNA biogenesis and regulation of bone remodeling. Arthritis Res Ther 2011;13:220. [ Links ]
12. Ritchie ME, Silver J, Oshlack A, Holmes M, Diyagama D, Holloway A, et al. A comparison of background correction methods for two-colour microarrays. Bioinformatics 2007;23:2700-7. [ Links ]
13. Mestdagh P, Van Vlierberghe P, De Weer A, Muth D, Westermann F, Speleman F, et al. A novel and universal method for microRNA RT-qPCR data normalization. Genome Biol 2009;10:R64. [ Links ]
14. Andersen CL, Jensen JL, Orntoft TF. Normalization of real-time quantitative reverse transcription-PCR data: a model-based variance estimation approach to identify genes suited for normalization, applied to bladder and colon cancer data sets. Cancer Res 2004;64:5245-50. [ Links ]
15. Kim DH, Saetrom P, Snove O Jr., Rossi JJ. MicroRNA-directed transcriptional gene silencing in mammalian cells. Proc Natl Acad Sci U S A 2008;105:16230-5. [ Links ]
16. Sun JY, Huang Y, Li JP, Zhang X, Wang L, Meng YL, et al. MicroRNA-320a suppresses human colon cancer cell proliferation by directly targeting beta-catenin. Biochem Biophys Res Commun 2012;420:787-92. [ Links ]
17. Yu F, Cui Y, Zhou X, Zhang X, Han J. Osteogenic differentiation of human ligament fibroblasts induced by conditioned medium of osteoclast-like cells. Biosci Trends 2011;5:46-51. [ Links ]
18. Trompeter HI, Dreesen J, Hermann E, Iwaniuk KM, Hafner M, Renwick N, et al. MicroRNAs miR-26a, miR-26b, and miR-29b accelerate osteogenic differentiation of unrestricted somatic stem cells from human cord blood. BMC Genomics 2013;14:111. [ Links ]
![]() Dirección para correspondencia:
Dirección para correspondencia:
Natalia García Giralt
c/Dr. Aigüader, 88
08003 Barcelona (España)
Correo electrónico: ngarcia@imim.es
Fecha de recepción: 20/10/2015
Fecha de aceptación: 10/12/2015











 texto em
texto em