My SciELO
Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Cited by Google
Cited by Google -
 Similars in
SciELO
Similars in
SciELO -
 Similars in Google
Similars in Google
Share
Archivos de Zootecnia
On-line version ISSN 1885-4494Print version ISSN 0004-0592
Arch. zootec. vol.60 n.231 Córdoba Sep. 2011
https://dx.doi.org/10.4321/S0004-05922011000300048
Desarrollo de un ensayo por minisecuenciación de ocho SNP asociados a producción láctea
Development of an eight-plex SNP assay associated with milk production
Acosta, A.C.1*, Sanz, A.2, Uffo, O.1, Ronda, R.1, Osta, R.2, Martin, I.2, Rodellar, C.2 y Zaragoza, P.2
1Laboratorio de Genética Molecular. Centro Nacional de Sanidad Agropecuaria. San José de las Lajas. La Habana. Cuba. *atzel@censa.edu.cu
2Laboratorio de Genética y Bioquímica. Facultad de Veterinaria. Universidad de Zaragoza. Zaragoza. España
RESUMEN
Los altos niveles de producción alcanzados en la explotación animal se deben en gran medida al trabajo de selección que se ha desarrollado en las principales especies de interés económico. El empleo de la información molecular en los programas de selección asistida ha abierto un amplio campo en la mejora animal. Se describe la metodología empleada para el genotipado simultáneo de los genes que codifican para las seis principales proteínas lácteas, la hormona del crecimiento y prolactina bovinas. Fueron utilizadas tres técnicas para el genotipado: digestión con enzimas de restricción, secuenciación y minisecuenciación para determinar SNPs (Single Nucleotide Polymorphisms), donde las dos primeras se usaron como control de los resultados obtenidos por minisecuenciación. Esta última metodología permite la automatización del análisis, el trabajo simultáneo con varias muestras y 10 o más marcadores en una sola reacción. Se observó que los genes que codifican para αs2-caseína y prolactina se comportaron monomórficos. Para la as1-caseína se identificaron los alelos B y C; en la β-caseína se identificaron los alelos A y B; por otra parte para la k-caseína fueron los alelos A y B los observados. En la α-lactoalbúmina y β-lactoglobulina se identificaron los alelos A y B, mientras que para la hormona del crecimiento se observaron los alelos L y V. En todos los casos resultaron ser los alelos que con mayor frecuencia se describen. Se corroboró que la técnica de minisecuenciación simplifica el análisis y es útil en la caracterización molecular dirigida a una futura selección asistida por marcadores.
Palavras chave: Proteínas lácteas. Selección asistida por marcadores. Hormona del crecimiento y prolactina.
SUMMARY
High production levels achieved in animal exploitation is due largely to the selection work that has developed in the main species of economic interest. The use of the molecular information in the programs of assisted selection has opened a big field in the animal improvement. The methodology for the simultaneous genoptyping of the genes that encode the six major milk proteins, growth hormone and bovine prolactin is described. Three techniques were used for genotyping: restriction enzyme digestion, sequencing and minisequencing to determine SNPs (Single Nucleotide Polymorphisms), where the first two were used as control to the Results of minisequencing. This latter methodology enables the automation of analysis, simultaneous work with several samples and ten or more markers in a single reaction. It was found that the genes encoding for αs2-casein and prolactin behaved as monomorphic. For the αs1-casein the alleles B and C were identified; in the β-casein the alleles A and B were identified; on the other hand for the kcasein the alleles A and B were the observed ones. In the a-lactoalbumin and β-lactoglobulin the alleles A and B were identified, moreover for the growth hormone, alleles L and V were observed. In all cases the most frequent alleles are the described ones. It was confirmed that the minisequencing technique simplifies the analysis and it is useful for the molecular characterization focused a future targeted molecular markerassisted selection.
Key words: Milk protein. Marker assisted selection. Growth hormone and prolactin.
Introducción
Con el progreso científico técnico se han incorporado nuevas metodologías para incrementar la eficiencia de los actuales programas de selección, como la determinación de los SNPs, uno de los marcadores empleados dentro de la más reciente generación de estos. En la práctica, los SNPs son bialélicos, debido a la baja probabilidad de que un nucleótido sustituya a los nucleótidos originales del SNP, que se estima entre 1x10-9 y 5x10-9 por nucleótido y por año sobre la posición inicial en los mamíferos (Li et al., 1981; Martínez-Arias et al., 2001).
El empleo de la información molecular en los programas de selección asistida ha abierto un amplio campo en la mejora animal, que se ha denominado selección asistida por marcadores MAS (Marker Assisted Selection) y que incrementa la precisión y eficiencia de esta selección (Hua et al., 2009). Un gran número de investigaciones han tenido como objetivo fundamental la identificación y estudio de loci asociados a parámetros cuantitativos QTL (Quantitative Trait Loci) lográndose incorporar esta información a los programas de selección artificial (Malveiro et al., 2001; Lara et al., 2002; Hua et al., 2009). Dentro de las ventajas de este sistema se destacan que la determinación de los genotipos en los animales puede ser estudiada en cualquier etapa de desarrollo del mismo, además de que dichos estudios se pueden realizar en animales que no expresan fenotípicamente el carácter de interés. Esto, junto a las tecnologías de la reproducción como la inseminación artificial y la fertilizaciónin vitro permiten incrementar rápidamente la proporción de genes favorables para un carácter de interés, lo que aumenta el progreso genético por generación.
Gran parte de los esfuerzos investigativos han estado dedicados a los genes candidatos a QTL, los cuales codifican proteínas que por su función biológica pudieran estar relacionadas con caracteres productivos de interés. Tal es el caso de los genes que codifican para las seis principales proteínas lácteas bovinas (αs1-caseína, αs2-caseína, β-caseína, k-caseína, α-lactoalbúmina y βlactoglobulina), así como la hormona del crecimiento y la prolactina. Las caseínas se comportan como una unidad genética en la cual la combinación de alelos puede ser una característica peculiar de una raza (Formaggioni et al., 1999). Dentro de este grupo de proteínas la αs2-caseína (CASA2) presenta la variante A como la más frecuentemente en poblaciones bovinas la cual fue descrita por primera vez por Brignon et al. (1977). En el caso de las tres restantes caseínas, se refiere en mayor o menor grado cierta relación con caracteres productivos o de composición de la leche. El alelo B de la αs1-caseína (CASA1) se ha asociado favorablemente a producción de leche, producción de grasa y proteína total (Ng-Kwai-Hang et al., 1986; Aleandri et al., 1990).Estos autores describen al alelo B de la βcaseína (CASB) asociado positivamente ala producción de grasa y porcentaje de caseína, mientras que el alelo A se asociafavorablemente a producción de leche yproteína total. Para la industria láctea elgenotipo de la k-caseína (CASK) tiene unagran importancia, pues la leche procedentede animales con genotipo BB presenta mayores porcentajes de proteína, mejores propiedades de coagulación y sinéresis de losquesos (Giovambattista et al., 1998).
Los genes que codifican para α-lactoalbúmina (LAA) y la β-lactoglobulina(LGB) se encuentran en el cromosoma 5 (Fries et al., 1993) y cromosoma 11 (Hayes y Petit 1993), respectivamente; esto explica su segregación independiente con respecto a las caseínas (Formaggioni et al., 1999). El alelo A de la LAA se describe asociado favorablemente a la producción de grasa y proteínas totales (Bleck y Bremel, 1993), mientras que para la LGB el alelo A se relaciona de manera favorable con producción de leche y proteínas del suero (Ng-Kwai-Hang et al., 1986; Van der Berg et al., 1990; Bolla et al., 1992) y el alelo B con producción de grasa, proteína total y porcentaje de caseína (Hill, 1993).
La hormona del crecimiento bovina (GH) ejerce un papel muy importante en el crecimiento, desarrollo y lactación de la glándula mamaria en el ganado bovino (Etherton, 2004; Akers 2006; Chagaset al.,2007; Thidar-Myint et al., 2008), por tal motivo el gen bGH, que codifica para la misma es blanco potencial para estudios de variación genética relacionados con producción de leche y rasgos de crecimiento en bovinos (Høj et al., 1993). La selección para rendimiento de leche se ha asociado con el incremento de los niveles en sangre de esta hormona (Lukes et al., 1989), pero esto también depende del estado fisiológico del animal y varía con el momento del día en que sea medida. La presencia del alelo V de la bGH se ha asociado con mayores rendimientos lecheros; en tal sentido Schlee et al. (1994) y Grochowska et al. (2001) describen una relación positiva entre dicho alelo y el mérito genético para la producción de leche en ganado Holstein. Sin embargo los resultados de Høj et al. (1993), contrastan con los anteriores pues indican un efecto positivo del alelo L sobre los caracteres asociados con la producción de leche.
La prolactina (PRL) tiene una gran importancia en el desarrollo mamario y producción de leche (Forsyth, 1983; Mellai et al., 2003); tras la ocurrencia del parto, sus concentraciones en sangre son reguladas por el estímulo de vaciado de la ubre, que limita la liberación del factor inhibitorio o PIF, lo que trae consigo la síntesis y liberación de PRL (Gorewitet al., 1992). Debido al gran número de rutas metabólicas en las que participa dicha hormona, se conoce como la más versátil del conjunto de hormonas secretadas por la hipófisis (Cabrera et al., 2000); esto explica el creciente interés de la comunidad científica en el estudio del gen que codifica su síntesis (Rocha et al., 1992; Udina et al., 2001; Brym et al., 2005). A partir de estas investigaciones se ha identificado el polimorfismo de longitud existente en el exón 3 debido al cambio de A.G y polimorfismo de microsatélite de la región regulatoria 5´ de dicho gen (Udina et al., 2001).
La técnica de minisecuenciación o primer extension implica la utilización de un cebador que solo se extiende una base (single base extensión, SBE primer) y cuyo extremo 3´ se ubica junto a la base que precede al SNP. Para lograr la extensión se agrega una ADN polimerasa y ddNTPs marcados con cuatro colorantes fluorescentes. Para analizar los resultados puede utilizarse cualquier método que permita separar los oligonucleótidos marcados de los ddNTPs marcados no incorporados. (Syvanen, 1999; Sauer et al., 2000). Esta metodología permite la automatización del análisis, el trabajo simultáneo con varias muestras y 10 o más marcadores en una sola reacción (Sanz, 2002), aspectos estos que facilitan las investigaciones en genética poblacional que se caracterizan por el estudio de muestras relativamente numerosas.
El objetivo del presente trabajo es describir la metodología empleada para la determinación por minisecuenciación de los SNPs en seis de las principales proteínas lácteas bovinas, la hormona del crecimiento y la prolactina.
Material y métodos
ANIMALES Y OBTENCIÓN DEL MATERIAL GENÉTICO
Fueron incluidos en el estudio un total de 20 vacas de la raza Taíno de Cuba (5/8 Holstein-3/8 Criollo cubano) a partir de los cuales se colectaron muestras de sangre periférica con pleno cumplimiento de las normas establecidas por el comité de ética y bienestar animal. Se emplearon 0,5 ml de 0,5 M EDTA como anticoagulante. Se procedió a la extracción y purificación del ADN mediante el método de precipitación salina descrito por Miller et al. (1988). Se determinaron las concentraciones de ADN por espectrofotometría, realizándose la lectura de absorbancia a 260 nm. Para estimar pureza fueron realizadas lecturas a 260 y 280 nm y se hicieron las diluciones correspondientes hasta alcanzar una concentración en cada muestra de 20 pmol/μl.
GENOTIPADO DE LAS SEIS PROTEÍNAS LÁCTEAS, LA HORMONA DEL CRECIMIENTO Y LA PROLACTINA
Digestión con enzimas de restricción
Las condiciones de reacción fueron descritas por Osta y Uffo (Osta, 1994; Uffo, 2003). La mezcla de reacción estaba compuesta de Buffer de Reacción 10X libre de magnesio, 50 mM MgCl2, 1,25 mM dNTPs, 20 nmol/μl oligonucleótidos correspondientes para cada uno de los fragmentos a amplificar (tabla I) y 5 U/μl de ADN polimerasa comercial (Promega). Se emplearon 20 ng/μl ADN por cada muestra y se completó el volumen a 30 μl con agua.
Tres metodologías fueron empleadas para establecer el genotipo de las muestras problemas: la creación de los sitios de restricción o ACRS (Amplification Created Restriction Sites) para CASA1 (Lien et al., 1993) y CASB (Medrano and Sharrow, 1991), tipificación del polimorfismo de los fragmentos de restricción (RFLP, Restriction Fragment Length Polymorphism) para la CASA2 (Osta, 1994), la LAA (Osta, 1994) y LGB (Medrano y Aguilar-Córdova, 1990) y la metodología oligonucleotidos alelo-específico (ASO, Allele Specific Oligonucleotides) empleada parael genotipado de la KCAS (Osta, 1994). A partir de 10 μl del producto amplificado se procedió a la digestión con las enzimas específicas para cada uno de los fragmentos las cuales se muestran en la tabla II. La temperatura de incubación fue de 37oC durante 3 horas (Osta, 1994; Uffo, 2003). El producto digerido fue visualizado en gel de electroforesis agarosa 4%, TBE 0,5X, con tinción por bromuro de etidio a una concentración de 10 mg/ml.
Secuenciación
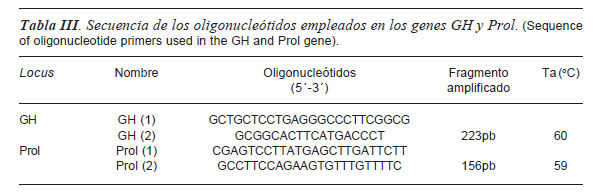
Los oligonucleótidos descritos en la tabla I se emplearon a una concentración de 20 nmol/μl para la obtención de los fragmentos a secuenciar. En el caso de la KCAS se preparó una mezcla de los oligonucleótidos KCAS(1) y KCAS(2) con una concentración inicial en ambos casos de 20 nmol/μl. Para el caso de la hormona del crecimiento (Schlee et al., 1994) y la prolactina (Mitra et al., 1995; Chrenek et al., 1998), se utilizaron los oligonucleótidos que se muestran en la tabla III, a una concentración de 20 nmol/μl.
A partir del producto amplificado se preparó una segunda reacción donde se utilizó el Kit BigDye terminator v 3,1 Cycle Squencing con los oligonucleótidos directos e inversos por separado, siguiendo las indicaciones del fabricante. El programa empleado fue de 96oC por 1min, 25 ciclos de 96oC por 10 seg 50oC por 5 seg y 60oC por 4 seg.
El producto amplificado para secuenciar se purificó con el Kit BigDye X TerminatorTM Purification, según indicaciones del fabricante. Se procedió a secuenciar el fragmento de interés en un secuenciador automático ABI Prism 3130 y se utilizó el programa BioEdit (Hall, 1999) para visualizar el resultado. En el caso de la secuenciación las muestras trabajadas fueron empleadas como controles en el proceso de minisecuenciación.
Reacción de minisecuenciación para determinar los SNPs
Se optimizaron las condiciones de reacción para la obtención de PCR múltiple que permitió la amplificación de los ocho fragmentos de interés. Se emplearon 2,5 μl de Kit QIAGEN® Multiplex PCR Master Mix 2X que contiene los dNTPs, tampón de PCR, MgCl2 y HotStarTaq DNA Polymerase, 0,5 μl solución QIAGEN®, 1,5 μl de la mezcla de oligonucleótidos (tablas I y III), excepto para el fragmento KCAS donde se diseñó una nueva pareja de cebadores que se muestran en la tabla IV, utilizando 1,25 nmol/μl como concentración final de cada cebador. Se utilizó 1 μl de la muestra de ADN a genotipar y 0,5 μl de agua para un volumen final de reacción de 6 μl. El programa empleado para la amplificación simultánea de los fragmentos fue diseñado en tres grupos de ciclos. Se partió de la desnaturalización a 95oC durante 15 min, 12 ciclos de por 95oC por 30 seg, 58oC por 1 min y 72oC por 1 min; 12 ciclos de 95oC por 30 seg, 60oC por 1 min y 72oC por 1 min; 17ciclos de 95oC por 30 seg, 62oC por 1 min y 72oC por 1 min; finalmente un periodo de elongación de 30 min a 72oC.
Para la purificación de los fragmentos amplificados se utilizaron 2 μl de la enzima Exosap y 5 μl del producto amplificado, que se mantuvieron en incubación a 37oC durante 45 min e inactivación de la enzima a 80oC durante 15 min. Tras la purificación de los fragmentos se preparó la reacción de minisecuenciación que permitió identificar el polimorfismo de una base descritos en la tabla V.
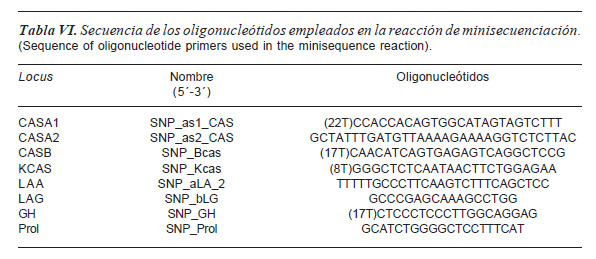
Se preparó previamente una mezcla de oligonucleótidos, los cuales se relacionan en la tabla VI y cuya orientación se muestra en la figura 1, dentro del panel de minisecuenciación. Se utilizó una concentración final de 2,5 nmol/μl de los cebadores en la mezcla de reacción; se empleó 1 μl de Kit SnatPshot, 1 μl de la mezcla de oligonucleótidos, 1,5 μl del producto amplificado y purificado y 2,5 μl de agua para completar un volumen de 6 μl.
El programa empleado incluyó 25 ciclos de 96oC por 10 seg, 50oC por 5 seg y 60oC durante 30 seg. El producto de esta reacción se purificó con la enzima Shrimp Alkaline Phosphatasa (SAP) agregándose 1 μl a cada producto amplificado, incubado a 37oC durante 1 hora y seguido de la inactivación de la enzima a 75oC en 15 min.
Se empleó la metodología de electroforesis capilar en el secuenciador automático ABI Prism 3130 con el polímero Pop7. Las muestras se prepararon tomando 1 μl del producto de la reacción de minisecuenciación, 0,3 μl de GeneScanTM -120 LizTMSize Standard y 9,7 μl de formamida Hi-Di para un volumen final de 11 μl. La visualización del resultado se realizó a través del programa GeneMapper v3,7 (GeneMapper 2004).
Resultados y discusión
Se pudieron identificar los dos alelos que con mayor frecuencia se han descrito para la CASA1 (Laraet al., 2002; Litwiñczuk y Król, 2002; Uffo et al., 2006): los alelos B y C, que se diferencian en la sustitución de A→G, lo que provoca un cambio en el aminoácido 192 durante la síntesis proteica (de ácido glutámico a glicina). El hallazgo del alelo C en la raza Taíno de Cuba corrobora la presencia de genes Bos indicus en el ganado Criollo de Cuba, previamente establecido por Sánchez et al., 1977. Uffo, (2003) y Uffo et al. (2006), si tenemos en cuenta las elevadas frecuencias del alelo CASA1C son típicas de animales acebuados y el origen del ganado Criollo de Cuba.
La CASA2 se comportó monomórfica para el alelo A; este hecho coincide con los resultados obtenidos en investigaciones previas en animales pertenecientes a la raza Siboney de Cuba, Cebú Cubano y Criollo de Cuba (Uffo et al., 2006). Esto también fue descrito en la raza española Rubia Gallega según Osta et al. (1995), aunque otros autores han encontrado el alelo D en razas europeas como Parda Alpina (n= 0,07), Asturiana de los Valles (n= 0,06), algunos rebaños de Frisona (n= 0,01) y las razas autóctonas francesas Montbèliarde y Vosgiènhe (Grosclaude, 1988), todos todas en frecuencias muy bajas. También Ikonen et al.(1996) y Erdhardt en el mismo año, encontraron el alelo A prácticamente fijado en las razas Ayrshire Finlandesa (n= 0,991) y Limpurger alemana (n= 0,984).
Para la CASB se identificaron los alelos A y B, que son los que aparecen con mayor frecuencia en poblaciones bovinas (Litwiñczuk y Król, 2002). El estudio de las variantes genéticas de esta proteína ha tenido un especial interés debido a que la variante B proporciona características idóneas para la producción de queso (Jacob y Puhan, 1992). Las diferencias entre estas dos variantes genéticas radican en la sustitución del nucleótido C por G que provoca un cambio aminoacídico en la posición 122, que cambia a una Serina por Arginina.
Se identificaron dos variantes genéticas para la KCAS, siendo estas las correspondientes con los alelos A y B, que difieren entre sí por un cambio de nucleótido A por C, lo que provoca la sustitución en la posición 148 del ácido aspártico por alanina. Estos alelos están descritos en poblaciones de razas portuguesas (Beja-Pereira et al., 2002), hindúes (Malik et al., 1997), venezolanas (Escoda et al., 1981), así como en poblaciones bovinas cubanas (Uffo, 2003).
El alelo A del gen que codifica para LAA está descrito como marcador racial de Bos indicus (Eigel et al., 1984). En investigaciones previas fue demostrada la presencia de genes Cebú dentro de las poblaciones de ganado Criollo Cubano debido al mestizaje que ha sufrido este último con el anterior (Uffo et al., 2006). En las muestras analizadas se identificaron los alelos A y B, lo que se corresponde con las razas que dieron origen al genotipo Taíno de Cuba. La diferencia entre estos alelos consiste en un cambio del nucleótido A por G que provoca en el momento de la síntesis proteica, una sustitución del aminoácido 10 de la estructura primaria de la proteína consistente en la sustitución de glicina por arginina.
Se identificaron los alelos A y B de la LGB, que se diferencian por el cambio de una T por una C, lo que trae consigo que en la estructura primaria de la proteína traducida, el aminoácido 118 (valina) sea sustituido por una alanina. Estos alelos se han descrito en poblaciones bovinas españolas (Osta, 1994), venezolanas (Escoda et al., 19981), polacas (Litwiñczuk y Król, 2002), así como en poblaciones cubanas (Uffo et al., 2006). Estas variantes proteícas, al igual que los de la KCAS han sido ampliamente utilizadas para la selección de animales con genotipo favorable para mejorar las características para la manufactura de los quesos (Ng-Kwai-Hang et al.,1986; Van der Berg et al., 1990; Bolla et al., 1992; Hill, 1993).
En el caso de la GH están descrito los alelos L y V (leucina/valina) en la posición 127 de la estructura primaria de la hormona para el ganado de carne Canchim (5/8Charolais y 3/8 Cebú) (Pereira et al., 2005) así como en descendientes de una vaca alta productora en Cuba (Uffo et al., 2002). En las muestras genotipadas en el presente trabajo se identificaron ambos alelos, lo cual permitirá hacer, en un futuro, estudios de asociación con caracteres productivos y de composición láctea en la raza en cuestión.
Para la PRL se han descrito dos alelos (A y B) en tres razas rusas (Udina et al., 2001), pero no fue encontrado el genotipo BB dentro de los animales estudiados por el mismo autor. Por otra parte las mayores frecuencias del alelo B se han observado en animales de la raza cebuína Sahiwal (0,51), mientras que la más baja se observó en animales de la raza Holstein (0,05) (Mitra et al., 1995). En nuestro estudio solo se pudo identificar la presencia del alelo A, con un comportamiento monomórfico para este gen. Esto puede deberse al pequeño número de muestras trabajadas.
Se observó que los genes que codifican para CASA2 y PRL se comportaron monomórficos mientras que para el resto de los genes estudiados (CASA1, CASB, KCAS, LAA, LAG y GH) se identificaron los polimorfismos descritos. En una única reacción se obtienen los resultados para ocho SNPs, lo que permite el aumento del número de muestras analizadas por unidad de tiempo. Otras investigaciones recomiendan el empleo de esta la metodología de minisecuenciación para la determinación de SNPs y se resaltan las amplias posibilidades de automatización de la misma, el trabajo de forma simultánea con varias muestras y la posibilidad de testar 10 o más marcadores en una misma reacción (Padilla et al., 2007).
Conclusión
Este trabajo describe la metodología empleada para el genotipado en una única reacción, de ocho SNPs en los genes que codifican para las seis principales proteínas lácteas, la hormona del crecimiento y prolactina, regiones importantes del genoma bovino para la mejora de la producción y la calidad de la leche y que permitirá el empleo de esta información un futuro programa de selección asistida por marcadores. La metodología de minisecuenciación ha resultado ser rápida y fiable para el genotipado de los genes de interés seleccionados para el presente estudio.
Agradecimientos
Es intención de los autores agradecer la concesión de una beca de investigación por MAEC-AECID que contribuye a la formación de personal calificado. También agradecemos las facilidades brindadas por la Dirección Nacional de Genética y la Empresa Pecuaria Genética del Este de la Habana para la obtención de las muestras de los animales Taíno de Cuba.
Bibliografía
Akers, R. 2006. Major advances associated with hormone and growth factor regulation of mammary growth and lactation in dairy cows. J. Dairy Sci., 89: 1222-1234. [ Links ]
Aleandri, R., Buttazzoni, L., Schneider, J., Carili, A. and Davoli, R. 1990. The effects of milk protein polymorphism on milk components and cheeseproducing ability. J. Dairy Sci., 73: 241-255. [ Links ]
Beja-Pereira, A., Erhardt, G., Matos, C., Gama, L. and Ferrand, N. 2002. Evidence of the geographical cline of casein haplotypes in Portuguese cattle breeds. Anin. Genet., 33: 295. [ Links ]
Bleck, G. and Bremel, R. 1993. Sequence and single base polymorphism of the bovine aLA 5'flanking region. Gene, 126: 213-218. [ Links ]
Bolla, P., Caroli, A., Rizzi, R., Barcoli, D. and Pedro, O. 1992. Effect of milk genetic markers on the lactation avive in dairy cattle. XXIII International Conference on Animal Genetics. Interlaken. Switzerland. [ Links ]
Brignon, G., Ribadeau-Dumas, B., Mercier, J. Pélissier, J. and Das, B. 1977. Complete aminoacid sequence of the precursor of ovine caseins. Lett, 76: 274-279. [ Links ]
Brym, P., Kamiñski, S. and Wójcik, E. 2005. Nucleotide sequence polymorphism within exon 4 of the bovine prolactin gene and its associations with milk performance traits. J. Appl. Genet., 45: 179-85. [ Links ]
Cabrera, V., Rodrígez, J., Méndez, I., Larrea, F. y Chapé, A. 2000. Caracterización bioquímica de prolactina de diferentes especies. Rev. Cubana Endocrinol., 11: 78-89. [ Links ]
Chagas, L.M., Bass, J.J., Blanche, D., Burke, C.R., Kay, J.K., Lindsay, D.R., Lucy, M.C., Martin, G.B., Meier, S., Rhodes, S., Roche, J.R., Thatcher, W.W. and Webb, R. 2007. Invited review: new perspectives on the roles of nutrition and metabolic priorities in the subfertility of highproducing dairy cows. J. Dairy Sci., 90: 40224032. [ Links ]
Chrenek, P., Vasicek, D., Bauerovf, M. and Bulla, J. 1998. Simultaneous analysis of bovine growth hormone and prolactin alleles by multiplex PCR and RFLP. C'zech. J. Anim. Sci., 43: 53-55. [ Links ]
Eigel, W.N., Butler, J.E., Erstrom, C.A., Farrel, H.M., Harwalker, V.R., Jenness, R. and Whitney, R. 1984. Nomenclature of protein of cow's milk: fifth revision. J. Dairy Sci., 67: 1599-1631. [ Links ]
Escoda, A.B, Alvarez, L.O. y Yerez, S. 1981. Estudio de los polimorfismos genéticos de las proteínas de la leche producida en algunas hacienda de la zona de Carora. Rev. Facultad Agronomía, 6: 714-716. [ Links ]
Etherton, T.D. 2004. Somatotropic function: the somatomedin hypothesis revisited. J. Anim. Sci., 82: 239-244. [ Links ]
Formaggioni, P., Summer, A., Malacarne, M. and Mariani, P. 1999. Milk protein polymorphism: detection and diffusion of the genetic variants in bos genus. Università degli Studi di Parma Annali della Facoltà di Medicina Veterinaria. Disponible en: http://www.unipr.it/arpa/facvet/annali/1999/. (02/02/09). [ Links ]
Forsyth, I.A. 1983. The endocrinology of lactation. In: Mepham T.B. Biochemistry of lactation. Elsevier Science Publishers BV. pp. 309-349. [ Links ]
Fries, R., Eggen, A. and Womack, J. 1993. The bovine genome map. Mamm. Genome, 4: 405-428. [ Links ]
Giovambattista, G., Ripoli, M.V., Peral-García, P., De Lúea, J.C. y Dulout, F.N. 1998. Estado actual del mapeo de qtls para produccion lechera. Agro sur (serial on the Internet). Disponible en: http://mingaonline.uach.cl/scielo.php?script=sci_arttext&pid=S0304-88021998000200009&lng=en&nrm=iso. (10/11/08). [ Links ]
Gorewit, R.C., Svennersten, K., Butler, W.R. and Uvnas-Moberg, K. 1992. Endocrine responses in cows milked by hand and machine. J. Dairy Sci., 75: 443-448. [ Links ]
Grochowska, R., Sørensen, P., Zwierzchowski, L., Snochowski, M. and Løvendahl, P. 2001. Genetic variation in stimulated GH release and in IGF-I of young dairy cattle and their associations with the leucine/valine polymorphism in the GH gene. J. Anim. Sci., 79: 450-476. [ Links ]
Grosclaude, F. 1988. Le polymorphisme genetique des principales lactoproteines bovines. Relation avec la queantite, la compisition et les aptitudes fromageres du lait. Inra Prod. Anim., 1: 15-17. [ Links ]
Hall, T.A. 1999. BioEdit: a user-friendly biological sequence alignment editor and analysis program for Windows 95/98/NT. Nucl. Acids. Symp. Ser., 41: 95-98. [ Links ]
Hayes, H.C. and Petit, E.J. 1993. Mapping of the alpha-lactoglobulin gene and of an immunoglobulin M heavy chain-like sequence to homologous cattle, sheep and goat chromosomes. Mammal. Genome, 4: 207. [ Links ]
Hill, J.P. 1993. The relationship between beta-lactoglobuline phenotypes and milk composition in New Zealand Dairy Cattle. J. Dairy Sci., 76: 281-286. [ Links ]
HØj, S., Fredholm, M., Karsen, N.J. and Nielsen, V.H. 1993. Growth hormone gene polymorphism associated with selection for milk fat production in lines of cattle. Anim. Genet., 24: 91-96. [ Links ]
Hua, G.H., Chen, S.L., Yu, J.N., Cai, K.L., Wu, C.J., Li, Q.L., Zhang, C.Y., Liang, A.X., Han, L., Geng, L.Y., Shen, Z., Xu, D.Q. and Yang, L.G. 2009. Polymorphism of the growth hormone gene and its association with growth traits in boer goat bucks. Meat Sci., 81: 391-395. [ Links ]
Ikonen, T., Ruottinen, O., Erhardt, G. and Ojala, M. 1996. Allele frequencies of the major milk protein in the finish ayrshire and detection of a new k-casein variant. Anim. Genet., 27: 179-181. [ Links ]
Jacob, E. and Puhan, Z. 1992. Technological properties of milk as influenced by genetic polymorphism of milk proteins. Int. Dairy J., 2: 157-178. [ Links ]
Lara, M.A.C., Gama, L.T., Bufarah, G., Sereno, J.R.B., Celegato, E.M.L. and De Abreu, U.P. 2002. Genetic polymorphism at the k-casein locus in Panteneiro cattle. Arch. Zootec., 51: 99-105. [ Links ]
Li, W.H., Gojobori, T. and Nei, M. 1981. Pseudogenes as a paradigm of neutral evolution. Nature, 292: 237-239. [ Links ]
Lien, S., Kaminski, S., Alestrom, P. and Rogne, S. 1993. A simple and powerful method for linkage analysis by amplification of DNA from single sperm cell. Genomics, 16: 41-44. [ Links ]
Litwiñczuk, Z. and Król, J. 2002. Polymorphism of main milk proteins in beef cattle maintained in East-Central Poland. Anim. Sci. Papers Rep., 20: 33-40. [ Links ]
Lukes, A., Barnes, M. and Pearson, R. 1989. Response to selection for milk yield and metabolic challenges in primiparous dairy cows. Dom. Anim. Endocrinol., 6: 287-293. [ Links ]
Malik, S., Sidhu, N.S., Kumar, S., Kumar, A. and Rani, R. 1997. Kappa-casein alleles in Zebu and cross-bred (1/2 Friesian, ¼ Jersey, ¼ Hariana) cattle form India using polymerase chain reaction and sequence-specific oligonucleotide probes. Genet. Anal., 14: 61-63. [ Links ]
Malveiro, E., Pereira, M., Marques, P.X., Santos, I.C., Belo, C. and Renaville, R. 2001. Polymorphisms at the five exons of the growth hormone gene in the algarvia goat: possible association with milk traits. Small Ruminant Res., 41: 163-170. [ Links ]
Martinez-Arias, R., Calafell, F., Mateu, E., Comas, D., Andres, A. and Bertranpetit, J. 2001. Sequence variability of a human pseudogene. Genome Res., 11: 1071-1085. [ Links ]
Medrano, J. and Aguilar-Córdova, E. 1990. Polymerase chain reaction amplification of bovine bLg genomic sequences and identification of genetic variants by RFLP analysis. Anim. Biotechon., 1: 73-77. [ Links ]
Medrano, J.F. and Sharrow, L. 1991. Genotyping of bovine beta-casein loci by restriction sitemodification of polymerase chain reaction (PCR) amplified genomic DNA. J. Dairy Sci., 74: 282. [ Links ]
Mellai, M., Giordano, M., D'Alfonso, S., Marchini, M., Scorza, R., Danieli, M.G., Leone, M., Ferro, I., Liguori, M., Trojano, M., Ballerini, C., Massacesi, L., Cannoni, S., Bomprezzi, R. and Momigliano-Richiardi, P. 2003. Prolactin and prolactin receptor gene polymorphisms in multiple sclerosis and systemic lupuserythematosus. Hum. Immunol., 64: 274-284. [ Links ]
Miller, S.A., Dykes, D.D. and Polesky, H.F. 1988. A simple salting out procedure for extracting DNA from human nucleated cells. Nucleic Acid. Res.,16: 12-15. [ Links ]
Mitra, A., Schlee, P., Balakrishnan, C.R. and Pirchner, F. 1995. Polymorphism at growthhormone and prolactin loci in Indian Cattle and Buffalo. J. Anim. Breed Genet., 112: 71-74. [ Links ]
Ng-Kwai-Hang, K.F., Hayes, J.F., Moxley, J.E. and Monardes, H.G. 1986. Relationships between milk protein polymorphisms and milk constituents in Holstein Friesian. J. Dairy Sci., 69: 22-26. [ Links ]
Osta, R. 1994. Caracterización genética de proteínas lácteas y sexaje de embriones en ganado vacuno mediante la aplicación de biotecnología al análisis de DNA. Universidad de Zaragoza. Zaragoza. 160 pp. [ Links ]
Osta, R., Marcos, S., Martin, I., García-Muro, E. y Zaragoza, P. 1995. Caracterización genética de proteínas lácteas en ganado vacuno mediante análisis de DNA. VI Jornadas sobre producción animal. Zaragoza. Eapaña. [ Links ]
Padilla, J., Portilla, J., Parejo, J., Corral, J., Mateos, S., Salazar, J., Izquierdo, M., Tabasco, A., Martínez-Trancón, M. y Sansinforiano, M.E. 2007. Identificación de genotipos de los loci HFABP, MC4R y LEPR en ganado porcino mediante Primer Extensión. AIDA. XII Jornadas sobre Producción Animal. ITEA, 28: 414-416. [ Links ]
Pereira, A.P., De Alencar, M.M., De Oliveira, H.N. and De Regitano, L.C. 2005. Association of GH and IGF-1 polymorphisms with growth traits in a synthetic beef cattle breed. Genet. Mol. Biol., 28: 230-236. [ Links ]
Rocha, J.L., Baker, J.F., Womackt, J.E., Sanders, J.O. and Taylor, J.F. 1992. Statistical associations between restriction fragment length polymorphisms and quantitative traits in beef cattle. J. Anim. Sci., 70: 3360-3370. [ Links ]
Sánchez, A., Betancourt, A. y Gutiérrez, C. 1977. Cromosomas del ganado Criollo. Congreso Panamericano de Veterinaria y Zoonosis. Colombia. [ Links ]
Sanz, P. 2002. El análisis de polimorfismos de ADN puntuales (SNPs) en toxicología. Rev. Toxicol., 19: 97-144. [ Links ]
Sauer, S., Lechner, D., Berlin, K., Lehrach, H., Escary, J.L., Fox, N and Gut, I.G. 2000. A novel procedure for efficient genotyping of single nucleotide polymorphisms. Nucleic. Acid. Res., 28: 13. [ Links ]
Schlee, P., Graml, R., Schallenberger, E., Schams, D., Rorhman, O., Olbrich-Blubau, A. and Pirchner, F. 1994. Growth-hormone and insulinelike growth factor I concentrations in bulls of various growth hormone genotypes. Proc. Appl. Genet., 88: 497-500. [ Links ]
Syvanen, A.C. 1999. From gels to chips: (minisequencing) primer extension analysis of point mutations and single nucleotide polymorphisms. Hum. Mutat., 13: 1-10. [ Links ]
ThidarMyint, H., Yoshida, H., Ito, T., He, M., Inoue, H. and Kuwayama, H. 2008. Combined administration of ghrelin and GHRH synergistically stimulates GH release in Holstein preweaning calves. Domes. Anim. Endocrinol., 34: 118-123. [ Links ]
Udina, I., Turkova, S., Kostyuchenko, M., Lebedeva, L. and Sulimova, G. 2001. Polymorphism of bovine prolactin gene: microsatellites, PCRRFLP. Russ. J. Genet., 37: 407-411. [ Links ]
Uffo, O., Sanz, A., Sosa, A. y Martínez, S. 2002. Identificación del polimorfismo del gen que codifica para la hormona del crecimiento bovina mediante PCR y detección de RFLP. Rev. Salud Anim., 24: 27-31. [ Links ]
Uffo, O. 2003. Aplicación de los marcadores moleculares al estudio de biodiversidad del ganado bivino cubano. Universidad Agraria de la Habana. 92 pp. [ Links ]
Uffo, O., Martín-Burriel, I., Martínez, S., Ronda, R., Osta, R., Rodellar, C. y Zaragoza, P. 2006. Caracterización genética de seis proteínas lácteas en tres razas bovinas cubanas. Agri. 39: 15-24. [ Links ]
Van der Berg, G., Escher, J., Koning, P. and Bovenhuis, H. 1990. Genetic polymorphismof kappa-casein and beta-lactoglobulin in relation to milk composition and processing properties. Neth. Milk Dairy J., 46: 145-168. [ Links ]
Recibido: 30-9-09
Aceptado: 24-2-10