Mi SciELO
Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO -
 Accesos
Accesos
Links relacionados
-
 Citado por Google
Citado por Google -
 Similares en
SciELO
Similares en
SciELO -
 Similares en Google
Similares en Google
Compartir
Archivos de Zootecnia
versión On-line ISSN 1885-4494versión impresa ISSN 0004-0592
Arch. zootec. vol.60 no.231 Córdoba sep. 2011
https://dx.doi.org/10.4321/S0004-05922011000300058
Marcadores moleculares asociados al veteado de la carne en bovinos Criollos uruguayos
Molecular markers related to marbling in Uruguayan Creole cattle
Armstrong, E.1*, Peñagaricano, F.1, Artigas, R.1, De Soto, L.1, Corbi, C.1, Llambí, S.1, Rincón, G.2 y Postiglioni, A.1
1Área Genética. Departamento de Genética y Mejora Animal. Facultad de Veterinaria. Universidad de la República. Uruguay. *eileen.armstrong@gmail.com
2Department of Animal Science. University of California. Davis. USA.
RESUMEN
En Uruguay existe una reserva genética de bovinos Criollos ubicada en el Departamento de Rocha. Estudios previos basados en marcadores moleculares altamente polimórficos mostraron que dicha población presenta una alta diversidad genética. En la década del 60 se produjo una migración de bovinos Criollos de la reserva a establecimientos ganaderos del norte del país. Este ganado fue utilizado en cruzamientos comerciales con las razas Aberdeen Angus, Hereford, Caracú y cebuinas, estando sus productos introducidos en el mercado cárnico uruguayo. Se presentan datos de la caracterización genética de la reserva y de dos poblaciones cruza (Rivera y Cerro Largo) para tres marcadores moleculares asociados al veteado de la carne: diacilglicerol acetil-transferasa (DGAT1), tiroglobulina (TG) y leptina (LEP), así como una metodología de genotipado novedosa y eficaz para el polimorFISmo del gen LEP mediante TETRA-ARMS PCR. La reserva de Criollos mostró una predominancia de los alelos y genotipos asociados a carne de bajo tenor graso en todos los marcadores. Las dos poblaciones cruza de Criollo con razas comerciales mostraron la misma tendencia para el gen DGAT1, pero mayores frecuencias de los alelos y genotipos que generan un incremento del veteado para TG y LEP. Los índices de diversidad genética resultaron de bajos a moderados (He mínima= 0,000 para DGAT1 en la población cruza de Rivera; He máxima= 0,632 para TG en la población cruza de Cerro Largo). No se observan diferencias significativas de los valores esperados para los parámetros poblacionales, según índices FIS y prueba de equilibrio Hardy-Weinberg, salvo en un caso (TG en el rodeo de Cerro Largo). Los valores de FST muestran la similitud genética entre las dos poblaciones cruza y su diferenciación con respecto a la reserva de Criollos Uruguayos puros. La introgresión de razas comerciales y los objetivos de selección en pos de una mayor productividad afectaron las frecuencias de los marcadores analizados en los rodeos cruza. Por otro lado, el aislamiento reproductivo, la ausencia de selección artificial y la incidencia de la deriva y la selección natural han moldeado los parámetros genéticos de la reserva de Criollos Uruguayos.
Palabras clave: Calidad de carne. Genética molecular. Recursos genéticos del Uruguay.
SUMMARY
The Uruguayan Creole cattle genetic reserve, located in the department of Rocha, has a high level of genetic variation, as previous studies using polymorphic markers showed. Cattle from this reserve were taken to commercial farms in the North of Uruguay during the 60'. These animals where used in crosses with commercial breeds (Aberdeen Angus, Hereford, Caracú) and zebu (Bos indicus) cattle for meat production. We analyzed a sample of Creole cattle from Rocha and from two crossbred populations from Northern Uruguay (Rivera and Cerro Largo) using three molecular markers associated with marbling: diacyl-glicerol acyl transferase (DGAT1), thyroglobulin (TG) and leptin (LEP). We describe a novel and cost-effective genotyping method based on TETRA-ARMS PCR for LEP polymorphism. The Creole cattle reserve showed higher frequencies of alleles and genotypes associated with low marbling scores in all markers. Both crossbred populations showed the same trend for DGAT1, but higher frequencies for alleles and genotypes associated with higher marbling in TG and LEP. Genetic diversity indexes were low to moderate (minimum He= 0.000 for DGAT1 in Rivera crossbred population; maximum He= 0.632 for TG in Cerro Largo crossbred population). No significant departures from the expected values were detected according to FIS and Hardy-Weinberg tests, except for one case (TG in Cerro Largo population). FST values showed the high genetic similarities between both crossbred populations and their differentiation from the purebred Uruguayan Creole reserve. Genetic introgression of commercial breeds, plus selection objectives tending to increase productivity, had affected the frequencies of the markers analyzed in the crossbred populations. On the other hand, reproductive isolation, absence of artificial selection and the incidence of genetic drift and natural selection had modelled the population parameters of the Uruguayan Creole cattle reserve.
Key words: Meat quality. Molecular genetics. Genetic resources of Uruguay.
Introducción
Los bovinos Criollos se encuentran distribuidos por todo el continente americano, estando adaptados a una gran variedad de ecosistemas. Descienden del ganado traído por los conquistadores españoles y portugueses desde el descubrimiento de América (Primo, 1992). En Uruguay existe una reserva genética de bovinos Criollos Uruguayos (Bos taurus) en el Parque Nacional de San Miguel, departamento de Rocha. Estos animales se encuentran aislados reproductivamente de otras poblaciones bovinas y no están sometidos a una rigurosa selección artificial. Estudios previos con microsatélites revelan una alta diversidad genética y bajo índice de endogamia (He>0,60; FIS= 0,02; Armstronget al., 2006), así como gran diversidad alélica en genes relacionados a producción de leche y derivados (Rincón et al., 2006). En la década de 1960 se produjo una migración de algunos de estos animales a establecimientos ganaderos del norte del país de los Departamentos de Cerro Largo y Rivera, en zonas fronterizas con Brasil. Este ganado Criollo fue utilizado en cruzamientos comerciales con las razas Aberdeen Angus, Hereford, Caracú y cebuinas, estando sus productos introducidos en el mercado cárnico uruguayo. Estudios con microsatélites identifican valores similares de diversidad genética e índices de consanguinidad a los hallados en la reserva de Criollos puros (Postiglioni et al., 2007).
El marmolado o veteado de la carne es de gran importancia en la industria cárnica, determinante de la calidad del producto, pero difícil de medir y de seleccionar por los métodos clásicos. Por este motivo la detección de marcadores moleculares asociados a esta característica puede asistir en la selección de reproductores. Grisartet al. (2004) identifican un polimorfismo en el gen bovino DGAT1 que genera dos variantes alélicas para la enzima diacil-glicerol acetil transferasa: alelo A (alanina en ubicación 232 del polipéptido) y alelo K (lisina en la posición 232). El alelo K hace que la enzima sintetice más triglicéridos en menos tiempo que el alelo A, generando más grasa intramuscular. El polimorfismo responsable del cambio aminoacídico es la sustitución de dos nucleótidos (AA por GC) en el exón VIII de DGAT1 (node acceso AY065621, posiciones 6829-30; Winter et al., 2002; Thaller et al., 2003).
Por otro lado, el gen TG codifica para la tiroglobulina, también asociada con los niveles de deposición de grasa intramuscular. Este gen presenta un polimorFISmo de nucleótido simple (SNP) en un elemento repetitivo corriente arriba del promotor que ha sido asociado a variaciones en los niveles del veteado en ganado vacuno y se incluye en pruebas comerciales de ADN. Una sustitución de una citosina por una timina da lugar a dos alelos: C y T (posición 422 del no de acceso GenBank X05380). Los animales heterocigotas u homocigotas para el alelo T (CT o TT) presentan mayores registros de veteado que los homocigotas para el alelo C (Barendse et al., 2004; Casas et al., 2005; Van Eenennaam et al., 2007).
El gen de la leptina (gen LEP) está involucrado en el metabolismo lipídico, afectando la deposición de grasa en la carne y por tanto asociado también a variaciones del veteado. Existe una sustitución de citosina por timina en el exón 2 del gen que genera un cambio aminoacídico de arginina por cisteína en la proteína resultante, estando el alelo T asociado a un mayor contenido de grasa (node acceso del SNP rs29004488; Buchanan et al., 2002; Soria y Corva, 2004; Schenkel et al., 2005). En este trabajo, se presenta además, un protocolo novedoso, sencillo y de bajo costo para el genotipado del SNP mencionado del gen de la leptina.
Con el propósito de analizar el potencial genético para producción de carne con mayor o menor veteado en el Criollo Uruguayo puro y en las poblaciones de bovinos comerciales con introgresión de bovino Criollo, se realizó el genotipado de estos marcadores asociados a esta característica de importancia en la industria cárnica.
Material y métodos
Se analizaron muestras de bovinos Criollos Uruguayos puros provenientes de la reserva genética de Rocha (población total: 575 animales) y de dos poblaciones de bovinos comerciales con introgresión de Criollo: Cerro Largo (Establecimiento San Joaquín, población total: 661 animales) y Rivera (Establecimiento Guaviyú, población total: 98 animales). El número de muestras analizadas por población es variable para cada gen.
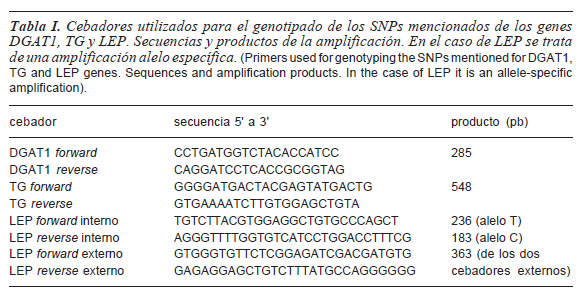
Las muestras fueron extraídas al azar y se obtuvo ADN a partir de sangre y folículo piloso con protocolos estándar. Los genes DGAT1 y TG se analizaron mediante PCRRFLP, utilizando las enzimas de restricción Mwo I (Rincón et al., 2006) y Psu I (Thaller et al., 2003), respectivamente. El protocolo de amplificación utilizado incluye 2,5 μl de buffer de PCR 10X, 1,5 μl de MgCl2 25 mM, 1,3 μl de DMSO, 1,0 μl de dNTPs 10 mM, 1,0 μl de cada cebador 10 pmol, 0,2 μl de Taq polimerasa 5 UI, más 10 μl de ADN genómico, en un volumen final de 25 μl. Los cebadores utilizados se presentan en la tabla I. El ciclado incluye un ciclo de desnaturalización inicial a 95oC durante 5 minutos, luego 35 ciclos de amplificación (fase de desnaturalización a 94oC por 30 segundos, fase de hibridación a 60oC por 30 segundos y fase de extensión a 72oC por 30 segundos) y un ciclo final de extensión a 72oC por 30 minutos. Para la posterior digestión con la enzima de restricción se utilizaron 5 μl del producto de PCR, 1,0 μl de buffer 10X, 1,0 μl de enzima de restricción 10 U/μl y 8,0 μl de agua esterilizada. El volumen final de 15 μl fue incubado a 37oC por espacio de 16 horas o toda la noche en el caso de la enzima Psu I (gen TG) y a 60oC por 2 horas en el caso de Mwo I (gen DGAT1).
Para el análisis del SNP mencionado del exón 2 del gen LEP se utilizó la técnica de TETRA-ARMS PCR (Tetra-primer Amplification Refractory Mutation System PCR) mediante un protocolo diseñado y puesto a punto en nuestro laboratorio. Este sistema se basa en la utilización de cuatro cebadores, dos externos que circunscriben la amplificación a la región génica donde se encuentra la mutación y dos internos que generan un segundo amplicón de tamaño variable según con cuál alelo, C o T, haya hibridado su extremo 3'. Para aumentar la especificidad de la reacción se introduce un cambio de base (mismatch) entre la primera y la tercera posición previas al extremo 3' de los cebadores internos. Ambos alelos se amplifican simultáneamente y son luego identificados por sus tamaños en electroforesis en gel (Ye et al., 2001). Las condiciones de PCR fueron las siguientes: 2,5 μl de buffer 10X, 3,0 μl de MgCl2 25mM, 1,0 μl de solución de DNTP's 10 mM, 1,3 μl de DMSO, 1,0 μl de solución 10 pmol de cada cebador interno, 0,5 μl de solución 10 pmol de cada cebador externo, 0,2 μl de Taq 5U/μl, 1 μl de DNA 50 ng/μl, en un volumen final de 25 μl. El ciclado es un touchdown con una temperatura de hibridación de 64oC para el primer ciclo, disminuyendo 1oC cada dos ciclos hasta 60oC, seguidos por 27 ciclos a 60oC. Los cebadores externos e internos utilizados fueron diseñados en base a la secuencia del gen de la leptina bovina (número de acceso Ensembl: ENSBTAG00000014911) y se presentan en la tabla I. La exactitud de los genotipos detectados fue confirmada mediante secuenciación automática de tres animales correspondientes a los tres genotipos posibles (CC, CT y TT), que habían sido genotipados previamente con el método que aquí se describe.
Las variantes alélicas fueron identificadas mediante corridas electroforéticas en geles de agarosa al 3% teñidos con bromuro de etidio y visualizados mediante luz UV.
Se calcularon frecuencias alélicas y genotípicas, heterocigosidad observada y esperada, estadísticos F (Weir y Cockerham, 1984) y prueba de equilibrio Hardy-Weinberg utilizando los programas Convert (Glaubitz, 2004) y Genepop v4 (Rousset, 2008).
Resultados
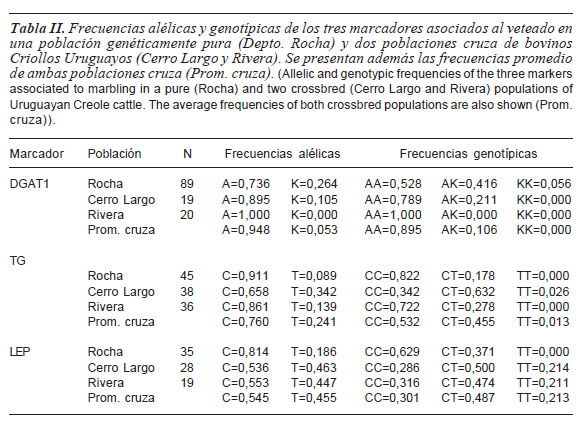
Las muestras analizadas y las frecuencias alélicas y genotípicas detectadas se detallan en la tabla II. La heterocigosidad observada y esperada se presentan en la tabla III. Los estadísticos F (índices FIS y FST) se presentan en la tabla IV. Los resultados de la prueba de equilibrio Hardy-Weinberg se detallan en el texto y no se muestran en las tablas.
Para el gen DGAT1, las tres muestras analizadas revelan una frecuencia mucho más elevada del alelo A y del genotipo AA, asociados a una menor proporción de grasa intramuscular. En la población de Rivera el alelo A se encuentra fijado. En el caso de la reserva de Criollos puros de Rocha se observa una mayor paridad de frecuencias de los genotipos AA y AK. Estos resultados determinan bajos índices de heterocigosidad en las poblaciones cruza, y niveles medios en la de Criollos puros. Los índices FIS son bajos y muestran una leve tendencia hacia el exceso de heterocigotas en las poblaciones en las que pudo ser calculado (Rocha y Cerro Largo), sin que eso genere desviaciones de lo esperado para el equilibrio Hardy-Weinberg (p>0,05 en ambos casos). Los índices FST reflejan la mayor diferencia en las frecuencias alélicas entre las poblaciones de Rivera y de Rocha, siendo para este gen las más distantes entre sí.
En el caso del gen TG se observa predominancia del alelo C, asociado a carne magra, en todos los rodeos y con frecuencias cercanas a la fijación en la población de Criollos puros. En los rodeos cruza, especialmente en el de Cerro Largo, la frecuencia del alelo T es mayor, con una frecuencia más importante de heterocigotas CT. Debido a esto, la heterocigosidad es mayor en Cerro Largo, habiendo una diferencia notoria entre los valores esperados y observados. Dicha diferencia se refleja en el índice FISpara ese rodeo, con un valor alto y negativo, que genera una desviación significativa de lo esperado para el equilibrio Hardy-Weinberg (p= 0,016 cuando la hipótesis alternativa es el exceso de heterocigotas y p= 0,027 en el test de probabilidad). En las poblaciones de Rocha y Rivera la heterocigosidad es menor, los índices FIS son bajos a medios y no se observan desvíos de lo esperado bajo una situación de equilibrio génico (p>0,05). Las mayores diferencias se observan entre las poblaciones de Rocha y Cerro Largo, lo cual se refleja en un índice FST más alto entre estos dos rodeos.
En la muestra de Criollos puros identificamos una frecuencia muy elevada del alelo C y del genotipo CC, asociados a carne de bajo tenor graso, para el gen de la leptina. La frecuencia del genotipo CT es importante, pero no se observan individuos homocigotas TT. Debido a esto, los valores observados y esperados de la heterocigosidad, así como del FIS, muestran un exceso de heterocigotas para este polimorFISmo. Sin embargo, esta diferencia no es suficiente para generar desvíos significativos de lo esperado en equilibrio Hardy-Weinberg (p>0,05 en todos los tests). Por otro lado, en las poblaciones cruza de Criollo con razas comerciales de carne observamos una mayor paridad en las frecuencias alélicas y un mayor número de heterocigotas CT para este gen. Los valores de heterocigosidad son medios, más elevados que en el caso del Criollo puro, y los índices FIS son cercanos a cero. No se observan diferencias significativas con lo esperado en el equilibrio génico (p>0,05 en todos los casos). Los valores de FST reflejan la mayor similitud observada entre las dos poblaciones de Criollos cruza que entre cualquiera de ellas con el rodeo de Criollos puros.
Discusión
En este trabajo se presentan las frecuencias génicas, así como otros parámetros poblacionales, de una población de Criollos Uruguayos y dos poblaciones de Criollos con introgresión de razas comerciales, principalmente británicas, para genes relacionados con el metabolismo lipídico. Los genes analizados, DGAT1, TG y LEP, han probado estar asociados a la cantidad de grasa intramuscular en trabajos previos de otros autores (Buchanan et al., 2002; Thaller et al., 2003; Barendse et al., 2004), siendo el veteado una característica fundamental en la determinación de la calidad de la carne vacuna. El análisis poblacional es un paso previo fundamental en caso de que se deseen utilizar polimorFISmos genéticos en selección asistida por marcadores.
La técnica desarrollada en nuestro laboratorio para el genotipado del polimorFISmo del gen LEP mostró ser altamente efectiva, habiendo coincidido plenamente con los datos obtenidos mediante la secuenciación de esa porción del gen. Al no requerir enzimas de restricción, el genotipado es más sencillo, rápido y de bajo costo.
Los resultados obtenidos para el gen DGAT1 muestran una clara predominancia del alelo y genotipos asociados con una menor proporción de grasa y coinciden con lo hallado previamente en una muestra menor de Criollos Uruguayos puros (Rincón et al., 2006). Esta tendencia es la usual en razas de Bos taurus, a diferencia de lo que ocurre en razas cebuinas y en razas lecheras seleccionadas para un mayor contenido graso en la leche, como Jersey, en las cuales se observa una muy alta frecuencia del alelo K (Thaller et al., 2003; Kaupe et al., 2004; Casas et al., 2005; Gill et al., 2009). Las frecuencias halladas en la reserva son muy similares a las reportadas previamente para las razas españolas Toro de Lidia y Asturiana de la Montaña (Kaupe et al., 2004). Otros autores encuentran frecuencias más equilibradas para ambos alelos en poblaciones de bovinos Criollos de Argentina y Bolivia, siendo una de las posibles causas la introgresión de Bos indicus (Lirón et al., 2002; Ripoli et al., 2006). Si bien puede existir un bajo nivel de introgresión de razas cebuinas en las poblaciones cruza analizadas aquí, su influencia en los resultados obtenidos no es evidente. En la reserva de Criollos Uruguayos no se ha detectado introgresión de Bos indicus en estudios citogenéticos y moleculares previos (Postiglioni et al., 2002).
En cuanto a los resultados para el polimorFISmo del gen TG, las frecuencias detectadas en los tres rodeos son similares a las halladas en razas británicas y europeas continentales (Thaller et al., 2003; Casas et al., 2007; Van Eenennaam et al., 2007). En este caso, la reserva de Criollos de Rocha es la que presenta las frecuencias más extremas, estando el alelo C muy cercano a la fijación. Dado que el tamaño poblacional de la reserva es reducido y ha sufrido cuellos de botella en el pasado, la deriva génica puede haber generado esta tendencia. Por otro lado, el rodeo de Cerro Largo presenta frecuencias más similares de ambos alelos. El exceso de heterocigotas observado en esta población y el desequilibrio generado puede deberse a eventos recientes de cruzamientos entre razas distintas. La tendencia hacia un mayor porcentaje del alelo T en las dos poblaciones comerciales, favorable a un aumento del veteado, puede ser provocada por los efectos de la selección tendiente a la produccion de carne de mejor calidad. Las investigaciones en este marcador y su efecto real sobre los niveles de engrasa-miento no son concluyentes, habiendo resultados conflictivos en cuanto a la validez de su uso en selección asistida por marcadores. Rincker et al. (2006) no detectan asociación entre este polimorfismo y la cantidad de grasa intramuscular en novillos Simmental; Van Eenenaam et al. (2007) tampoco detectan asociación con el nivel de veteado en varias razas, aunque sí con una tendencia al aumento del grado de calidad de la carne; Casas et al. (2007) sólo hallan una asociación significativa con el veteado en ganado Wagyu. La tendencia a la fijación del alelo C en casi todas las razas estudiadas no contribuye a dilucidar este problema y pone en evidencia la necesidad de efectuar estudios poblacionales previos a la posible aplicación de los marcadores moleculares en producción animal.
Para el SNP analizado del gen LEP, las frecuencias alélicas y genotípicas en ambos rodeos del norte del país resultaron semejantes a las descriptas para razas comerciales, las cuales presentan porcentajes similares de ambos alelos (Buchanan et al., 2002; Schenkel et al., 2005; Gill et al., 2009). Estos resultados pueden explicarse por la selección indirecta a favor del alelo que genera aumento de los depósitos de grasa (alelo T), y por ende mayor ganancia de peso, lo cual es un objetivo de selección permanente en producción de carne. La reserva de Criollos Uruguayos puros se aparta claramente de esta tendencia, mostrando una frecuencia muy elevada del alelo C. Al igual que ocurre con el polimorfismo del gen TG, los resultados obtenidos en esta población reflejan la ausencia de selección artificial y posibles efectos de la deriva génica. La concordancia de los datos obtenidos en los tres rodeos con los resultados esperados según su evolución y composición racial ratifica la técnica de genotipado utilizada.
Al igual que la heterocigosidad, los índices FIS fueron en general moderados o bajos. Esta situación es usual en los marcadores dialélicos en ausencia de eventos que desvíen las frecuencias observadas de lo esperado bajo la hipótesis de equilibrio Hardy-Weinberg.
Los valores de FST entre las poblaciones reflejan las tendencias descriptas para cada gen. Del análisis global de FST tomando en cuenta todos los marcadores surge que los rodeos de Criollos cruza son más semejantes entre sí que cualquiera de ellos con la reserva de Criollos puros de Rocha. La relación genética entre las tres poblaciones se reduce a la introgresión de animales de la reserva en estos rodeos comerciales en la década de 1960. La composición genética y los objetivos de selección compartidos por ambas poblaciones comerciales las hacen más semejantes, mientras que la ausencia de selección artificial, el tiempo transcurrido y la deriva génica han aumentado la distancia genética de la reserva con las poblaciones cruza.
Resulta interesante observar qué ocurre con estos marcadores relacionados con características productivas en una población que nunca ha sido sometida a selección artificial, como es el caso de la reserva de Rocha. En el gen DGAT1, el alelo A también se asocia a un bajo contenido de grasa en la leche, lo cual a su vez está relacionado a un aumento en la actividad folicular de los ovarios que redunda en una mayor fertilidad de la hembra (Lucy et al., 1992; Kaupe et al., 2004). Como la producción de leche con bajo nivel de grasa insume un menor gasto energético, estos dos factores combinados podrían generar una ventaja selectiva natural para los portadores del alelo A, especialmente para poblaciones de ganado que han sobrevivido bajo condiciones naturales sin prácticamente ningún tipo de manejo. La ausencia de selección tendiente a un mayor aumento de peso, sumado a la baja o nula introgresión de razas comerciales, británicas o cebuinas, no ha interferido en las frecuencias de los tres SNP analizados.
Los resultados muestran el potencial genético del Criollo Uruguayo, que puede orientarse hacia la producción de carne de bajo tenor graso, apta para mercados que exigen un producto más saludable. También subrayan la importancia de estudios poblacionales previos a la implementación de selección asistida por marcadores. Estos estudios, junto con el análisis de la validez de los marcadores en los distintos sistemas de producción, son fundamentales para determinar el alcance y eficacia de posibles planes de mejora genética que utilicen marcadores moleculares (Ibeagha-Awemuet al., 2008; Neuner et al., 2009). El análisis fenotípico y de otros marcadores relacionados con la calidad de la carne en estas poblaciones permitirá evaluar el potencial productivo del bovino Criollo Uruguayo y sus cruzas, en el marco de la caracterización y conservación de los recursos genéticos animales mundiales.
Agradecimientos
Los autores agradecen a la Sra. Iris Hernández por su colaboración técnica en el Laboratorio del Área Genética, al proyecto No 156 Fondo Clemente Estable-PDT por la financiación y al Área Genética de la Facultad de Veterinaria por su financiación parcial y apoyo en equipamiento.
Bibliografía
Armstrong, E., Postiglioni, A., Martínez, A., Rincón, G. and Vega-Pla, J.L. 2006. Microsatellite analysis of a sample of Uruguayan Creole bulls (Bos taurus). Genet. Mol. Biol., 29: 267-272. [ Links ]
Barendse, W., Bunch, R., Thomas, M., Armitage, S., Baud, S. and Donaldson, N. 2004. The TG5 thyroglobulin gene test for a marbling quantitative trait loci evaluated in feedlot cattle. Aust. J. Exp. Agr., 44: 669-674. [ Links ]
Buchanan, F.C., Fitzsimmons, C.J., Van Kessel, A.G., Thue, T.D., Winkelman-Sim, D.C. and Schmutz, S.M. 2002. A missense mutation in the bovine leptin gene is correlated with carcass fat content and leptin mRNA levels. Genet. Sel. Evol., 34: 1-12. [ Links ]
Casas, E., White, S.N., Riley, D.G., Smith, T.P.L., Brenneman, R.A., Olson, T.A., Johnson, D.D., Coleman, S.W., Bennett, G.L. and Chase, C.C. 2005. Asessment of single nucleotide polymorphisms in genes residing on chromosomes 14 and 29 for association with carcass composition traits in Bos indicus cattle. J. Anim. Sci., 83: 13-19. [ Links ]
Casas, E., White, S.N., Shackelford, S.D., Wheeler, T.L., Koohmaraie, M., Bennett, G.L. and Smith, T.P.L. 2007. Assessing the association of single nucleotide polymorphisms at the thyroglobulin gene with carcass traits in beef cattle. J. Anim. Sci., 85: 2807-2814. [ Links ]
Gill, J.L., Bishop, S.C., McCorquodale, C., Williams, J.L.and Wiener, P. 2009. Association of selected SNP with carcass and taste panel assessed meat quality traits in a commercial population of Aberdeen Angus-sired beef cattle. Genet. Sel. Evol., 41: 36. [ Links ]
Glaubitz, J. 2004. Convert: a user-friendly program to reformat diploid genotypic data for commonly used population genetic software packages. Mol. Ecol. Notes, 4: 309-310. [ Links ]
Grisart, B., Farnir, F., Karim, L., Cambisano, N., Kim, J., Kvasz, A., Mni, M., Simori, P., Frere, J., Coppieters, W. and Georges, M. 2004. Genetic and functional confirmation of the causality of the DGAT1 K232A quantitative trait nucleotide in affecting milk yield and composition. Proc. Nat. Acad. Sci., 101: 2308-2403. [ Links ]
Ibeagha-Awemu, E., Kgwatalala, P. and Zhao, X. 2008. A critical analysis of productionassociated DNA polymorphisms in the genes of cattle, goat, sheep and pig. Mamm. Gen., 19: 591-617. [ Links ]
Kaupe, B., Winter, A., Fries, R. and Erhardt, G. 2004. DGAT1 polymorphism in Bos indicus and Bos taurus cattle breeds. J. Dairy Res., 71: 182-187. [ Links ]
Lirón, J.P., Ripoli, M.V., De Luca, J.C., Peral-García, P. and Giovambattista, G. 2002. Analysis of genetic diversity and population structure in Argentine and Bolivian Creole cattle using five loci related to milk production. Genet. Mol. Biol., 25: 413-419. [ Links ]
Lucy, M.C., Savio, J.D., Badinga, R.L., De la Sota, R.L. and Thatcher, W.W. 1992. Factors that affect ovarian follicular dynamics in cattle. J. Anim. Sci., 70: 3615-26. [ Links ]
Neuner, S., Edel, C., Emmerling, R., Thaller, G. and Götz, K. 2009. Precision of genetic parameters and breeding values estimated in marker assisted BLUP genetic evaluation. Genet. Sel. Evol., 41: 26. [ Links ]
Primo, A.T. 1992. El ganado bovino ibérico en las Américas: 500 años después. Arch. Zootec., 41: 421-432. [ Links ]
Postiglioni, A., Rincón, G., Kelly, L., Llambí, S., Fernández, G., D'Angelo, M., Gagliardi, R., Trujillo, J., Bethencourt, M. de, Guevara, K., Castellano, A. y Arruga, M.V. 2002. Biodiversidad genética en bovinos Criollos del Uruguay. Análisis con marcadores moleculares. Arch. Zootec., 51: 18. [ Links ]
Postiglioni, A., Armstrong, E., Peñagaricano, F., Caffaro, M.J., Iriarte, A., Artigas, R., Barbieri, G., Camio, G. y Rincón, G. 2007. Caracterización genética de dos poblaciones de bovinos de carne con introgresión de Criollos uruguayos. V Jornadas Técnicas de la Facultad de Veterinaria. UDELAR. 25 pp. [ Links ]
Rincker, C., Pyatt, N., Berger, L. and Faulkner, D. 2006. Relationship among GeneSTAR marbling marker, intramuscular fat deposition, and expected progeny differences in early weaned Simmental steers. J. Anim. Sci., 84: 686-693. [ Links ]
Rincón, G., Armstrong, E. and Postiglioni, A. 2006. Analysis of population structure in Uruguayan Creole cattle inferred from milk major gene polymorphisms. Genet. Mol. Biol., 29: 491-495. [ Links ]
Ripoli, M.V., Corva, P. and Giovambattista, G. 2006. Analysis of a polymorphism in the DGAT1 gene in 14 cattle breeds through PCR-SSCP methods. Res. Vet. Sci., 80: 287-290. [ Links ]
Rousset, F. 2008. Genepop v4: a complete reimplementation of the genepop software for Windows and Linux. Mol. Ecol. Res., 8: 103-106. [ Links ]
Soria, L.A. y Corva, P.M. 2004. Factores genéticos y ambientales que determinan la terneza de la carne bovina. Arch. Lat. Prod. Anim., 12: 73-88. [ Links ]
Schenkel, F.S., Miller, S.P., Ye, X., Moore, S.S., Nkrumah, J.D., Yu, C.L., Mandell, I.B., Wilton, J.W. and Williams, J.L. 2005. Association of single nucleotide polymorphisms in the leptin gene with carcass and meat quality traits of beef cattle. J. Anim. Sci., 83: 2009-2020. [ Links ]
Thaller, G., Kühn, C., Winter, A., Ewald, G., Bellmann, O., Wegner, J., Zühlke, H. and Fries, R. 2003. DGAT1, a new positional and functional candidate gene for intramuscular fat deposition in cattle. Anim. Genet., 34: 354-357. [ Links ]
Van Eenennaam, A.L., Li, J., Thallman, R.M., Quaas, R.L., Dikeman, M.E., Gill, C.A., Franke, D.E. and Thomas, M.G. 2007. Validation of commercial DNA tests for quantitative beef quality traits. J. Anim. Sci., 85: 891. [ Links ]
Weir, B.S. and Cockerham, C.C. 1984. Estimating F statistics for the analysis of population structure. Evolution, 38: 1358-1370. [ Links ]
Winter, A., Kramer, W., Werner, F.A.O., Kollers, S., Kata, S., Durstewitz, G., Buitkamp, J., Womack, J.E., Thaller, G. and Fries, R. 2002. Association of a lysine-232/alanine polymorphism in a bovine gene encoding acyl CoA: diacylglycerol acyltransferase DGAT1 with variation at a quantitative trait locus for milk fat content. Proc. Nat. Acad. Sci., 99: 9300-9305. [ Links ]
Ye, S., Dhillon, S., Ke, X., Collins, A. and Day, I. 2001. An efficient procedure for genotyping single nucleotide polymorphisms. Nucl. Acids Res., 29: e88. [ Links ]
Recibido: 15-4-09
Aceptado: 29-3-10