Meu SciELO
Serviços Personalizados
Journal
Artigo
Indicadores
-
 Citado por SciELO
Citado por SciELO -
 Acessos
Acessos
Links relacionados
-
 Citado por Google
Citado por Google -
 Similares em
SciELO
Similares em
SciELO -
 Similares em Google
Similares em Google
Compartilhar
Archivos de Zootecnia
versão On-line ISSN 1885-4494versão impressa ISSN 0004-0592
Arch. zootec. vol.63 no.244 Córdoba Dez. 2014
https://dx.doi.org/10.4321/S0004-05922014000400010
MiPoB: un programa de simulación para el aprendizaje en mejora genética animal
MiPoB: a simulation program for learning in animal breeding
Alfonso, L.
Escuela Técnica Superior de Ingenieros Agrónomos. Universidad Pública de Navarra. Campus Arrosadia. Pamplona. España. leo.alfonso@unavarra.es
RESUMEN
Se ha desarrollado un programa de simulación de apoyo a la docencia de los cursos introductorios a la Mejora genética animal. El programa surgió ante la ausencia de este tipo de herramientas docentes orientadas a estudiantes de primeros cursos de grado universitario, especialmente en lengua española. El programa simula una población animal cerrada de censo reducido en la que los estudiantes deben de ir tomando las decisiones de elección de futuros reproductores y cómo aparearlos. De esta forma se facilita la introducción de los conceptos básicos sobre selección y evaluación genética, y conservación y genética de poblaciones. El programa considera el caso particular del vacuno de carne y se pueden realizar hasta un total de diez generaciones de selección. Tras haber sido probado durante dos cursos consecutivos en enseñanza reglada universitaria, está disponible de forma gratuita para su libre utilización y distribución.
Palabras clave: Genética. Selección. Docencia. Software. Simulación.
SUMMARY
A simulation program has been developed to support the teaching of introductory courses in animal breeding. The program was conceived because the absence of this kind of educational tool aimed at students of first university degree courses, especially in Spanish language. The program simulates a closed animal population of small size in which students should take decisions of choice and mating of future parental. This facilitates the introduction of the basic concepts of selection and genetic evaluation, and also of conservation and population genetics. The program considers the particular case of beef cattle and a total of ten generations of selection can be simulated. Having been tested for two consecutive courses in university formal education, it is available for free use and distribution.
Key words: Genetics. Selection. Teaching. Software. Simulation.
Introducción
Al igual que en cualquier otra disciplina existen distintos métodos y actividades para afrontar el aprendizaje y la enseñanza de la mejora genética animal, pero la simulación mediante ordenador es especialmente atractiva dado el inconveniente, entre otros (Carlson, 1985), de la imposibilidad de realizar experimentos reales de selección genética con animales ganaderos en el breve tiempo que ocupa una asignatura o curso. La selección es un proceso lento de cambio de las características genéticas de las poblaciones animales, que sólo a medio-largo plazo cobra interés dado el carácter acumulativo de los cambios producidos generación tras generación.
La simulación no es una herramienta docente nueva, ni siquiera reciente. Heidhues y Henderson (1961), y Willham (1968) ya propusieron la utilización de la simulación como herramienta docente y de extensión, para entender las claves de la evaluación genética en ganado vacuno sin tener necesariamente que disponer de unos conocimientos estadísticos muy avanzados. Así, se desarrollaron algunos programas informáticos que con el tiempo se fueron adaptando para su uso en ordenadores personales (Buchanan et al., 1988), o más recientemente a través de internet (Medrano et al., 2010).
Desde el inicio se constató que la utilización de estas herramientas permitía aumentar el interés, la comprensión, la aplicación y el conocimiento de la mejora genética animal (DeRouen et al., 1989), y así ha sido personalmente después de haber utilizado durante varios años Cowgame (Buchanan et al., 1988) en el desarrollo de distintas asignaturas y cursos sobre mejora genética animal. Los contenidos de los cursos de Mejora genética animal, así como las formas de estudio y aprendizaje de los estudiantes universitarios han ido variando, y actualmente es necesario disponer de herramientas que se adecuen mejor a los objetivos docentes, las competencias a adquirir por parte de los alumnos, su forma de estudiar, y a las actividades docentes a desarrollar.
Para el desarrollo de los primeros cursos introductorios a la Mejora genética animal hay otros programas de apoyo a la docencia, como Genup (Kinghorn, 1992) o PQGen (Altarriba etal., 2010), que también resultan de gran utilidad, permitiendo ir más allá de los objetivos iniciales de los primeros programas de simulación, basados en entender y familiarizarse con la evaluación genética de reproductores, y así introducir otros contenidos importantes de genética de poblaciones y genética cuantitativa. No obstante, el planteamiento de los contenidos, y su funcionamiento, no obliga a la toma de decisiones continua y autónoma por parte del alumno, ni a la asunción de las consecuencias de las decisiones tomadas, lo cual le aleja en cierto modo de la reflexión sobre los resultados que se van alcanzando en un rebaño sometido a selección.
En este contexto, se pensó en desarrollar un programa (MiPob) que reuniera las siguientes características: 1) fuera sencillo de utilizar y no precisara un excesivo conocimiento previo de las bases teóricas de la genética y la selección animal; 2) lo pudiesen utilizar de forma gratuita usuarios hispanoparlantes, sin grandes necesidades de equipamiento informático, en el entorno habitual MS-Windows, y sin necesidad de conexión a internet; 3) ayudase a entender el interés, las limitaciones y las consecuencias de seleccionar una población animal de censo reducido como estrategia de mejora genética, a través de la simulación de una población y la toma de decisiones -por parte del alumno- de cómo realizar los apareamientos entre animales; 4) ayudase a introducir en los cursos de mejora, si así se considerara, no sólo el concepto de valor genético de los animales, su predicción y la precisión de esas predicciones, si no también otros conceptos como la deriva genética, la consanguinidad, el parentesco entre animales, la depresión consanguínea de las producciones, los apareamientos dirigidos, la respuesta genética a la selección, el tamaño efectivo de población, etc.
Material y métodos
PROCESO DE SIMULACIÓN
Para el desarrollo del programa se consideró un rebaño de vacuno de carne con las siguientes características:
1) Inicialmente está formado por 5 toros y 50 vacas, es decir se considera una población de reducido tamaño. El número de hembras debe mantenerse constante a lo largo de las generaciones, pero el número de machos se puede reducir a un mínimo de dos.
2) La población se considera genéticamente singular frente a otras poblaciones de vacuno por ser portadora de 5 alelos singulares, en 5 loci autosómicos, dialélicos, y ubicados en distintos cromosomas (es decir independientes), con distintas frecuencias (por defecto cuatro de ellos presentes en baja frecuencia, 0,05, y otro a frecuencia intermedia, 0,5).
3) El objetivo productivo de la población es el peso al sacrificio (peso al año asumiendo que los animales se sacrifican con una edad de 12 meses). Ese carácter tiene por defecto, en el programa, unos valores de 480 kg de media, 50 kg de desviación típica, y una heredabilidad de 0,35 (por simplicidad, no se consideran diferencias en el crecimiento de machos y hembras).
4) En cada generación padres e hijos pueden ser candidatos a la selección, es decir escogidos como futuros reproductores para formar la siguiente generación. Se considera un intervalo entre partos de 24 meses y una edad a la primera cubrición de 15 meses. Aunque el intervalo entre partos es demasiado largo, y la primera cubrición algo temprana, se facilita la simulación, al poder escoger simultáneamente a padres e hijos. El intervalo generacional mínimo resultante es de 36 meses.
5) En todos los animales se controla el parentesco (padre y madre, asumiendo que no hay errores de asignación). En base a toda la información genealógica disponible desde la creación de la población se calculan los coeficientes de consanguinidad y parentesco de los animales.
6) La fertilidad del rebaño tiene un valor inicial por defecto del 85 % (aunque se puede modificar), y la probabilidad de sexos al nacimiento está fijada en un 50 %. La fertilidad se ve afectada negativamente por el aumento de la consanguinidad, reduciéndose en un determinado porcentaje por cada 10 % de aumento de la consanguinidad (por defecto un 2 %). Por simplicidad, esa disminución de la fertilidad no afecta al número de partos, es decir se asume que se ve compensada por un aumento del número de cubriciones.
7) En la población se produce cada generación un determinado porcentaje de bajas de animales (por defecto un 5 %). Si la baja se produce antes del año de edad quiere decir que no se dispondrá del dato de peso de ese animal, pues el control se realiza al año. Por simplicidad el porcentaje de bajas se considera independiente a la edad, y no se establece una longevidad máxima de los animales.
8) El valor genético de los animales para el peso al sacrificio se predice en función del peso individual de cada animal y del de todos sus descendientes. Al hacer la predicción del valor genético de los animales, se calcula también su precisión (fiabilidad).
9) Se pueden realizar un máximo de 10 generaciones de selección.
SIMULACIÓN DE VALORES GENÉTICOS Y FENOTÍPICOS
El fenotipo para el carácter Peso al sacrificio se simula, para el animal i, según la expresión:
Pi=μ+Gi+Ei
siendo:
μ= la media del carácter (480 por defecto)
Ei= la componente no heredable del carácter que se simula según una variable normal de media 0 y desviación típica σe (20,16 por defecto) [N(0,σ2e)]
Gi= componente genética heredable del carácter (valor genético) que se simula según distintas expresiones:
- para los animales de la población inicial según una variable normal de media 0 y desviación típica σa (14,79 por defecto) [N(0,σ2a)]
- para los animales de las siguientes generaciones como la semisuma del valor genético de sus padres (Gp y Gm) más una componente de muestreo mendeliano(Φi), es decir: Gi = [(Gp+Gm)/2] + Φi , donde la componente de muestreo mendeliano (Φi) se obtiene teniendo en cuenta el coeficiente de consanguinidad de padre (Fp) y madre (Fm), a partir de una variable aleatoria con distribución normal de media 0 y desviación típica (1/4).(1-((Fp+Fm)/2))(1/2).σa (ver p.ej. Sorensen y Kennedy, 1984).
Cada uno de los cinco genes singulares se simula inicialmente para cada animal de la población base. Se asigna aleatoriamente uno de los dos alelos posibles a cada uno de los cromosomas de acuerdo a las frecuencias alélicas consideradas. Para las siguientes generaciones, a cada animal se le asigna aleatoriamente uno de los dos alelos de cada parental, y se calcula la frecuencia de cada alelo en la población mediante conteo simple.
PREDICCIÓN DEL VALOR GENÉTICO
El valor genético de los animales se calcula teniendo en cuenta el fenotipo propio y el de todos sus hijos mediante un índice de selección (ver p.ej. Van Vleck et al., 1987). Asumiendo que no existen factores ambientales no aleatorios en el rebaño (tal como se simula), el cálculo se realiza mediante la siguiente expresión:
EBV = bi (Pi - Pm) + bh·(Ph-Pm)
donde:
EBV = Valor genético predicho (Expected breeding value)
Pi = Peso al sacrificio del animal
Ph = Media del peso al sacrificio de todos sus hijos
Pm = Media del peso al sacrifico de todos los animales de la población
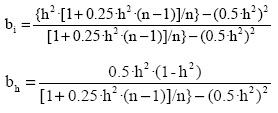
siendo h2 la heredabilidad, y n el número de hijos con registros.
La precisión de la predicción de esos valores genéticos se calcula según la expresión:

Aunque se podía haber considerado una evaluación genética basada en el modelo animal y su correspondiente resolución BLUP, se optó por la utilización de un índice de selección simple basado en regresión múltiple para facilitar la comprensión a estudiantes sin conocimientos estadísticos del modelo mixto.
CÁLCULO DE LOS COEFICIENTES DE CONSANGUINIDAD Y PARENTESCO
El cálculo de los coeficientes de consanguinidad y parentesco se realiza siguiendo el método tabular (ver p.ej. Van Vleck et al., 1987): se construye una matriz con todos los animales simulados hasta el momento ordenados cronológicamente (se trata una matriz simétrica) y se asignan sus elementos empezando por el primer elemento de la primera fila, y continuando con esa misma fila, siguiendo como reglas: a) elementos de la diagonal = 1 + 1/2 de la suma de los coeficientes de relación entre sus padres si los conocemos; en caso contrario 1; b) elementos de fuera de la diagonal = 1/2 de la suma del valor de sus padres en esa fila. Como resultado se obtienen los coeficientes de relación aditiva de parentesco entre todos los animales implicados, y a partir de esos valores se deducen los coeficientes de consanguinidad y de parentesco de Malécot. El coeficiente de consanguinidad de un animal será el coeficiente de relación de parentesco consigo mismo (elementos de la diagonal de la matriz) menos 1. El coeficiente de parentesco de Malécot entre dos animales será el coeficiente de relación aditiva de parentesco (elementos de fuera de la diagonal) dividido por 2. Es interesante señalar que este coeficiente de parentesco entre dos individuos es igual al coeficiente de consanguinidad de un hipotético descendiente, lo que indica qué apareamientos es deseable evitar para mantener la variabilidad.
CÁLCULO DEL TAMAÑO EFECTIVO DE POBLACIÓN
El tamaño efectivo de población (Ne) se calcula para cada generación teniendo en cuenta el número de machos (Nm) y hembras (Nh) que han actuado como reproductores según la expresión:

Es obligado indicar inmediatamente que esa expresión asume apareamientos al azar y homogeneidad en el número de hijos producidos por cada reproductor, por lo que sólo se puede considerar como una estimación del límite superior que podría tener el tamaño efectivo de población. Bajo selección, se puede considerar que el tamaño efectivo de población será de aproximadamente un 70 % del calculado mediante las fórmulas anteriores (FAO, 2013).
Se ha considerado interesante contemplar el concepto de tamaño efectivo de población dentro del programa por ser muy utilizado en el terreno práctico de la conservación de poblaciones, y considerarlo útil a efectos de introducir la discusión sobre la relación existente entre la selección animal y la conservación de poblaciones.
Para interpretar los resultados que ofrece el programa hay que tener en cuenta que si en una generación se produce la baja de algún animal que ha actuado como reproductor, el tamaño efectivo calculado no se podrá deducir a partir de la estructura final de la población en esa generación. Tampoco se podrá deducir a partir del aumento de la consanguinidad de la población, dado que este se calcula como el valor medio de los coeficientes de consanguinidad de los animales presentes en la población, y no a partir del tamaño efectivo calculado.
INTERFAZ GRÁFICA
El programa, desarrollado en entorno gráfico mediante Liberty Basic (version 4.04, Shoptalk Systems), utiliza ficheros de formato Comma Separated Value (CSV) para alimentar ciertos datos por parte del usuario. Según la configuración de la computadora es posible que los ficheros CSV no se abran directamente con la hoja de cálculo predeterminada sino a través de Internet Explorer de Windows; si eso resulta inconveniente o incómodo para trabajar, en la página de soporte técnico y ayuda de Microsoft se puede acceder al paquete de configuración de Internet Explorer para abrir documentos de Office. Por otra parte, también hay que tener en cuenta al importar los ficheros a la hoja de cálculo que esos ficheros CSV utilizan el punto (.) como símbolo decimal.
Resultados y discusión
VISIÓN GENERAL: COMPONENTES
El programa se estructura en base a ocho menús desplegables:
1) Inicio: permite conocer en que generación de la simulación nos encontramos [Situación actual] e iniciar el proceso de simulación creando un nuevo rebaño de partida [Crear población].
2) Aparear: permite definir y realizar los apareamientos entre los reproductores activos.
3) Simular: permite simular los descendientes de los apareamientos previamente diseñados en hasta un máximo de diez generaciones.
4) Resultados VG: permite consultar las producciones, información genealógica, coeficiente de consanguinidad y valor genético predicho (EBV o VG) y su precisión, para los animales que forman parte de la población en cada una de las generaciones simuladas -con la información adicional de si ha sido dado de baja o continúa en activo-.
5) Consultas fxy: permite consultar cuales son las hembras emparentadas con cada uno de los machos activos de la población, cuantificando su grado de relación familiar mediante el coeficiente de parentesco (fxy).
6) Evolución: ofrece los resultados de las características de la población en cada una de las generaciones simuladas.
7) Utilidades: facilita el acceso a algunas aplicaciones de MS-Windows (calculadora, notas, explorador).
8) Sobre MiPob: da acceso al manual de usuario, información del programa y fundamentalmente a los valores de los parámetros de simulación que por defecto considera el programa (y que pueden ser modificados -con precaución- si lo desea el usuario).
CREACIÓN DEL REBAÑO
Para empezar se debe crear un rebaño [Inicio [Crear Población]]. Al hacerlo se borra toda la información que exista si se hubiera creado un rebaño anterior.
Los datos de cada generación que se vayan creando se pueden consultar en [Resultados_VG] Los datos se abren con la hoja de cálculo predeterminada en el ordenador para poder consultarlos, ordenarlos, etc., pudiendo guardar los cambios para recordar los criterios de elección de reproductores que se utilicen en el proceso de selección. La información que se tendrá de cada animal es su Identificación, Grupo (Toro, Vaca, Ternero, Ternera), Peso (kg), EBV (valor genético predicho), Precisión (de su EBV), Estado (si ha sido dado de Baja), Padre, Madre y Coef. Consanguinidad (coeficiente de consanguinidad) (figura 1). Dado que se simula el peso al año, los animales jóvenes que aparecen en cada generación y se han dado de baja no disponen de registro de peso.
ELECCIÓN DE LOS FUTUROS REPRODUCTORES Y TOMA DE DECISIONES DE APAREAMIENTO
De los animales presentes en el rebaño en cada generación, se deben escoger los machos (toros o terneros) y hembras (vacas o terneras) que se quieran utilizar como futuros reproductores. Una vez escogidos, se debe introducir la información a través del menú [Aparear].
Primero, se recuerdan las reglas del manejo reproductivo del rebaño, que son usar un mínimo de 2 machos (máximo 5) y un máximo de 25 hembras por macho (con un total de 50 hembras reproductoras por generación). Luego, se debe proceder a definir los apareamientos en la hoja de cálculo que a tal efecto se abrirá (figura 2). En ella se debe trabajar de la siguiente forma: (a) Identificar a los machos a aparear en la primera fila de cada columna; (b) Indicar debajo el número de hembras que va a cubrir cada macho; (c) Listar debajo las hembras que cubrirá cada macho.
Tras guardar los apareamientos diseñados (siempre manteniendo el formato CSV) se puede pasar a simular la siguiente generación. Por si hay algún error en el diseño de los apareamientos en ese momento se controla: 1) que se haya usado un mínimo de 2 machos, 2) que se haya apareado un total de 50 hembras, 3) que no se hayan apareado más de 25 hembras por macho, 4) que los animales realmente existan y no estén dados de baja, 5) que no se haya confundido algún macho con alguna hembra y viceversa, 6) que se intente aparear dos veces una misma hembra, 7) que se intente aparear dos lotes de hembras con un mismo macho. El programa emite el correspondiente aviso si ocurre alguno de los errores anteriores para poder volver al fichero de apareamientos y hacer la oportuna corrección.
SIMULACIÓN DE LAS GENERACIONES
Usando los apareamientos indicados en [Aparear] se creará la próxima generación de animales mediante la opción [Simular]. Para ello se debe de haber salvado y cerrado la hoja de cálculo de apareamientos.
Una vez creada la nueva generación de animales se deberá trabajar del mismo modo que con la población inicial, es decir, escogiendo los futuros reproductores y decidiendo cómo aparearlos. La única diferencia es que en esa y sucesivas generaciones se podrá tener en cuenta: 1) la consanguinidad de los animales al escogerlos como futuros reproductores; 2) el parentesco entre animales al decidir cómo aparearlos.
El coeficiente de consanguinidad aparece en la última columna de los ficheros de consulta accesibles en [Resultados_VG] (figura 1). El parentesco se puede consultar (a partir de la primera generación) en [Consulta_fxy] con funcionamiento análogo a la consulta de datos productivos y predicción del valor genético, abriendo una hoja de cálculo. En este caso la información que se obtiene es, para cada uno de los machos en activo, la relación de hembras en activo con él emparentadas, y su coeficiente de parentesco (no aparecen listadas las hembras no emparentadas) (figura 3). Los valores que aparecen corresponden al coeficiente de parentesco de Malécot.
El programa está pensado para poder trabajar con él en varias sesiones de modo que si se cierra se guarde la información generada hasta el momento, y que sólo se borre esa información cuando decidamos crear otro rebaño. Por eso, puede ser útil consultar cual fue la última generación que simulamos para retomar el trabajo; se puede hacer en cualquier momento en [Inicio[Situación actual]], apareciendo un mensaje que informa de la última generación simulada. También se puede deducir la última generación simulada si se consulta la evolución de las características de la población en [Evolución]. Cabe indicar que se puede repetir una determinada generación volviendo a simularla, en cuyo caso todas las generaciones posteriores que ya se hubieran simulado también se borrarían.
EVALUACIÓN DE LOS CAMBIOS PRODUCIDOS EN LA POBLACIÓN
Mediante la consulta [Evolución] se puede consultar el Resumen de las generaciones simuladas de la población, y por lo tanto evaluar las consecuencias de las decisiones de apareamiento que se hayan ido tomado hasta el momento (figura 4).
Concretamente se puede ver cómo evolucionan, en función de cómo se hayan realizado los apareamientos: 1) el tamaño efectivo de la población; 2) el número de animales según grupo y estado; 3) la media del peso al sacrificio; 4) la media del valor genético verdadero; 5) la consanguinidad media de la población; 6) la fertilidad media del rebaño; y 7) la frecuencia de los alelos singulares de la población.
PARÁMETRO DE SIMULACIÓN
Los valores utilizados para los distintos parámetros de la simulación se pueden consultar en [Sobre_MiPob [Valores simulación]] (figura 5). Esos valores se pueden modificar abriendo el archivo de texto param.txt aunque sólo tiene sentido hacerlo antes de crear una nueva población, y nunca en medio de alguna generación (los resultados serían totalmente incoherentes). Si se modifican hay que salir y volver a entrar en el programa para hacer efectivos los cambios.
Conclusiones
El programa MiPob permite entender el interés, las limitaciones y las consecuencias de seleccionar una población animal de censo reducido como estrategia de mejora genética. Se basa en la simulación de un proceso de selección a lo largo de varias generaciones partiendo de la idea de que es más fácil aprender haciendo, que viendo, o escuchando. El estudiante se ve obligado a tomar sus propias decisiones en su propio rebaño, motivándose para querer saber que puede ocurrir ante distintas decisiones. Por ello, es una herramienta académica útil para introducir conceptos como la deriva genética, la consanguinidad, el parentesco entre animales, la depresión consanguínea de las producciones, el valor genético de los animales, la precisión en la predicción de esos valores genéticos, los apareamientos dirigidos y la respuesta genética a la selección. No obstante, hay que tener en cuenta que es solo una herramienta de aprendizaje, y como tal debería utilizarse para alimentar el debate y la discusión entre alumnos y profesores, nunca sustituirlo ni mermarlo. Utilizarlo sin más, podría conducir a confundir ciertos conceptos, no sólo de los procesos de selección, sino también de su papel en los programas de mejora y en la producción animal en general.
El programa ha sido utilizado durante dos cursos consecutivos en la docencia de una asignatura introductoria a la Mejora genética animal en la Escuela Técnica Superior de Ingenieros Agrónomos de la Universidad Pública de Navarra (España). La acogida por parte de los estudiantes ha sido positiva, y sus comentarios han permitido corregir errores y mejorar su funcionamiento. Se puede descargar, gratuitamente y sin ningún tipo de limitación de uso, en la siguiente dirección: http://www.unavarra.es/rmga/mipob. No requiere instalación ni importantes recursos informáticos, de modo que se puede utilizar guardándolo en cualquier carpeta de cualquier soporte físico de memoria. Incluye un Manual de uso en el que someramente se explica tanto el funcionamiento como los fundamentos teóricos del programa. Aunque está expresamente desarrollado para estudiantes hispanohablantes por la ausencia de material específico en su lengua materna, también hay disponible una versión del programa en inglés. Pese a que el programa se distribuye tal cual, se prevé su actualización y corrección de posibles errores como respuesta al envío de sugerencias y comentarios al autor de esta contribución.
Bibliografía
1. Altarriba, J.; Moreno, C. and Varona, L. 2010. Population and quantitative genetics (PQgen): A software program for teaching on animal breeding. 9th World Congress on Genetics Applied to Livestock Production. Leipzig, Germany. pp. 1-97. [ Links ]
2. Buchanan, D.S.; Burdit, L.G. and Willham, R.L. 1988. COWGAME: A beef cattle selection simulation program. J Hered, 79: 215. [ Links ]
3. Carlson, J.P. 1985. Overcoming animal breeding teaching problems. NACTA Journal 29: 71-73. [ Links ]
4. DeRouen, S.M.; Moss, J.W. and Franke, D.E. 1989. Using a computer simulation program for animal breeding instruction. NACTA Journal33: 72-74. [ Links ]
5. FAO. 2013. In vivo conservation of animal genetic resources. FAO Animal Production and Health Guidelines. No. 14. Rome. 242 pp. [ Links ]
6. Heidhues, T. and Henderson, C.R. 1961. Teaching selection principles with herd records generated by an electronic computer. J Anim Sei, 20: 659-664. [ Links ]
7. Kinghorn, B.P. 1992. GENUP - a suite of programs to help teach animal breeding theory. Proc. 10th Australian Association of Animal Breeding and Genetics 10: 555-559. [ Links ]
8. Medrano, J.F., Ahmadi, A. and Casellas, J. 2010. Dairy Cattle Breeding Simulation Program: A simulation program to teach animal breeding principles and practices. J Dairy Sci, 93: 2816-2826. [ Links ]
9. Sorensen, D.A. and Kennedy, B.W. 1984. Estimation of response to selection using least-squares and mixed model methodology. J Anim Sci, 58: 1097-1106. [ Links ]
10. Van Vleck, L.D., Pollak, E.J. and Oltenacu, E.A.B. 1987. Genetics for the animal sciences. W.H. Freeman and Company. New York. 391 pp. [ Links ]
11. Willham, R. L. 1968. New goals in undergraduate teaching of genetics. J Anim Sci, 27: 888-892. [ Links ]
Recibido: 9-8-14.
Aceptado: 24-11-14.