My SciELO
Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Cited by Google
Cited by Google -
 Similars in
SciELO
Similars in
SciELO -
 Similars in Google
Similars in Google
Share
Actas Urológicas Españolas
Print version ISSN 0210-4806
Actas Urol Esp vol.35 n.7 Jul./Aug. 2011
Cáncer de próstata: la revolución de los genes de fusión
Prostate Cancer: The Revolution of the Fusion Genes
A. Fernández-Serraa, J. Rubio-Brionesb, Z. García-Casadoa, E. Solsonab y J.A. López-Guerreroa
aLaboratorio de Biología Molecular, Fundación Instituto Valenciano de Oncología, Valencia, España
bServicio de Urología, Fundación Instituto Valenciano de Oncología, Valencia, España
Este trabajo ha sido financiado por las ayudas: FIS PI061619 y PI10/01206, Madrid, Astra Zeneca, España y ACOMP/2009/176 de la Generalitat Valenciana.
Dirección para correspondencia
RESUMEN
Contexto: Los reordenamientos TMPRSS2-ETS constituyen una alteración específica y frecuente en tumores prostáticos que conlleva la sobreexpresión de los genes ETS que codifican para la familia E26 de factores de transcripción, promoviendo la proliferación celular. De entre estos ERG sobreexpresa en casi el 50% de los carcinomas prostáticos.
Síntesis de evidencia: TMPRSS2-ERG sobreexpresa a ERG en respuesta a andrógenos. Estructuralmente este reordenamiento se debe a una deleción intersticial y, en menor medida, a una translocación recíproca, y tiene un papel clave en el metabolismo celular. Casi todos los transcritos del gen de fusión producen una proteína ERG truncada, y la presencia de una determinada isoforma de este gen indica la clonalidad del tumor, de modo que la metástasis comparte isoforma de TMPRSS2-ERG con su localización primaria. Aunque las implicaciones pronósticas de TMPRSS2-ERG no están totalmente elucidadas se considera un campo de gran potencial diagnóstico, por lo que el desarrollo de técnicas que permitan determinar la presencia y características de este gen de forma no invasiva es muy interesante. La presencia del gen de fusión constituye dos grupos moleculares dentro del CaP con un comportamiento evolutivo claramente diferencial, lo que hace que farmacológicamente el gen de fusión constituya una diana terapéutica potencial. En este sentido, el uso de fármacos anti-HDAC (tricostatina), antagonistas del receptor de estrógenos alfa y acetato de abiraterona han mostrado resultados prometedores.
Conclusiones: Esta revisión expone el gran potencial que representa la investigación de los genes de fusión en el CaP y la necesidad de profundizar en su estudio.
Palabras clave: TMPRSS2-ERG. Cáncer de próstata. Pronóstico. Diagnóstico. Genes de fusión.
ABSTRACT
Background: TMPRSS2-ETS fusion gene rearrangements constitute a very common and specific alteration in prostate cancer cells. These genetic alterations lead the overexpression of ETS genes which encode the E26 family of transcription factors involved in cell proliferation. Of this family, the ERG oncogene is overexpressed in almost 50% of prostate cancer cases.
Evidence synthesis: TMPRSS2-ERG overexpresses ERG through an androgen-mediated response. Structurally, the rearrangement is due to interstitial deletion and to a lesser extent to reciprocal translocation and plays a key role in cellular metabolism. Almost all fusion gene transcripts produce a truncated ERG protein and the presence of a specific isoform of this gene suggests the clonality of the tumor; hence, metastasis shares the fusion gene status of their primary lesion. Although the prognostic implications of TMPRSS2-ERG have not been fully elucidated, they constitutes a field of great diagnostic potential and, therefore, the development of techniques to identify and to analyze the presence and characteristics of this gene in a non-invasive fashion deserves great interest in this area. Currently, there is evidence supporting the hypothesis that the presence of fusion gene differentiates two molecular groups within prostate cancer with a differential behaviour making the fusion gene a potential therapeutic target. In this regard, the use of anti-HDAC (trichostatin), antagonists of estrogen receptor alpha and abiraterone acetate have shown promising results.
Conclusions: This review describes the great potential offered by the investigation of fusion genes in PC and the need for further studies.
Key words: TMPRSS2-ERG. Prostate cancer. Prognostic. Diagnostic. Fusion genes.
Contexto
El cáncer de próstata (CaP) es el tercer tipo de tumor más frecuente en hombres. La aparición de esta neoplasia está ligada a la edad. En la Unión Europea el CaP es causante directo de la muerte del 3% de los varones y del 10% de las muertes oncológicas. La incidencia del CaP ha aumentado en los últimos años, en primer lugar debido al incremento significativo de la esperanza de vida, y en segundo lugar por la introducción de la determinación de los niveles séricos de PSA en el cribado del CaP, aumentando el diagnóstico en fase preclínica. En España la situación epidemiológica del CaP no difiere significativamente de lo que se observa en el resto de Europa. Anualmente se diagnostican unos 13.300 nuevos casos (el 13,6% de los tumores entre varones españoles), siendo la supervivencia a los 5 años en torno al 65%, con una edad media de fallecimiento de 75 años1.
Histológicamente el CaP está constituido por una mezcla heterogénea de células, principalmente epiteliales y estromales2. Este proceso comienza con una displasia que se inicia como atrofia inflamatoria proliferativa (PIA), progresa a neoplasia intraepitelial prostática (PIN) y en algunos casos desemboca en un carcinoma. Existen evidencias que apuntan a que uno de los desencadenantes de la tumorogénesis podría ser una inflamación prostática debida a agentes infecciosos o ingesta de carcinógenos. Paralelamente, algunas células acumulan alteraciones génicas que, junto con la señalización andrógenica, estimulan el crecimiento y proliferación del tumor3.
Clínicamente hay dos grandes grupos de CaP: aquellos tumores prostáticos capaces de diseminarse que acabarán siendo letales y otros que son relativamente indolentes3, lo cual plantea de entrada el problema de cómo distinguir unos tumores de otros y el modo de abordaje clínico óptimo en cada caso. Actualmente los niveles séricos de PSA aportan información altamente órgano-específica, pero poco específica de enfermedad. Así, tanto en hiperplasia prostática benigna como en prostatitis se producen aumentos séricos de este biomarcador, pero además muchos pacientes con CaP localizado presentan valores de PSA que se solapan con los de sujetos sanos, resultando una zona gris de difícil interpretación el intervalo entre 4 y 10 ng/ ml4. Por otra parte, numerosos estudios sugieren que el CaP está sobrediagnosticado en un 30-50% de los casos, es decir, no todos los pacientes con PSA elevado tienen un tumor prostático. Después del diagnóstico el principal factor pronóstico es el grado de Gleason, que consiste en asignar un grado del 1 al 5 de mayor a menor diferenciación a cada uno de los dos focos principales del tumor. La suma de ambos valores constituye el score. Aunque este parámetro es el gold standard en el manejo clínico del CaP, presenta ciertos problemas: por una parte, la determinación se realiza sobre tejido obtenido de una biopsia prostática, procedimiento quirúrgico que presenta una cierta comorbilidad, especialmente significativa en pacientes añosos; por otro lado, este score sufre de variación interpretativa5.
En el pronóstico de la enfermedad la carencia de un método fiable que permita determinar el momento en el que el tumor prostático se convertirá en hormonorresistente es problemática, pues a partir de aquí el pronóstico del paciente empeora y frecuentemente se producen metástasis óseas, para las que actualmente sólo se dispone de tratamiento paliativo6.
Por todo esto es muy importante la identificación de nuevos biomarcadores que representen herramientas útiles en el diagnóstico y en el manejo clínico del CaP. Estos marcadores deben ser determinables mediante técnicas objetivas, cuantitativas y mecanismo-específicas, y en la medida de lo posible deben ser accesibles por métodos no invasivos.
La presente revisión pretende ofrecer una visión global del estado de algunos de estos biomarcadores, haciendo especial hincapié en los genes de fusión TMPRSS2-ETS y su posible implicación en el manejo clínico del paciente con CaP.
Nuevos biomarcadores en cáncer de próstata
Un abordaje en la búsqueda de biomarcadores es estudiar la expresión de genes relacionados con el CaP. Así, se ha descrito la infraexpresión del gen GSTP1 (Glutation-S-transferasa PI) (11q13) por hipermetilación de su región promotora. Este gen cataliza la detoxificación molecular mediada por glutatión7. Esta alteración se encuentra en la gran mayoría de neoplasias prostáticas y en un 70% de las neoplasias prostáticas intraepiteliales (PIN). La mayoría de tumores con esta alteración se asocian también a la hipermetilación de otros genes como p16INK4A (9p21), p14ARF (9p21) y MGMT (10q26)8.
También se ha comprobado la sobreexpresión de AMACR (5p13.2-q11.1) en un 88% de los CaP. Este gen codifica para alfa-metil coenzima A racemasa, una enzima implicada en la beta-oxidación de ácidos grasos largos de cadena ramificada7 y a la que se ha propuesto como factor clave en la relación del CaP con ciertos hábitos alimenticios. Dada su alta expresión en CaP algunos autores han desarrollado test diagnósticos en orina con resultados prometedores9, si bien las series analizadas no son lo suficientemente consistentes como para ofrecer unos datos definitivos.
La expresión de otros genes se ha asociado a la progresión del CaP. Así, el gen PAR-2 (5q13.3), que codifica para un receptor acoplado a proteínas G activadas por serín-proteasas específicas, parece implicado en la metástasis10 y sobreexpresa en aproximadamente un 40% de los CaP11. Otros genes presentan una asociación con parámetros clínicos, como HEPSIN (TMPRSS1) (19q11-q13.2), que codifica para una proteína transmembrana con actividad serín-proteasa, y Pim-1 (6p21.2) que codifica para una proteína con actividad serina-treonina quinasa12. SPINK-1 (5q32) codifica para una proteína inhibidora de la secreción pancreática de tripsina y está sobreexpresado en aproximadamente un 10% de los casos de CaP. Su expresión es detectable en muestras de orina y parece relacionado con el intervalo libre de progresión bioquímica13.
PTEN (10q23.3) es un gen supresor de tumores que codifica para una proteína que desfosforila el fosfatidilinositol-3-fosfato (PIP3). La pérdida de función de este gen es una de las anomalías génicas más usuales en diversos tipos de cáncer. Concretamente en CaP se produce en aproximadamente un 40% de los casos. Las deleciones de PTEN se han asociado con grados de Gleason > 7, así como con la recaída bioquímica y metástasis ganglionar. La deleción de este gen produce una activación constitutiva de la vía PI3K que es clave en muchos procesos oncológicos14.
PCA-3 (DD3) es un gen que codifica para un ARN mensajero (ARNm) no codificante con alta tasa de expresión en el tejido tumoral prostático, y cuya función biológica está todavía por esclarecer. Cada vez hay más evidencia de que su determinación en orina tras masaje prostático es mucho más específica que los niveles de PSA para la detección de CaP. Está en discusión su capacidad pronóstica, habiendo ofrecido resultados controvertidos al respecto15. A día de hoy es el único de estos biomarcadores moleculares de uso clínico en algunos centros de referencia en Estados Unidos y en Europa (www.pca3.org)16.
El descubrimento de TMPRSS2-ETS en el cáncer de próstata
Los genes de fusión se forman cuando dos cromosomas, o dos regiones del mismo, se rompen y cambian de posición dando lugar, en ocasiones, a un nuevo gen (llamado también gen quimérico) con una nueva función. Dichos genes son resultado de aberraciones cromosómicas estructurales (translocaciones recíprocas, deleciones o inversiones), que han sido descritas en muchos tipos de cáncer17. No obstante, teniendo en cuenta el tamaño del genoma humano y el bajo porcentaje de ADN codificante, la mayoría de roturas cromosómicas que, finalmente resultan en una translocación balanceada, no producirían un efecto fenotípico porque no afectan a regiones codificantes.
Los genes de fusión han sido extensamente caracterizados en linfomas, leucemias y sarcomas, pero sólo recientemente han empezado a identificarse y caracterizarse en carcinomas, principalmente en CaP aunque también se están identificando en carcinoma papilar y folicular de tiroides, en algunos carcinomas de riñón y en carcinomas mucoepidermoides18. La tardanza en el descubrimiento de estos genes en carcinomas probablemente se debe a problemas en el uso de técnicas citogenéticas clásicas en este tipo de tumores13.
En el campo de las fusiones fenotípicamente significativas implicadas en el cáncer tenemos dos grandes grupos, en las que se produce la sobreexpresión de un oncogén, como por ejemplo COL1A1-PDGF-Beta, presente en dermatofibrosarcoma protuberans, y el segundo grupo es en el que se da una proteína quimérica que implica a un factor de transcripción, por ejemplo EWS-FLI1 en sarcomas de Ewing19. Si bien la presencia de genes quiméricos no se puede observar directamente usando técnicas de análisis de alto rendimiento (arrays de ADNc), en conjuntos de datos obtenidos en estos experimentos de expresión pueden ponerse de manifiesto reordenamientos génicos y alteraciones del número de copias, siempre que se usen tratamientos analíticos no tradicionales para el procesamiento de los datos. El test de la "t" de Student, por ejemplo, detecta genes que sobreexpresan constantemente en un determinado tipo de cáncer, es decir, tienen un perfil de biomarcadores, pero no es efectivo a la hora de poner de manifiesto genes que sobreexpresan en un subconjunto de tumores dentro de un determinado tipo de cáncer (outliers)13.
En 2005 la revista Science publicó un artículo del grupo de Arul M. Chinnaiyan, de la Universidad de Michigan, en el que se pone a punto un método bioinformático denominado COPA (Cancer Outlier Profile Analysis). Dicho test busca encontrar y poner de manifiesto genes que sobreexpresan en un subconjunto de los casos de un determinado cáncer (en este caso en CaP). Para ello, analizaron datos de 10.486 experimentos de microarrays contenidos en la Oncomine Database (https://www.oncomine.org/)13. En el desarrollo de este método subyace la idea de que con la evaluación de la varianza, usando la mediana en lugar de la media, se mantendrán los picos de expresión de los genes que sólo sobreexpresan en un subconjunto de los datos. En este análisis se incluyeron genes con una sobreexpresión conocida para un determinado tipo de cáncer a modo de confirmación, por ejemplo RUNXT1 en leucemias. Los genes ERG (21q22.3) y ETV1 (7p21.2) fueron los que presentaron un mayor perfil de outliers para el CaP. Ambos genes son factores de transcripción de la familia ETS (E26) implicados en procesos de proliferación celular.
El patrón de expresión de estos factores de transcripción de la familia ETS es mutuamente excluyente en un subconjunto de casos de CaP, comportamiento común a otros tumores como el sarcoma de Ewing, en el que la sobreexpresión de estos genes está relacionada con fusiones génicas EWS-ETS20. Partiendo de esta analogía, el grupo de Chinnaiyan formuló la hipótesis de que la sobreexpresión de ERG y ETV1 podía deberse a una fusión génica. A fin de comprobar la hipótesis emplearon la técnica de amplificación rápida de extremos de ADNc (RLM-RACE) determinando que el gen TMPRSS2 estaba fusionado en la posición 5' de estos miembros de la familia ETS13.
Síntesis de evidencia
Implicaciones biológicas
TMPRSS2 está situado en el locus 21q22.3 y codifica para un receptor transmembrana de la familia STP (serín proteasa transmembrana de tipo II) con estructura multimérica. Esta proteína está formada por un dominio proteasa de la familia S1, un dominio LDLRA que forma un sitio de unión para el calcio, y un tercer dominio transmembrana. TMPRSS2 está regulada por andrógenos, ya que tiene un elemento de respuesta a andrógenos en la región promotora de su gen, se encuentra en el proteoma del fluido seminal y está muy expresada en tejido prostático y, en menor medida, en tejido epitelial del colon, estómago, epidídimo y mama. Funcionalmente, al activarse esta proteína el dominio serín-proteasa se libera de la superficie celular al espacio extracelular y activa a PAR-2 (receptor activado proteasa), que como se ha mencionado anteriormente es un receptor G que desempeña un papel importante en la metástasis del CaP10. No obstante, su significación no está muy clara, ya que en modelos murinos knock-out para TMPRSS2 mostraron un fenotipo normal, lo que parece indicar que se trata de un gen redundante21.
Por su parte, ERG (21q22.2) codifica para una proteína nuclear de 363 residuos que se une específicamente al ADN en regiones ricas en purina y actúa como factor de transcripción22. Está formado por 17 exones que abarcan 300 kilobases y genera, al menos, 9 isoformas por splicing alternativo, de las cuales 7 codifican para proteínas. Este gen se expresa en tejidos endoteliales, células hematopoyéticas, riñón y tracto genitourinario. Concretamente en CaP ERG es el gen más persistentemente sobrexpresado23. De entre los distintos miembros de la familia ETS (que incluye también a ETV4, ETV5, etc.), muchos están envueltos en procesos de invasividad y metástasis24. Esta familia génica está caracterizada por la presencia en todos sus miembros de dominios ETS de unión al ADN y varios dominios de unión a proteínas. Concretamente ERG interactúa con metiltransferasas histona H3 específica (ESET), pudiendo participar en el silenciamiento epigenético de otros genes25. Una hipótesis de la actuación de este gen en CaP sería que responde a señales mitogénicas y de estrés transducidas por la vía de las MAPK, actuando sobre genes diana causantes del proceso de tumorogénesis23 (Figura 1).

Figura 1. Actuación de los factores de transcripción de la
familia ETS sobre reacciones de estrés mediadas por la vía
de transducción de señales de las MAPK. Adaptado de
Petrovics et al23. El fondo rojo indica sobreexpresión del gen
y el azul infraexpresión. Resaltado en rojo ERG.
TMPRSS2 y ERG tienen la misma orientación transcripcional, por tanto la fusión de ambos genes se debe principalmente a una deleción intersticial en la que se pierde una región de aproximadamente 3 megabases en el locus 21q22 y, en menor medida, a una translocación recíproca13. En los casos delecionados la región que se pierde en el brazo largo del cromosoma 21 contiene 15 genes, de los cuales al menos dos (ETS2 y HMGN1) han sido relacionados con la progresión del CaP26.
Desde el descubrimiento inicial de dos fusiones implicando a los genes TMPRSS2 y ERG y otras dos isoformas de TMPRSS2-ETV1 por el grupo de Chinnaiyan en 200513, se han descrito muchas otras formas. Actualmente hay publicadas 20 fusiones implicando a ERG, otras dos implicando a ETV4 y otra a ETV5, todas con TMPRSS2 en posición 5'. También se han caracterizado fusiones con otros genes en 5' como SLC45A313 (Figura 2). Estas últimas translocaciones, aunque interesantes desde el punto de vista biológico, tienen un potencial clínico bastante limitado debido a sus bajas incidencias (1-4%) (Tabla 1).
A nivel proteico casi todos los transcritos del gen de fusión producen principalmente una proteína ERG truncada más que una proteína quimérica13, ya que la región traslocada de TMPRSS2 corresponde a la región promotora no codificante del gen.
Tenemos evidencias de que cuando en el tumor primario se produce ARNm de TMPRSS2-ERG, la expresión se pierde cuando se bloquea la señalización androgénica del paciente, recuperándose en el estadio hormonorresistente del tumor. Al fusionarse TMPRSS2 y ERG se produce una activación hormonodependiente del factor de transcripción. Por tanto, esta desregulación también afecta a todos los genes diana de ERG, de los cuales el más frecuentemente cosobreexpresado es HDAC-1, que cataliza la desacetilación de histonas promoviendo la expresión génica18.
Globalmente la sobreexpresión de ERG puede conducir a alteraciones en la ruta Wnt (responsable del dispositivo de proliferación celular), reprogramación epigenética y desregulación de vías de muerte celular. Es de resaltar el papel dentro de estas vías de la E-cadherina (CDH1) proteína a la que se considera una importante supresora de la metástasis. En CaP el gen se encuentra infraexpresado por hipermetilación de su región promotora27. También es muy significativa la implicación del receptor de andrógenos (RA), que actúa como regulador maestro de la progresión de la transición de las fases G1-S del ciclo celular induciendo señales que promueven G128 (Figura 3).
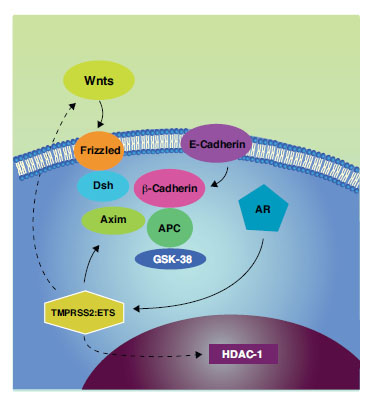
Figura 3.Mecanismo molecular de interacción de
TMPRSS2-ERG con las vías de señalización de hormonas
androgénicas características del CaP, y otras más generales
en oncología como Wnt y hedgehog.
También hay una marcada cooperatividad entre la presencia del gen de fusión y la vía PI3K, que desempeña un papel fundamental en el metabolismo del cáncer. Por una lado se ha demostrado la correlación existente entre la presencia de TMPRSS2-ERG, deleciones en PTEN y expresión de MYC y, en segundo lugar, modelos murinos transgénicos para el gen de fusión desarrollan PIN, pero sólo en el contexto de una activación generalizada de la vía PI3K29.
Importancia clínica
El descubrimiento de esta familia de genes de fusión tiene varias facetas especialmente interesantes: por un lado puede contribuir a elucidar funcionalmente el comportamiento hormonodependiente del CaP, y por otro puede constituir una diana terapéutica13; además, arroja interesantes posibilidades diagnósticas y pronósticas.
Si bien existen numerosas fusiones que involucran a TMPRSS2 y otros miembros de la familia ETS, en adelante se revisará exclusivamente TMPRSS2-ERG, dado que se trata del gen quimérico más incidente y el más ampliamente estudiado13.
TMPRSS2-ERG presenta una frecuencia del 40-70% en CaP, lo que da idea de su potencial como biomarcador13,30. Aunque existen muchos reordenamientos implicando a ambos genes, la mayor parte de ellos implican al exón 1 de TMPRSS2 y al 4 de ERG (T1E4) seguida de la fusión de los exones 1 de TMPRSS2 con el 2 de ERG (T1E2), que globalmente representan aproximadamente el 80% de estos reordenamientos30. La expresión de la proteína codificada por la isoforma T1E4 podría favorecer el crecimiento tumoral.
La carcinogénesis prostática es, generalmente, un proceso multicéntrico en el cual coexisten varias vías patogénicas con o sin el concurso de la vía ETS18. El CaP multifocal es un conjunto heterogéneo de enfermedades originadas en expansiones clonales múltiples e independientes. Una forma de determinar la clonalidad del CaP es determinar la presencia e isoforma de TMPRSS-ERG en los distintos focos del tumor y en la metástasis. Se han encontrado focos en una misma próstata portando distintas isoformas del gen de fusión o sin tenerlo, e incluso, dos isoformas en el mismo foco. Ahora bien, la metástasis siempre comparte el estatus del gen de fusión con su tumor primario, indicando que es un único clon el que evoluciona de la localización original para hacer metástasis18.
A nivel diagnóstico el potencial de la detección de TMPRSS2-ERG es innegable, dada su especificidad. Si bien está perfectamente establecida la presencia del gen de fusión en piezas quirúrgicas congeladas18 y en piezas fijadas en formol e incluidas en parafina (FFPE)30, tiene un evidente interés la determinación en otras muestras menos invasivas, como sangre31 y orina13.
Aún no existe consenso en las implicaciones pronósticas de TMPRSS2-ERG18 (Tabla 2). Esto puede deberse a la heterogeneidad de las series estudiadas y a las distintas técnicas usadas en la determinación: hibridación fluorescente in situ y RT-PCR principalmente.
Hay numerosas evidencias que parecen apuntar al hecho de que los CaP con el gen TMPRSS2-ERG, y los que no lo contienen, constituyen dos grupos diferenciados dentro de la enfermedad. Por una parte, un estudio de expresión génica reveló que los tumores con el gen de fusión tienen distinto perfil transcriptómico. Concretamente, 87 genes relacionados con la vía de señalización de las hormonas estrogénicas están sobreexpresados en tumores TMPRSS2-ERG positivos con respecto a los casos que no portan este gen32.
Nuestro grupo estudió los factores clinicopatológicos por análisis multivariante, estratificando la población según la presencia o ausencia del gen TMPRSS2-ERG. Los casos con el gen de fusión tuvieron, como factores pronósticos independientes, los niveles séricos de PSA en el momento del diagnóstico, el score de Gleason en la pieza de prostatectomía y los márgenes quirúrgicos, mientras que los casos no reordenados presentaron valor pronóstico: el cT, el score de Gleason y los márgenes30, lo que abre la posibilidad de considerar tratamientos más optimizados en función del estatus del gen de fusión.
El tercer argumento de la estratificación molecular del CaP es la distribución geográfica de las anomalías génicas. La población oriental tiene una menor incidencia de CaP que los occidentales. Mao et al han publicado recientemente que esta diferencia en incidencia lleva aparejadas diferencias génicas. Mientras que la frecuencia de reordenamiento TMPRSS2-ERG ronda el 50% de los casos en occidentales, en población china sería de un 2,5%, y las deleciones del gen PTEN con una frecuencia aproximada del 40% en población occidental sólo tendrían una incidencia del 7,6% en chinos33.
TMPRSS2:ERG constituye, también, una interesante diana terapéutica. En líneas celulares que contienen el gen reordenado el tratamiento con inhibidores de HDAC (tricostatina) reduce drásticamente el crecimiento tumoral34. Además, estos mismos casos TMPRSS2-ERG + parecen responder bien a fármacos agonistas del receptor de estrógenos (RE) α y antagonista de RE β32. Por último, y en ensayos clínicos, el acetato de abiraterona, molécula que bloquea la señalización androgénica inhibiendo el citocromomo P17, ha obtenido resultados prometedores en casos con TMPRSS2-ERG13.
Conclusiones
La elevada incidencia y la conexión con la vía de señalización androgénica de TMPRSS2-ERG lo convierten en un biomarcador con mucho potencial a nivel diagnóstico y pronóstico, si bien es necesario aún mucho trabajo13,18,23,30,35-40. Traslacionalmente constituye una clara diana terapéutica.
También resulta de elevado interés seguir indagando en la implicación del gen de fusión en vías de señalización (andrógenos, PI3K, Wnt, Hedgehog), lo que puede contribuir a elucidar la aún misteriosa biología molecular de este tipo de tumores.
Finalmente es prometedora la detección de esta anomalía de modo mínimamente invasivo. En resumen, dentro de la biología molecular oncológica, el campo de los genes de fusión resulta prometedor tanto a nivel básico como clínico y traslacional.
Conflicto de intereses
Los autores declaran no tener ningún conflicto de intereses.
Agradecimientos
A la Dra. Ana Cervera Ferri por su inestimable ayuda en el apartado gráfico.
Bibliografía
1. López-Abente G, Pollán M, Aragonés N, Gómez BP, Barrera VH, Lope V, et al. Situación del cáncer en España: incidencia State of cancer in Spain: incidence. An Sist Sanit Navar. 2004; 27:165-73. [ Links ]
2. Nelson WG, De Marzo AM, Isaacs WB. Prostate cancer. N Engl J Med. 2003; 349:366-81. [ Links ]
3. Taichman RS, Loberg RD, Mehra R, Pienta KJ. The evolving biology and treatment of prostate cancer. J Clin Invest. 2007; 117:2351-61. [ Links ]
4. Balk SP, Ko YJ, Bubley GJ. Biology of prostate-specific antigen. J Clin Oncol. 2003; 21:383-91. [ Links ]
5. Evans AJ, Henry PC, Van der Kwast TH, Tkachuk DC, Watson K, Lockwood GA, et al. Interobserver variability between expert urologic pathologists for extraprostatic extension and surgical margin status in radical prostatectomy specimens. Am J Surg Pathol. 2008; 32:1503-12. [ Links ]
6. Msaouel P, Pissimissis N, Halapas A, Koutsilieris M. Mechanisms of bone metastasis in prostate cancer: clinical implications. Best Pract Res Clin Endocrinol Metab. 2008; 22:341-55. [ Links ]
7. Makarov DV, Loeb S, Getzenberg RH, Partin AW. Biomarkers for Prostate Cancer. Annu Rev Med. 2009; 60:139-51. [ Links ]
8. Wright JL, Lange PH. Newer potential biomarkers in prostate cancer. Rev Urol. 2007; 9:207-13. [ Links ]
9. Rogers CG, Yan G, Zha S, Gonzalgo ML, Isaacs WB, Luo J, et al. Prostate cancer detection on urinalysis for alpha methylacyl coenzyme a racemase protein. J Urol. 2004; 172(4 Pt 1):1501-3. [ Links ]
10. Wilson S, Greer B, Hooper J, Zijlstra A, Walker B, Quigley J, et al. The membrane-anchored serine protease, TMPRSS2, activates PAR-2 in prostate cancer cells. Biochem J. 2005; 388(Pt 3):967-72. [ Links ]
11. Black PC, Mize GJ, Karlin P, Greenberg DL, Hawley SJ, True LD, et al. Overexpression of protease-activated receptors-1,-2, and-4 (PAR-1,-2, and-4) in prostate cancer. The Prostate. 2007; 67:743-56. [ Links ]
12. Dhanasekaran SM, Barrette TR, Ghosh D, Shah R, Varambally S, Kurachi K, et al. Delineation of prognostic biomarkers in prostate cancer. Nature. 2001; 412:822-6. [ Links ]
13. Tomlins SA, Bjartell A, Chinnaiyan AM, Jenster G, Nam RK, Rubin MA, et al. ETS gene fusions in prostate cancer: from discovery to daily clinical practice. Eur Urol. 2009; 56:275-86. [ Links ]
14. McMenamin ME, Soung P, Perera S, Kaplan I, Loda M, Sellers WR. Loss of PTEN expression in paraffin-embedded primary prostate cancer correlates with high Gleason score and advanced stage. Cancer research. 1999; 59:4291. [ Links ]
15. Nakanishi H, Groskopf J, Fritsch e HA, Bhadkamkar V, Blase A, Kumar SV, et al. PCA3 molecular urine assay correlates with prostate cancer tumor volume: implication in selecting candidates for active surveillance. J Urol. 2008; 179:1804-10. [ Links ]
16. Groskopf J, Aubin SMJ, Deras IL, Blase A, Bodrug S, Clark C, et al. APTIMA PCA3 molecular urine test: development of a method to aid in the diagnosis of prostate cancer. Clin Chemistry. 2006; 52:1089. [ Links ]
17. Frohling S, Dohner H. Chromosomal Abnormalities in Cancer. N Eng J Med. 2008; 359:722. [ Links ]
18. Kumar-Sinha C, Tomlins SA, Chinnaiyan AM. Recurrent gene fusions in prostate cancer. Nat Rev Cancer. 2008; 8:497-511. [ Links ]
19. López-Guerrero JA, Pellín A, Noguera R, Carda C, Llombart-Bosch A. Molecular analysis of the 9p21 locus and p53 genes in Ewing family tumors. Lab Invest. 2001; 81:803-14. [ Links ]
20. Thompson AD, Teitell MA, Arvand A, Denny CT. Divergent Ewing's sarcoma EWS/ETS fusions confer a common tumorigenic phenotype on NIH3T3 cells. Oncogene. 1999; 18:5506. [ Links ]
21. Kim TS, Heinlein C, Hackman RC, Nelson PS. Phenotypic analysis of mice lacking the Tmprss2-encoded protease. Mol Cell Biol. 2006; 26:965. [ Links ]
22. Murakami K, Mavrothalassitis G, Bhat NK, Fisher RJ, Papas TS. Human ERG-2 protein is a phosphorylated DNA-binding protein-a distinct member of the ets family. Oncogene. 1993; 8:1559-66. [ Links ]
23. Petrovics G, Liu A, Shaheduzzaman S, Furusato B, Sun C, Chen Y, et al. Frequent overexpression of ETS-related gene-1 (ERG1) in prostate cancer transcriptome. Oncogene. 2005; 24:3847-52. [ Links ]
24. Seth A, Watson DK. ETS transcription factors and their emerging roles in human cancer. Eur J Cancer. 2005; 41:2462-78. [ Links ]
25. Saramäki OR, Harjula AE, Martikainen PM, Vessella RL, Tammela TLJ, Visakorpi T. TMPRSS2: ERG fusion identifies a subgroup of prostate cancers with a favorable prognosis. Clin Cancer Res. 2008; 14:3395. [ Links ]
26. Yoshimoto M, Joshua AM, Chilton-Macneill S, Bayani J, Selvarajah S, Evans AJ, et al. Three-color FISH analysis of TMPRSS2/ERG fusions in prostate cancer indicates that genomic microdeletion of chromosome 21 is associated with rearrangement. Neoplasia. 2006; 8:465-9. [ Links ]
27. Graff JR, Herman JG, Lapidus RG, Chopra H, Xu R, Jarrard DF, et al. E-cadherin expression is silenced by DNA hypermethylation in human breast and prostate carcinomas. Cancer Res. 1995; 55:5195. [ Links ]
28. Quinn M, Babb P. Patterns and trends in prostate cancer incidence, survival, prevalence and mortality. En: Quinn P., Babb editors. Part I: international comparisons. Blackwell Science, Ltd; 2002. 162-73. [ Links ]
29. King JC, Xu J, Wongvipat J, Hieronymus H, Carver BS, Leung DH, et al. Cooperativity of TMPRSS2-ERG with PI3-kinase pathway activation in prostate oncogenesis. Nature Genetics. 2009; 41:524-6. [ Links ]
30. Rubio-Briones J, Fernández-Serra A, Calatrava A, García-Casado Z, Rubio L, Bonillo MA, et al. Clinical Implications of TMPRSS2-ERG Gene Fusion Expression in Patients With Prostate Cancer Treated With Radical Prostatectomy. J Urol. 2010; 183:2054-61. [ Links ]
31. Mao X, Shaw G, James SY, Purkis P, Kudahetti SC, Tsigani T, et al. Detection of TMPRSS2: ERG fusion gene in circulating prostate cancer cells. Asian J Androl. 2008; 10:467-73. [ Links ]
32. Setlur SR, Mertz KD, Hoshida Y, Demichelis F, Lupien M, Perner S, et al. Estrogen-dependent signaling in a molecularly distinct subclass of aggressive prostate cancer. JNCI. 2008; 100:815. [ Links ]
33. Mao X, Yu Y, Boyd LK, Ren G, Lin D, Chaplin T, et al. Distinct Genomic Alterations in Prostate Cancers in Chinese and Western Populations Suggest Alternative Pathways of Prostate Carcinogenesis. Cancer Res. 2010; 70:5207-12. [ Links ]
34. Iljin K, Wolf M, Edgren H, Gupta S, Kilpinen S, Skotheim RI, et al. TMPRSS2 fusions with oncogenic ETS factors in prostate cancer involve unbalanced genomic rearrangements and are associated with HDAC1 and epigenetic reprogramming. Cancer Res. 2006; 66:10242-6. [ Links ]
35. Hermans KG, Bressers AA, van der Korput HA, Dits NF, Jenster G, Trapman J. Two unique novel prostate-specific and androgen-regulated fusion partners of ETV4 in prostate cancer. Cancer Res. 2008; 68:3094. [ Links ]
36. Tomlins SA, Laxman B, Dhanasekaran SM, Helgeson BE, Cao X, Morris DS, et al. Distinct classes of chromosomal rearrangements create oncogenic ETS gene fusions in prostate cancer. Nature. 2007; 448:595-9. [ Links ]
37. Pflueger D, Rickman DS, Sboner A, Perner S, LaFargue CJ, Svensson MA, et al. N-myc downstream regulated gene 1 (NDRG1) is fused to ERG in prostate cancer. Neoplasia (New York, NY). 2009; 11:804. [ Links ]
38. Demichelis F, Fall K, Perner S, Andren O, Schmidt F, Setlur SR, et al. TMPRSS2: ERG gene fusion associated with lethal prostate cancer in a watchful waiting cohort. Oncogene. 2007; 26:4596-9. [ Links ]
39. Darnel AD, LaFargue CJ, Vollmer RT, Corcos J, Bismar TA. TMPRSS2-ERG fusion is frequently observed in Gleason pattern 3 prostate cancer in a Canadian cohort. Cancer Biol Ther. 2009; 8:125. [ Links ]
40. Saramäki OR, Harjula AE, Martikainen PM, Vessella RL, Tammela TLJ, Visakorpi T. TMPRSS2: ERG rearrangement is an independent marker of good prognosis in prostate cancer. Eur Urol Suppl. 2008; 14:3395-400. [ Links ]
![]() Dirección para correspondencia:
Dirección para correspondencia:
Correo electrónico: jalopez@fivo.org
(J.A. López-Guerrero)
Recibido 26 Octubre 2010
Aceptado 5 Noviembre 2010















