My SciELO
Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Cited by Google
Cited by Google -
 Similars in
SciELO
Similars in
SciELO -
 Similars in Google
Similars in Google
Share
Revista de la Asociación Española de Neuropsiquiatría
On-line version ISSN 2340-2733Print version ISSN 0211-5735
Rev. Asoc. Esp. Neuropsiq. n.84 Madrid Oct./Dec. 2002
ARTÍCULO ORIGINAL
Una revisión de la genética del autismo
A overview of the genetics os Autism
L. Alison Mcinnes
MD, Profesora Adjunta de Psiquiatría y Genética Humana Facultad de Medicina de Mount Sinaí Nueva York, EE.UU.
Dirección para correspondencia
RESUMEN
El autismo es un severo trastorno del desarrollo neuropsicológico, caracterizado por una alteración de la comunicación y de la interacción social, y por restringidas, repetitivas y obsesivas pautas de conducta. Afecta más a hombres que a mujeres y, con frecuencia, existe un bajo cociente intelectual. Recientes estimaciones de su prevalencia en California muestran un creciente aumento. Se piensa en posibles causas genéticas y ambientales con un carácter fuertemente hereditario. Existen progresos recientes en lo que respecta al aislamiento de los genes de susceptibilidad, habiéndose confirmado definitivamente los factores de predisposición no ambientales. En la presente revisión se exponen los más recientes hallazgos epidemiológicos, así como los resultados de los actuales estudios sobre la genética del trastorno.
Palabras clave: Autismo, Epidemiología, aumento de prevalencia, estudios genéticos.
ABSTRACT
Autism is a severe, neurodevelopmental disorder characterized by impairment in social interaction and communication, and by restricted, repetitive and obsessive patterns of behavior. The disorder affects males more fredquently than females and autistic subjects frequently exhibit reduced I.Q.S. Recent estimates of the prevalence of autism in California show that it is increasing. Investigators are looking into the possible genetic and environmental causes of autism. As the disorder is the most heritable psychiatric illness, investigators are making progress isolating susceptibility genes although no environmental factors predisposing to autism have yet been definitely confirmed. In this review I will discuss recent epidemiological findings and the results of current genetic studies.
Key words: autism, epidemiology, increased prevalence, genetic studies.
Introducción
Estudios epidemiológicos recientes sugieren que la incidencia de autismo y de trastornos generalizados del desarrollo (PDD, Pervasive Development Disorders), están aumentando en nuestra sociedad. Es posible que este incremento se deba a la aplicación de nuevos procedimientos de muestreo y técnicas de diagnóstico más precisas. En la actualidad se estima que la frecuencia de autismo es 1/1000, y la de PDD de 5/1000, cifras que delatan la importancia de este trastorno en la salud pública. La etiología de este trastorno es desconocida y el análisis genético es una herramienta que promete esperanzadora para ayudar a determinar cuales son las bases genéticas, neurológicas y ambientales que originan el autismo. En esta revisión describiré una visión global sobre datos recientes de estudios epidemiológicos y genéticos, haciendo hincapié en el hecho de que varias publicaciones independientes han obtenido resultados que asocian la enfermedad con determinadas regiones cromosómicas. Hasta la fecha no se conocen datos epidemiológicos y genéticos en poblaciones de origen hispano, por ello la autora y otros investigadores están llevando a cabo un proyecto de investigación en Andalucía que permitirá el avance en el conocimiento de esta enfermedad y ayudará a rectificar esta deficiencia en la confrontar los datos obtenidos con la información existente.
Epidemiología
En el momento de escribir este artículo existen en la literatura 32 publicaciones de estudios epidemiológicos sobre autismo que han sido revisados recientemente por Fombonne (1). Estos trabajos se han llevado a cabo en 13 países: EE.UU., Reino Unido, Japón, Francia, Suecia, Noruega, Finlandia, Islandia, Dinamarca, Indonesia, Alemania, Irlanda y Canadá. Las edades de los pacientes incluidos en los estudios eran desde su nacimiento hasta la edad adulta, con una media de 8 años. El tamaño de las poblaciones investigadas osciló entre 826 y 899.750, la media fue de 65.300. El número de pacientes autistas evaluados dentro de estas poblaciones estudiadas varió entre 6 y 427, con un valor medio de 51. En total se ha investigado una población de 5 millones de niños e identificándose un total de 2.380 casos de autismo. La razón varón / hembra fluctuó entre 1,.33 ay 16, con un promedio de 4,3.
La mayoría de los estudios se llevaron a cabo siguiendo dos etapas. La primera etapa consistió en la captación de pacientes mediante el envío de circulares y escalas diagnósticas a colegios y profesionales de la salud. Esta estrategia tiene el inconveniente de que puede quedar una parte de la población fuera del estudio, no se documenta las cifras de los casos que rehusan participar y puede existir entre los estudios una variabilidad en el desarrollo de los servicios y de los instrumentos diagnósticos. La segunda etapa consistió en la evaluación de los pacientes para identificar sus características fenotípicas. El ADI (Autism Diagnostic Interview (2)) y más recientemente el ADI-R (ADI-Revisado (3) se consideran instrumentos de diagnóstico esenciales para fines de investigación sobre el autismo, sin embargo solo se utilizaron en los últimos 5 estudios y en la mayoría de ellos no se evaluó la fiabilidad del procedimiento diagnóstico utilizado. Los criterios diagnósticos del ICD-10 ó del DSM III fueron utilizados a partir de 1990. En algunos casos se utilizaron criterios de diagnóstico ICD-10 ó DSMIII.
En algunos de los 32 estudios se evaluaron el IQ, la incidencia de epilepsia y una serie de afecciones clínicas que pueden estar relacionadas con el autismo. En 20 estudios se determinó el IQ, en el 30% de los pacientes tienen IQ eran normales, el 30% tenían un retraso leve a moderado y el 40 % restante mostró un retraso severo. En 9 estudios se determinó la incidencia de epilepsia, con una media del 16,8% (4,8% a 26,4%). Se observaron picos bimodales de epilepsia en la infancia y adolescencia, aunque los fenómenos siendo la epilepsia más frecuente se manifestaron con mayor frecuencia en aquellos pacientes que tenían un retraso de moderado a severo. En 14 estudios se investigaron afecciones clínicas asociadas al autismo, entre ellas rubéola congénita, fenilcetonuria, neurofibromatosis y cromosoma X frágil, destacando la esclerosis tuberosa que fue 100 veces más frecuente entre niños autistas que en la población general.
Croen et al (4,5), en un estudio realizado en California con 4.381 nacimientos desde 1989 a 1994, ponen de manifiesto un incremento en el factor de riesgo de autismo con la edad de la madre, su nivel cultural y con nacimientos múltiples1. Es de resaltar que en este estudio se sugiere que el riesgo de autismo es menor en México. Hasta la fecha no existen nuevos datos epidemiológicos que avalen estas observaciones.
La frecuencia de autismo detectada en las poblaciones humanas ha aumentado en los últimos 15 años. No se sabe si este aumento se debe a una mayor incidencia de este trastorno o a procesos de evaluación, selección y diagnóstico más precisos. Fombonne et al (1), ha documentado que en datos epidemiológicos entre 1966 y 1991 la frecuencia de autismo era de 4,4/10.000 y entre 1992 y 2001 de 12,7/10.000. Actualmente se acepta que la frecuencia de autismo en la población mundial es de 1/1000. Dentro del espectro autista, los trastornos generalizados del desarrollo no autistas (PDD no autistas), son más comunes que el autismo (15/10.000). Entre los PDD no autistas existen pocos estudios epidemiológicos, existiendo pocos datos sobre el Síndrome de Asperger (AS) debido a que el diagnóstico se añadió recientemente al DSM-IV e ICD-10. En los pocos estudios llevados a cabo, se ha estimado una frecuencia de 2,5/10.000, es decir una relación de autismo comparada con AS de 4:1. Con respecto al trastorno desintegrativo infantil (Childhood Desintegrative Disorder) se ha estimado una frecuencia muy baja, 1,7/100.000. Considerando todos los trastornos dentro del espectro autista (autismo+AS+PDDNOS) se ha estimado una frecuencia de 27,.5/10.000, aunque en el Reino Unido un estudio reciente ha estimado una incidencia de 62,6/10.00050006.
En relación con el aparente aumento de la incidencia de autismo (y tal vez PDD), ¿Cómo podemos determinar que dicho aumento es real? Una de las mejores maneras de hacerlo es analizar la frecuencia de autismo en la misma zona geográfica, en períodos sucesivos, y utilizando los mismos protocolos de diagnóstico. La autora y sus colegas de la Universidad de Córdoba y del Hospital Universitario Reina Sofía de Córdoba, intentan obtener dicha información en un estudio de epidemiología genética en Andalucía. Son varias las ventajas que se presentan para llevar a cabo este estudio. Primero, es que los servicios de salud mental de Andalucía están coordinados por un ente común, el Servicio Andaluz de Salud (SAS), existiendo unidades de salud mental en cada una de las 8 provincias andaluzas. Segundo, todas las historias clínicas están centralizadas y próximamente se informatizarán, facilitando así el acceso a los datos y por tanto el proyecto de investigación. Tercero, casi el 100% de los casos diagnosticados con autismo son evaluados o están registrados en el mismo sistema de salud. Por esta razón existe la posibilidad de comparar el número de casos de autismo en años sucesivos, y determinar de una forma real si existe aumento de la incidencia de trastornos generalizados del desarrollo. Además como todos los niños con trastornos de desarrollo se someten a un análisis de cromosoma X frágil, se podrá hacer una estimación precisa de los casos de autismo asociados a esta anomalía. Si la frecuencia de autismo en Andalucía es distinta que en otras poblaciones, sería interesante investigar factores genéticos y ambientales que pudieran originar tal diferencia. La autora también está estudiando al mismo tiempo los casos de autismo en una población aislada del Valle Central de Costa Rica (CVCR) y en la zona oriental de Guatemala. La población del VCCR fue fundada en el siglo XVI, principalmente por inmigrantes del sur de España, lo que supone una oportunidad excepcional para determinar genes asociados al autismo con las muestras del VCCR y Andalucía.
Diseño de estudios genéticos:
Los estudios genéticos diseñados para detectar los genes que contribuyen a los problemas severos de comportamiento son en general de dos tipos: estudios de población o estudios familiares. En los estudios de población, los casos individuales (pacientes) son seleccionados de la población general y su genoma es investigado con intervalos de espacios estrechos para obtener el locus genético que podría estar asociado a la enfermedad o ser causante de la misma. El genoma debe ser investigado a intervalos estrechos porque los individuos estudiados están relacionados en forma distante, de tal manera que el DNA que comparten en las áreas alrededor del gene de la enfermedad es realmente pequeño ya que los cromosomas se recombinan en función de la distancia entre cada generación (también denominada paso meiótico) demonstrado en la figura 1. El mapeo por asociación o desequilibrio es el proceso de búsqueda de esta región de DNA que es compartida alrededor del gen de la enfermedad "idénticamente por descendiente"(identically by descendent = IBD).
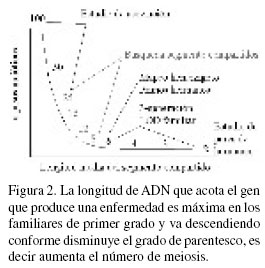
En los casos seleccionados para los estudios familiares se explora la posibilidad de que otros miembros también sufran del mismo trastorno. Hasta ahora todas las mayores investigaciones sobre el autismo han sido estudios familiares los cuales han investigado específicamente a parejas de hermanos en familias en donde por lo menos dos hermanos tienen autismo. Los resultados genéticos en dichos estudios son analizados utilizando el análisis de ligamiento que determina un puntaje de LOD. A pesar de que este método puede ser de utilidad para proveer una localización amplia del gene de la enfermedad, tiene como desventaja el hecho de que tal vez hay que estudiar cientos de genes dentro del mismo intervalo (Figura 2). Es por ello que la autora cree que la combinación de estudios genéticos familiares y estudios genéticos de población será el mejor método para encontrar los genes del autismo.
Análisis genéticos sobre el autismo..
Hasta la fecha se han realizado 7 estudios con hermanos autistas de varios grupos étnicos para determinar loci asociados al autismo. Los marcadores utilizados estaban separados 10 cM y abarcaban todo el genoma humano. Todos los estudios utilizaron el ADI-R como instrumento diagnóstico. La determinación fenotípica de los pacientes autistas se realizó mediante el ADI-R y con muy pocas variaciones entre los distintos estudios. Se ha encontrado ligamiento del autismo a varios loci, especialmente en los cromosomas 2 y 7. Silverman et a (7) han propuesto que el retraso en el lenguaje de frases (PSD, del inglés, Phrase Speech Delay), el cual se define como la falta de adquisición del lenguaje de frases antes de los 36 meses de edad, es un endofenotipo del autismo muy heredable, si se pone como tope de esta capacidad 36 meses de edad. Este hecho hace que en las familias con niños autistas se determine la presencia de PSD en otros miembros de la misma.
Mapeo genético.
En 1998, el International Genetic Study of Autism Consortium (IMGSAC), publicó los primeros datos asociando determinadas regiones cromosómicas a la enfermedad(8). Los resultados que más se destacaban en un grupo de 56 parejas de hermanos del Reino Unido eran los del ligamiento de los marcadores D7S530 y D7S684 de la región 7q con un valor multipuntaje máximo de LOD de 3,55, en un grupo de 56 parejas de hermanos en el Reino Unido. El gen SPCH1, que está asociado a un trastorno específico del desarrollo del habla y lenguaje (SDDSL), y que afecta al lenguaje expresivo, se localizó también en esta región, en la posición 7q31 (9,10). Posteriormente, Ashley-Koch et al. (11) estudiando una familia de tres hermanos (dos varones autistas y una niña con trastorno de lenguaje expresivo), identificaron que los tres heredaron una inversión paracéntrica en el cromosoma 7 en la región anteriormente aludida. Warburton et al. (12). observaron reestructuraciones en la región 7q31 en dos individuos no emparentados, uno con autismo y otro con SDDSL. Petek et al. (13) también encontraron una duplicación en 7q31 en un varón de 13 años con el Síndrome de Tourette y con retraso del desarrollo del lenguaje. Esta concordancia de observaciones relacionadas con trastornos del lenguaje sugiere que un locus en el cromosoma 7, que actualmente se le denomina AUTS1, contiene un gen que contribuye al menos a un aspecto fenotípico del autismo. Desgraciadamente el gen responsable no se ha identificado todavía a pesar de los múltiple intentos para lograrlo. El gen SPCH1 fue clonado y caracterizado recientemente (14) y no parece estar asociado con el autismo (15).
Wassink et al. (16), analizaron el gen WNT2 que también se encuentra en la región 7q, encontrando dos cambios de la secuencia de aminoácidos en individuos autistas en dos familias distintas. También se tipificaron SNPs (single nucleotide polimorfism) en regiones reguladoras y se obtuvo un resultado que indicaba una tendencia a la transmisión de un alelo preferente en un grupo de hermanos con PSD (Phrase Speech Delay). Otros resultados sin embargo no han encontrado una asociación entre mutaciones en el gen WNT2 y el autismo (McCoy et al. 2002) (17). Bonora et al. (18), analizaron cuatro genes (PEF1/MEST, COPG2, CPA1 y CPA5) en la región 7q32 sin encontrar ningún resultado significativo. Puesto que la región candidata del cromosoma 7, que va de 7q22 a 7q34, tiene unos 50Mb y se estima que contiene más de 190 genes, la tarea que queda por hacer es considerable.
En un estudio de colaboración sobre genética de autismo, denominado Collaborative Linkage Study of Autism (CLSA), en el que se evaluaron 75 parejas de hemanos (19), se encontraron asociaciones de la enfermedad a tres marcadores, dos en el cromosoma 13 y uno en el 7. Posteriormente utilizando el carácter PSD como endofenotipo1(20,21), se profundizó en la región del cromosoma 7 dividiendo a las 75 familias en dos subgrupos, uno de 50 en la que ambos sujetos tenían PSD y otro de 25 donde uno o ambos sujetos tenían un comportamiento normal en cuanto a la edad en que adquirieronían la capacidad de elaboración de frases. El LOD determinado en el grupo de las 50 familias, considerando el marcador D7S1813 fue de 2,77, superior al calculado anteriormente para las 75 familias (2,2). El LOD para el grupo de 25 familias con lenguaje normal fue de 0. También se analizó si los padres presentaron PSD y/o anomalías a la hora de aprender a leer y dificultades paraa deletrear. No hubo diferencias entre el grupo de 50 y de 25, lo cual no es lo que se esperaría si los trastornos en el lenguaje sones realmente un endofenotipo dentro del autismo.
Phillipe et al (22), publicaron los resultados de un análisis genético llevado a cabo con un grupo de 51 parejas de hermanos autistas. En esta población obtuvieron una asociación de la enfermedad con una región del cromosona 6 (6q21) y sóolo una relación moderadamente positiva con la región 7q. Otras regiones cromosómicas con valores positivos se localizaban en los cromosomas 18, (this was not included in your original version), 16romosomas 18, 16, 19 y 2. Recientemente, profundizando en la región 6q21, se ha detectado una mutación en el gen GLUR6 en algunos sujetos autistas (23). GLUR6 codifica un receptor de glutamato, la mutación consiste en el cambio de una aminoácido en un dominio muy conservado de la proteína. La mutación está presente en el 8% de individuos autistas en comparación con el 4% en la población control.
En otro estudio, Risch et al. (24) llevaron a cabo un escrutinio genómico en un total de 147 parejas de hermanos repartidos en dos grupos, uno en los que ambos hermanos estaban afectados y otro de control en los que uno era autista y el otro no. Estos autores concluyeron que en el autismo puede haber implicados unos 10 loci. De sus resultados se desprende que uno de ellos estaría situado en el cromosoma 1p 1p donde el marcador D1S1631 localizado en 1p, dio un valor de Z de 3,44. Otras tres regiones que dieron resultados positivos se localizaban en el cromosoma 18, coincidiendo con los resultados de Phillipe et al. (22), y en 7q y 13q.
Bauxbaum et al. (25) han presentado resultados que asocian ligamiento de autismo en la región del cromosoma 2q utilizando los marcadores D2S364 y D2S335. Al restringir el análisis a un grupo de familias (N=49) con dos o más individuos con diagnóstico de autismo y PSD, obtuvieron un valor de MMHLOD (here you have MMHLOD) más alto. Phillipe et al. (22) también encontraron valores LOD positivos con el marcador D2S364 y otro próximo a él.
El IMGSAC (26) llevó a cabo un escrutinio completo del genoma en 83 parejas de hermanos autistas. Se establecieron 1913 regiones candidatas y con ellas en el punto de mira realizaron un segundo estudio con 69 hermanos autistas adicionales. Los datos más destacados apuntaron a una región en el cromosoma 2, la misma región 2q propuesta por Buxbaum et al. (25). El ligamiento del marcador D2S2188 aumentó cuando restringieron la muestra a parejas de hermanos con una definición fenotípica estricta (criterio ADI-R con uno o ambos sujetos con retraso en el inicio del habla). De este estudio también se obtuvo ligamiento de la enfermedad con las regiones 7q y 16p, y algo menor en el cromosoma 17.
Liu et al. (27) seleccionaron 110 familias del AGRE (Autism Genetic Resource Exchange) y obtuvieron resultados que apoyaban ligamientos de la enfermedad a marcadores situados en los cromosomas 5, X y 19 con ciertos indicios de epistasia (define) entre e epistasia entre loci situados en los cromosomas X y 19.
Shao et al (28) realizaron un escrutinio del genoma en dos etapas, en la primera analizaron 52 familias en las que determinaron regiones concretas del genoma con posibilidades de tener loci implicados y en la segunda etapa añadieron al estudio 52 (you have 47) familias para analizar las regiones candidatas de forma más concreta. Encontraron una región susceptible en el brazo corto del cromosoma 3p. Otras regiones con posibilidades se localizaban en los cromosomas 2, 7, 19 y X que coincidían con los resultados de otros estudios. Un subgrupo de 45 parejas de hermanos con PSD, lo analizaron incluyendo marcadores del cromosoma 2 utilizados por Buxbaum et al. (25), obteniéndose resultados de mayor ligamiento en 2q (Shao et al (28). Combinando los datos de ambos grupos se puede concluir que la región de 2q susceptibles a contener loci implicados es de 35 MB y contiene más de 100 genes. Es interesante destacar que se ha determinado que en las regiones 2q y 7q hay secuencias homólogas, lo que podría indicar que los genes relacionados con el autismo presentes en estas regiones podrían estar funcionalmente relacionados.
Regiones con genes candidatos: Cromo-soma 15q11-q13 y el transportador de serotonina.
Algunas alteraciones cromosómicas identificadas mediante técnicas citogenéticas están asociadas con síndromes heredados. Existen datos que confirman que determinados pacientes autistas presentan alteraciones cromosómicas en la región 15q11-q13 con más frecuencia que en la población en general (Gillberg et al. (30,31), Cook et al. (32). En estas anormalidades se incluye el cromosoma 15 dicéntrico y supernumerario, duplicaciones de origen materno en la región 15q11-q13 y deleciones genómicas sub-microscópicas en dicha región. Esta región cromosómica puede asociarse al autismo, aunque no está presente siempre en todos los individuos autistas de una misma familia (Bailey et al (32). El síndrome de Angelman, un trastorno neurológico que tiene en parte características fenotípicas similares al autismo, parece originarse como consecuencia de la falta de expresión de un gen o genes maternos en la región 15q11-q13. En individuos con el síndrome de Angelman se han detectado en esta región mutaciones en el gen UBE3A, que codifica la E6-AP proteína ubiquitina ligasa, pero Veenstra-VanderWeele et al. (34) tras un minucioso escrutinio no encontraron mutaciones de este gen en pacientes autistas. Otros genes de interés que se encuentran en la región 15q11-q13 están relacionados con receptores del ácido gamma-aminobutírico. Cook et al. (35), investigaron el locus GABAAβ3 utilizando 9 marcadores en 140 familias con un miembro autista, encontrando asociación de la enfermedad con un solo marcador (GABRB3 155CA-2). Recientemente, Buxbaum et al. (36) utilizaron este marcador para estudiar ligamiento del mismo a la enfermedad en 80 familias obteniendo resultados similares. En contraste, Salmon et al (37) no encontraron resultados positivos de asociación entre autismo y este locus, analizando de forma detallada 147 parejas de hermanos autistas. Otros estudios llevados a cabo por el CLSA ó IMGSAC tampoco encontraron asociación del autismo con el cromosoma 15.
Se ha determinado en varias ocasiones que los niveles de serotonina en plaquetas de individuos autistas es superior que en el resto de la población. El tratamiento a base de inhibidores de la recaptación de serotonina ha aliviado en algunos casos de forma moderada determinados síntomas del autismo. Este resultado ha llevado a analizar el gen que codifica la proteína transportadora de este neurotransmisor. Cook et al. (38) estudiando 86 familias autistas con una única persona afectada, no encontraron ligamiento entre autismo y polimorfismo (secuencias VNTR) en el segundo intrón del gen que codifica el transportador de serotonina, pero sí observaron un incremento de la frecuencia de la variante corta de un polimorfismo existente en el promotor de dicho gen (5HTTLPR). Resultados contradictorios fueron observados por Klauck et al.(39) que detectó una asociación débil del autismo con la variante larga y la ausencia de polimorfismo en las familias catalogadas en el IMGSAC. Otros estudios también han dado resultados negativos (40). Utilizando otras estrategias, Kim et al. (41) han propuesto que el autismo podría estar relacionado con otras mutaciones que afectan al transportador de la serotonina. Se espera que otros grupos confirmen estos descubrimientos.
Conclusiones
Es muy posible que en un futuro cercano se descubran genes implicados en la etiología del autismo. No obstante, puede ser que algunas mutaciones residan en secuencias no codificantes, o/y que ocurran varias mutaciones con baja frecuencia en diferentes loci y que contribuyan en varias combinaciones al fenotipo, en tal caso se tardará bastante tiempo en determinar y comprobar la naturaleza de las mismas. mismas. No obstante puede ser que algunas mutaciones residan en secuencias no codificantes o que múltiples mutaciones de baja frecuencia ocurran dentro del loci, en tal caso se tardará bastante tiempo en comprobar que éstas contribuyen al fenotipo. No obstante puede ser que algunas mutaciones residan en secuencias no codificantes o que ocurran múltiples mutaciones con baja frecuencia, en tal caso se tardará bastante tiempo en comprobar que éstas constribuyen al fenotipo. El estudio de muestras de gran tamaño y de poblaciones aisladas junto con un enfoque que se base en la comparación de genomas para detectar polimorfismos asociados a la enfermedad, permitirá determinar tanto la implicación de secuencias codificantes como no codificantes. El lector interesado en profundizar en este tema puede dirigirse a una revisión exhaustiva y reciente sobre la genética de autismo, donde se incluyen tablas y figuras, que ha sido publicada recientemente en Nature Reviews Genetics (42).
Notas
1Nota añadida en fase de prueba: Recientemente Byrd y otros han informado que la incidencia y la prevalencia se han incrementado en California. Nuevas estimaciones de la prevalencia dan cifras de 10-12/1000 en esta población. http://mindinstitute.ucdavis.edu/news/1exec_summ.pdf
Bibliografía
1. Fombonne E. "Epidemiological trends in rates of autism". Mol Psychiatry. 2002;7 Suppl 2:S4-6. [ Links ]
2. Le Couteur A, Rutter M, Lord C, Rios P, Robertson S, Holdgrafer M, McLennanJ. "Autism diagnostic interview: a standardized investigator-based instrument". J Autism Dev Disord. 1989 Sep;19(3):363-87. [ Links ]
3. Lord C, Rutter M, Le Couteur A. "Autism Diagnostic Interview-Revised: a revised version of a diagnostic interview for caregivers of individuals with possibsiblingle pervasive developmental disorders". J Aut Develop Disord. 1994;24:659-685. [ Links ]
4. Croen LA, Grether JK, Selvin S. "Descriptive epidemiology of autism in a California population: who is at risk?" J Autism Dev Disord. 2002 Jun;32(3):217-24. [ Links ]
5. Croen LA, Grether JK, Hoogstrate J, Selvin S. "The changing prevalence of autism in California". J Autism Dev Disord. 2002 Jun;32(3):207-15. [ Links ]
6. Chakrabarti S, Fombonne E. "Pervasive developmental disorders in preschool children". JAMA. 2001 Jun 27;285(24):3093-9. [ Links ]
7. Silverman, J.M., Smith, C.J., Schmeidler, J., Hollander, E., Lawlor, B.A., Fitzgerald, M., Buxbaum, J.D., Delaney, K., Galvin, P. "Symptom domains in autism and related conditions: evidence for familiality". American Journal of Medical Genetics, 2002, 114(1) 64-73. [ Links ]
8. International Molecular Genetic Study of Autism Consortium. A full genome screen for autism with evidence for linkage to a region on chromosome 7q. Human Molecular Genetics. 1998;7:571–578. [ Links ]
9. Fisher SE, Vargha-Khadem F, Watkins KE, Monaco AP, Pembrey ME. "Localisation of a gene implicated in a severe speech and language disorder". Nature Genetics. 1998;18:168-170. [ Links ]
10. Lai, C.S.; Fisher, S.E.; Hurst, J.A.; Levy, E.R.; Hodgson, S.; Fox, M.; Jeremiah, S.; Povey, S.; Jamison, D.C.; Green, E.D.;C.S., Fisher, S.E., Hurst, J.A., Levy, E.R., Hodgson, S., Fox, M., Jeremiah, S., Povey, S., Jamison, D.C., Green, E.D., Vargha-Khadem, .F;F, Monaco, A.P. " The SPCH1 region on human 7q31: genomic characterization of the critical interval and localization of translocations associated with speech and language disorder". American Journal of Human Genetics, 2000, 67(2):357-368. [ Links ]
11. Ashley-Koch A, Wolpert CM, Menold MM, et al. "Genetic studies of autistic disorder and chromosome 7". Genomics. 1999;61:227–236. [ Links ]
12. Warburton P, Baird G, Chen W, et al. "Support for linkage of autism and specific language impairment to 7q3 from two chromosome rearrangements involving band 7q31". Am J Med Genet. 2000;96:228-234. [ Links ]
13. Petek E, Windpassinger C, Vincent JB, et al. "Disruption of a novel gene (IMMP2L) by a breakpointin 7q31 associated with Tourette syndrome". Am J Hum Genet. 2001:68(4):848-858. [ Links ]
14. Lai CS, Fisher SE, Hurst JA, Vargha-Khadem F, Monaco AP. "A forkhead-domain gene is mutated in a severe speech and language disorder". Nature. 2001 Oct 4;413(6855):519-23. [ Links ]
15. D. F. Newbury, E. Bonora, J. A. Lamb, S. E. Fisher, C. S. L. Lai, G. Baird, L. Jannoun, V. Slonims, C. M. Stott, M. J. Merricks, P. F. Bolton, A. J. Bailey, A. P. Monaco, and the International Molecular Genetic Study of Autism Consortium. "FOXP2 Is Not a Major Susceptibility Gene for Autism or Specific Language Impairment". American Journal of Human Genetics, 2002, 70(5):1318-1327. [ Links ]
16. Wassink TH, Piven J, Vieland VJ, Huang J, Swiderski RE, Pietila J, Braun T, Beck G, Folstein SE, Haines JL, Sheffield VC. "Evidence supporting WNT2 as an autism susceptibility gene". Am J Med Genet. 2001 Jul 8;105(5):406-13. [ Links ]
17. McCoy PA, Shao Y, Wolpert CM, Donnelly SL, Ashley-Koch A, Abel HL, Ravan SA, Abramson RK, Wright HH, DeLong GR, Cuccaro ML, Gilbert JR, Pericak-Vance MA."No association between the WNT2 gene and autistic disorder". Am J Med Genet. 2002 Jan 8;114(1):106-109. [ Links ]
18. Bonora E, Bacchelli E, Levy ER, Blasi F, Marlow A, Monaco AP, Maestrini E. "Mutation screening and imprinting analysis of four candidate genes for autism in the 7q32 region". Mol Psychiatry. 2002;7(3):289-301. [ Links ]
19. Barrett S, Beck JC, Bernier R, et al. "An autosomal genomic screen for autism. Collaborative Linkage Study of Autism". Am J Med Genet. 1999;88:609–615. [ Links ]
20. Folstein SE, Mankoski RE. "Chromosome 7q: where autism meets language disorder?" Am J Hum Genet. 2000:67:278-281. [ Links ]
21. Bradford Y, Haines J, Hutcheson H, et al. "Incorporating language phenotypes strengthens evidence of linkage to autism". Am J Med Genet (Neuropsychiatric Genetics). 2001;105:539-547. [ Links ]
22. Philippe A, Martinez M, Guilloud-Bataille M, et al. "Genome-wide scan for autism susceptibility genes. Paris Autism Research International SibSiblingpair Study". Hum Molecular Genet. 1999:8:805–812. [ Links ]
23. Jamain S, Betancur C, Quach H, Philippe A, Fellous M, Giros B, Gillberg C, Leboyer M, Bourgeron T. "Linkage and association of the glutamate receptor 6 gene with autism".Mol Psychiatry. 2002;7(3):302-10. [ Links ]
24. Risch, N., Spiker, D., Lotspeich, L., Nouri, N., Hinds, D., Hallmayer, J., Kalaydjieva, L., McCague, P., Dimiceli, S., Pitts, T. et al. "(1999) A genomic screen of autism: evidence for a multilocus etiology". American Journal of Human Genetics, 1999, 65:493–507. [ Links ]
25. Buxbaum JD, Silverman JM, Smith CJ, et al. "Evidence for a susceptibility gene for autism on chromosome 2 and for genetic heterogeneity". Am J Hum Genet. 2001;68:1514-1520. [ Links ]
26. International Molecular Genetic Study of Autism Consortium. Search for autism susceptibility loci: genome screen follow-up and fine-mapping of a candidate region on chromosome 7q. Presented at Collegium Internationale Neuro-Psychopharamacologium 22nd Congress, Brussels, July 9-13, 2000. International Molecular Genetic Study of Autism Consortium. m A genomewide screen for autism: strong evidence for linkage to chromosomes 2q, 7q, and 16p. American Journal of Human Genetics, 2001, 69:570-581. http://www.well.ox.ac.uk/ ~maestrin/news2000.html [ Links ]
27. Liu J, Nyholt DR, Magnussen P, et al. A "genomewide screen for autism susceptibility loci". Am J Hum Genet. 2001;69:327-340. [ Links ]
28. Shao Y, Wolpert CM, Raiford KL, Menold MM, Donnelly SL, Ravan SA, Bass MP, McClain C, von Wendt L, Vance JM, Abramson RH, Wright HH, Ashley-Koch A, Gilbert JR, DeLong RG, Cuccaro ML, Pericak-Vance MA. "Genomic screen and follow-up analysis for autistic disorder". Am J Med Genet. 2002 Jan 8;114(1):99-105. [ Links ]
29. Shao Y, Raiford KL, Wolpert CM, Cope HA, Ravan SA, Ashley-Koch AA, Abramson RK, Wright HH, DeLong R, Gilbert JR, Cuccaro ML, Pericak-Vance MA. "Phenotypic homogeneity provides increased support for linkage on chromosome 2 in autistic disorder". Am J Hum Genet. 2002 Apr;70(4):1058-61. [ Links ]
30. Gillberg, C. (1998) "Chromosomal disorders and autism". Journal of Autism and Developmental Disorders, 28:415–425. [ Links ]
31. Gillberg C, Steffenburg S, Wahlstrom J, et al. "Autism associated with marker chromosome". J Am Acad Child Adolesc Psychiatry. 1991;30:489–494. [ Links ]
32. Cook EH Jr, Lindgren V, Leventhal BL, et al. "Autism or atypical autism in maternally but not paternally derived proximal 15q duplication". Am J Hum Gen. 1997;60:928–934. [ Links ]
33. Lamb JA, Moore J, Bailey A, Monaco AP. "Autism: recent molecular genetic advances". Human Molecular Genetics. 2000;9:861-868. [ Links ]
34. Veenstra-VanderWeele, J., Gonen, D., Leventhal, B.L., Cook, E.H. Jr. (1999) "Mutation screening of the UBE3A/E6-AP gene in autistic disorder". Molecular Psychiatry, 4(1):64-67. [ Links ]
35. Cook EH Jr, Courchesne RY, Cox NJ, et al. "Linkage-disequilibrium mapping of autistic disorder, with 15q11–13 markers". Am J Hum Gen. 1998;62:1077–1083. [ Links ]
36. Buxbaum JD, Silverman JM, Smith CJ, Greenberg DA, Kilifarski M, Reichert J, Cook EH Jr,Fang Y, Song CY, Vitale R. " Association between a GABRB3 polymorphism and autism". Mol Psychiatry. 2002;7(3):311-6. [ Links ]
37. Salmon B, Hallmayer J, Rogers T, et al. "Absence of linkage and linkage disequilibrium to chromosome 15q11–q13 markers in 139 multiplex families with autism". Am J Med Genet. 1999;88:551–556. [ Links ]
38. Cook EH Jr, Courchesne R, Lord C, et al. "Evidence of linkage between the serotonin transporter and autistic disorder". Mol Psychiatry. 1997;2:247-250. [ Links ]
39. Klauck SM, Poustka F, Benner A, Lesch KP, Poustka A. "Serotonin transporter (5-HTT) gene variants associated with autism?" Hum Molec Genet. 1997;6:2233-2238. [ Links ]
40. Zhong N, Ye L, Ju W, Brown WT, Tsiouris J, Cohen I. "5-HTTLPR variants not associated with autistic spectrum disorders". Neurogenetics. 1999;2:129–131. [ Links ]
41. Kim S-J., Cox N., Courchesne R., Lord C., Corsello C., Akshoomoff N., Guter S., Leventhal B.L., Courchesne E., Cook Jr E.H., Correspondence: E H Cook Jr, MD* "Transmission disequilibrium mapping at the serotonin transporter gene (SLC6A4) region in autistic disorder". Molecular Psychiatry, 2002, 7(3) 278-288. [ Links ]
42. Folstein SE, Rosen-Sheidley B. "Genetics of Autism: Complex aetiology for a heterogeneous disorder". Nature Reviews Genetics, 2001 (2): 943-955. [ Links ]
![]() Dirección para correspondencia:
Dirección para correspondencia:
Correo electrónico: Alison.mcinnes@mssm.edu
Recibido: 20-06-02