INTRODUCCIÓN
El estado nutricional de un individuo es, principalmente, el resultado de su conducta alimentaria, pero también de sus ritmos circadianos. Estos corresponden a las variaciones periódicas de 24 horas que son controladas por el sistema nervioso central y que regulan los ciclos luz-oscuridad (1).
Entre los genes que están asociados a la regulación de los ritmos circadianos, tenemos al circadian locomotor output cycles kapu (CLOCK), el cual sincroniza el ciclo luz-oscuridad con los tiempos de ingesta-ayuno y de actividad-reposo en los individuos (2).
En la actualidad, se ha descrito un gran número de variantes genéticas del tipo polimorfismo de nucleótido único (single nucleotide polymorphisms [SNP]) del gen CLOCK, entre las cuales está el rs3749474T/C (3). Los portadores del alelo T de este polimorfismo presentan una mayor obesidad abdominal (circunferencia de cintura), un alto índice de masa corporal (IMC) y un elevado consumo de energía, principalmente a partir de ácidos grasos, en comparación con sujetos homocigotos CC del mismo polimorfismo (4-6).
Por otro lado, se ha establecido que quienes poseen los genotipos TT y TC de rs3749474 tienden a tener una mejor pérdida de peso después de un tratamiento que implica la restricción de grasas en la dieta, en comparación con quienes poseen el homocigoto de tipo CC (7). Además, quienes presentan los polimorfismos en homocigosis o heterocigosis del rs3749474 y suelen tener un hábito de ingesta nocturna de carbohidratos y poseen un IMC más alto que la población general (8). Se ha demostrado que existe una asociación entre el ser portador de los alelos menores de rs3749474 del gen CLOCK y la ingesta de energía, lo cual conllevaría a aumentar el riesgo de desarrollar obesidad (5).
La figura 1 representa el modelo de determinación de los genotipos rs3749474 en relación con el IMC. Mientras que los portadores del alelo T, es decir TT y CT, presentan una asociación directamente proporcional entre consumo de carbohidratos (y lípidos) e IMC, los que presentan el genotipo CC poseen una leve asociación inversa, o bien ausencia de asociación, dependiendo del estudio y la población analizada. La relación causal entre los genotipos portadores del alelo T y el incremento del IMC es, entonces, un factor de riesgo de obesidad. Como consecuencia de lo anterior, los individuos portadores del alelo T presentan una mejor respuesta ante la disminución de carbohidratos y lípidos en la dieta, disminuyendo el IMC (8).
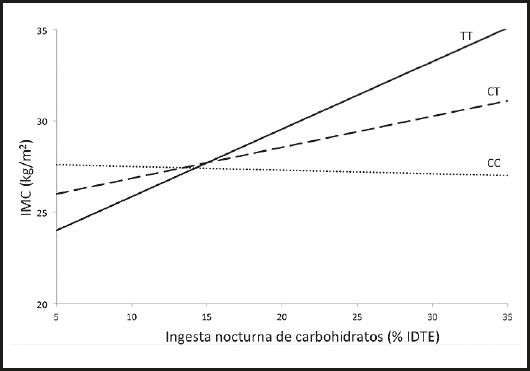
Figura 1. Modelo de interacción de los genotipos del SNP CLOCK rs3749474 en relación con la ingesta de nutrientes, basado en Camblor y cols. (8). IMC: índice de masa corporal; IDTE: ingesta diaria total de energía.
Dado lo anterior, resulta relevante conocer la incidencia de los genotipos portadores del alelo T en diferentes poblaciones. El alelo C es el alelo ancestral, por lo que se espera se presente en mayor frecuencia en poblaciones africanas y en menor frecuencia en otras poblaciones, especialmente las más recientes, como la latinoamericana. No obstante, factores microevolutivos ocurridos en la historia de las poblaciones humanas podrían haber producido diferentes patrones de variabilidad en este marcador genético.
En este estudio analizamos el patrón de variabilidad de los genotipos portadores del alelo T del marcador CLOCK rs3749474 en poblaciones humanas actuales, lo que podría aportar evidencia de base para futuros estudios biomédicos y epidemiológicos, así como para la implementación de pruebas genéticas-clínicas para medidas preventivas e intervenciones clínicas personalizadas.
MATERIAL Y MÉTODOS
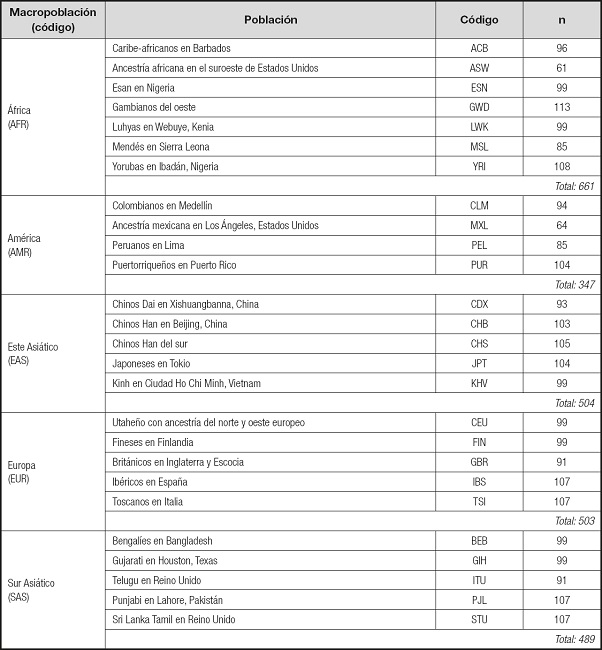
Se obtuvo el genotipo del SNP rs3749474 de 2.504 individuos a partir de la base de datos 1000 Genomes (9). Para esto, se descargó el total de la base de datos en su fase 3, para luego extraer los genotipos de rs3749474 para cada individuo usando la herramienta VCFtools. Esta base de datos abarca 2.504 individuos de 26 poblaciones. Los datos de baja cobertura (4x) están presentes para todos estos individuos, así como los de 24 individuos con alta cobertura (50x), con fines de validación. La muestra incluyó las siguientes macropoblaciones: África, Este Asiático, Sur Asiático, Europa y Latinoamérica, abarcando en total 26 poblaciones, cuyo detalle se muestra en la tabla I. El porcentaje de mujeres en la muestra es del 50,9 %. No obstante, variables como la edad y el sexo de los participantes no influyen en los resultados, pues rs3749474 es un marcador genético autosomal.
ANÁLISIS ESTADÍSTICO
El genotipo CC se codificó como genotipo de riesgo (1), mientras que los otros dos (CT y TT) se codificaron como de no riesgo (0), esto considerando que el alelo T confiere una mejor respuesta a una dieta baja en carbohidratos (8). Se estimó la frecuencia del genotipo de riesgo en las cinco macropoblaciones y se compararon sus frecuencias usando la prueba exacta de Fisher.
Tanto la construcción de la base de datos (disponible como material suplementario) como la estadística descriptiva (estimación de frecuencias) y la estadística inferencial (comparación de frecuencias) se realizaron con el programa R (10).
RESULTADOS
A continuación, se presentan los resultados según distribución de las cinco macropoblaciones relevantes (Fig. 2). La menor frecuencia de genotipos portadores del alelo T se encuentra en África, seguida de Europa, Latinoamérica, Sur Asiático y Este Asiático, en orden creciente. Es notoria la baja frecuencia observada entre África y las otras cuatro macropoblaciones.
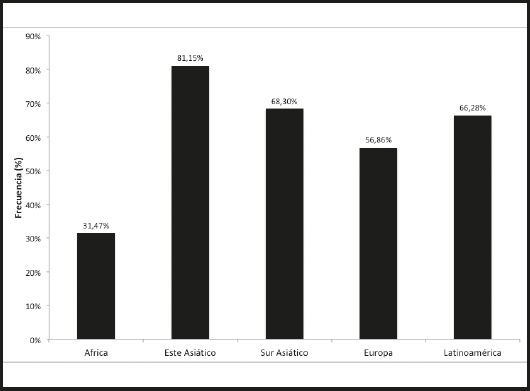
Figura 2. Frecuencia de genotipos portadores del alelo T (TT y CT) en las cinco macropoblaciones de este estudio.
Al comparar las frecuencias genotípicas entre todos los pares de poblaciones, se confirma la alta divergencia de África. Además, existen diferencias significativas (pFisher < 0,05) en todas las comparaciones excepto entre Latinoamérica y Sur Asiático (Tabla II).
Tabla II. Prueba exacta de Fisher para la comparación del número de genotipos de riesgo (TT + CT) y no riesgo (CC) entre pares de macropoblaciones.
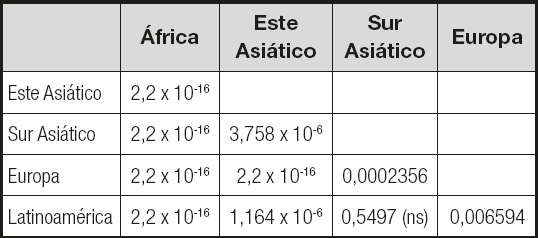
ns: no significativo.
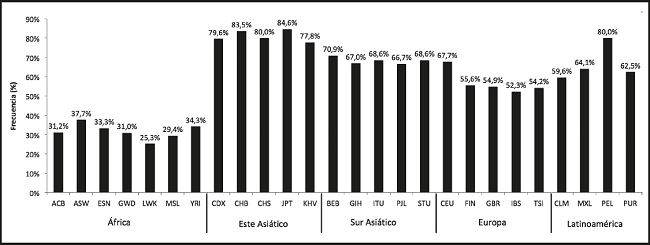
La figura 3 muestra las frecuencias de genotipos portadores del alelo T en las 26 poblaciones de este estudio. Se destaca la alta homogeneidad de las frecuencias dentro de cada macropoblación. La excepción a este patrón corresponde a Lima, Perú, que muestra una mayor frecuencia de genotipos de no riesgo en relación con las otras poblaciones latinoamericanas (pFisher < 0,05).
DISCUSIÓN
Nuestros resultados muestran una alta divergencia para la frecuencia de genotipos portadores del alelo T en África, en relación con las otras cuatro macropoblaciones. Mientras que en África esta frecuencia alcanza solo el 31,47 %, en las restantes macropoblaciones esta frecuencia varía entre el 56,86 % (Europa) y el 81,15 % (Este Asiático). Por lo tanto, si nos guiamos por los resultados obtenidos por Camblor-Murube y cols. (8), se podría predecir que poblaciones africanas presentarían menor aumento del IMC debido al consumo de carbohidratos y, al mismo tiempo, menor respuesta ante dietas bajas en ellos. Por otra parte, se espera encontrar un patrón inverso para personas del Este Asiático, las cuales presentarían una alta asociación entre IMC y el consumo de carbohidratos y una alta respuesta a dietas bajas en este tipo de nutrientes.
La alta diferenciación de las poblaciones africanas podría ser consecuencia de patrones dietarios ancestrales, probablemente menos dependientes de la ingesta de carbohidratos, lo que es consistente con estudios enfocados en los patrones dietarios en estas poblaciones (11). Así, podría inferirse que la diferenciación observada respondería a los cambios dietarios experimentados durante la última transición alimentaria (12).
La mayor diferencia se observa al comparar macropoblaciones entre sí, puesto que existiría una alta homogeneidad dentro de cada una de ellas (a excepción de la macropoblación latinoamericana, donde la población de Lima, en Perú, destaca sobre el resto). Esta observación se traduce en que la predicción del éxito de dietas bajas en carbohidratos requeriría de la cuantificación de ancestría genética para estas cinco macropoblaciones ancestrales, siendo marginal el efecto de aquellas derivadas de poblaciones locales.
En Chile, es escasa la investigación referente al gen CLOCK, y los estudios que se han realizado en relación con el SNP rs3749474 T/C han contado con tamaños muestrales pequeños y solo han participado sujetos jóvenes. No obstante, los resultados confirman lo encontrado en investigaciones en otras regiones geográficas, asociando un mayor riesgo de obesidad en portadores del alelo T y aportando evidencias de interacción con el SNP rs4864548A (13,14). Dado lo anterior, posteriores investigaciones podrían incluir otras poblaciones latinoamericanas, a fin de contrastar la evidencia aquí reportada y contribuir así a la creación de un perfil de variabilidad a nivel subcontinental para diferentes loci de CLOCK asociados a obesidad y a la respuesta a dietas.
Si bien el presente estudio permite predecir la incidencia del alelo rs3749474 T dentro de las cinco macropoblaciones analizadas, una limitación de este estudio tiene relación con la existencia de particularidades poblacionales que no son aquí abarcadas. Por ejemplo, la existencia de poblaciones de diferentes ancestrías dentro de las macropoblaciones, como es el caso de poblaciones indígenas, que pueden presentar diferenciación genética respecto de sus respectivas macropoblaciones. Otra limitación tiene relación con el hecho de que la muestra con que se creó la base de datos 1000 Genomes corresponde a sujetos voluntarios, por lo que los resultados tienen un sesgo asociado al hecho de ser una muestra intencionada.
Finalmente, este trabajo presenta evidencia que contribuye a la generación de modelos probabilísticos para el éxito de dietas bajas en carbohidratos según las cinco macropoblaciones estudiadas. El tránsito hacia terapias médicas basadas en evidencia genómica personalizada, considerando la ancestría genética como uno de los factores causales, se hace cada vez más factible de incorporar a las decisiones biomédicas, como sería en el caso del gen CLOCK y su relación con ciclos circadianos, susceptibilidad a la obesidad y respuesta dietaria.