INTRODUCCIÓN
A nivel mundial, los indicadores de sobrepeso y obesidad están aumentando de manera alarmante. Ambas condiciones implican una acumulación de grasa en el tejido adiposo, lo que deriva en hipertrofia y desequilibrio de los procesos hemostáticos en que está involucrado (1). Estas condiciones de salud aumentan el riesgo de hipertensión, diabetes, eventos cardiovasculares, osteopatía e incluso cáncer (2-4).
En la actualidad, las estrategias dietéticas ya no solo se elaboran en función de la cantidad de calorías o macronutrientes consumidos sino también en la distribución circadiana a lo largo del día (ciclo circadiano) vinculada con la ingesta alimentaria (5-7). Esta distribución circadiana está regulada, entre otros, por el gen CLOCK (Circadian Locomotor Output Cycles Kapu), el cual influye en la regulación del peso, cuyo proceso es mediado por áreas del cerebro que estarían involucradas en el control de las funciones endocrinas relevantes a través de hormonas y neurotransmisores anoréxicos y orexígenos (8). Diferentes estudios sugieren que la diferenciación y la proliferación de adipocitos están reguladas por ritmos circadianos (9).
Se ha demostrado que ciertos polimorfismos del gen CLOCK están asociados con disfunciones metabólicas que aumentan el riesgo de obesidad y sobrepeso. Entre ellos está el polimorfismo rs48 64548 A/G, donde el alelo G es el de mayor frecuencia (10). No obstante, los portadores del alelo A son los que presentan mayor riesgo de obesidad y sobrepeso (11). En el caso del polimorfismo rs1554483 C/G, los portadores del alelo G, al estar en forma de haplotipo con los rs11932595A, rs6850524G y rs4864548A (GAGA), han presentado mayor riesgo de desarrollar hígado graso (11). Cuando los polimorfismos rs1554483 y rs4864548 se encuentran como haplotipo GA (rs1554483G y rs4864548A), el riesgo de desarrollar sobrepeso u obesidad aumenta 1,5 veces (12). Además, ha presentado asociación con la presión arterial diastólica y sistólica, la hipertensión arterial, los niveles de triglicéridos y otros componentes del síndrome metabólico (13).
En este estudio analizamos el patrón de variabilidad del haplotipo de riesgo GA de los polimorfismos rs1554483G y rs4864548A en 26 poblaciones y 5 macropoblaciones, con el objeto de obtener evidencia que contribuya al estudio y diseño de terapias en individuos portadores afectados de enfermedades relacionadas con el metabolismo de los lípidos y el ciclo circadiano.
METODOLOGÍA
OBTENCIÓN DE LOS DATOS
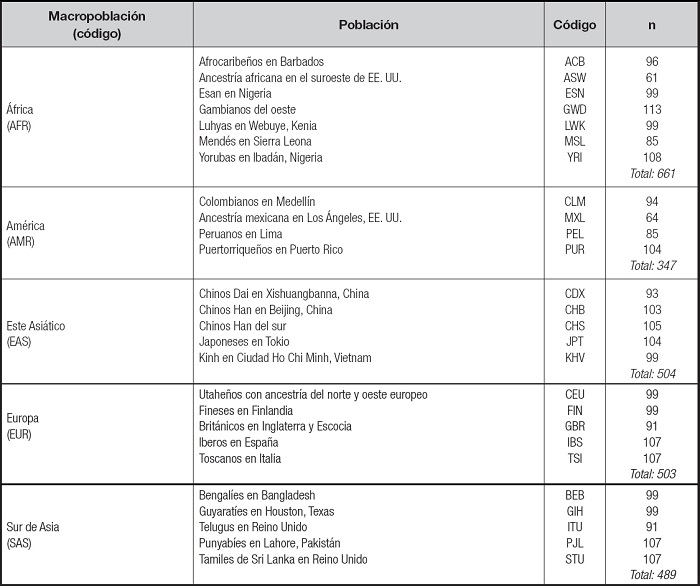
Se obtuvieron los genotipos de los SNP rs1554483 y rs4864548 de 2504 individuos a partir de la base de datos “1000genomes phase 3” (14). La muestra comprende las siguientes macropoblaciones: África, Este Asiático, Sur Asiático, Europa y Latinoamérica, abarcando 26 poblaciones (Tabla I).
La base de datos usada incluye la fase de los alelos de cada marcador, es decir, la información de cada SNP se encuentra segregada para ambos miembros de cada par cromosómico. Para extraer los 5008 haplotipos de la muestra, los genotipos de cada población se descargaron en formato VCF (variant call format) y luego se transformaron a formato FASTA a través de un script ad hoc escrito en R (15). Usando el mismo software, se obtuvo la frecuencia de cada haplotipo observado según cada población y macropoblación.
Análisis estadístico
Se compararon las frecuencias del haplotipo de riesgo GA de los polimorfismos rs1554483 y rs4864548 para cada par de macropoblaciones usando la prueba exacta de Fisher, utilizando el programa R con un intervalo de confianza del 95 % y un α = 0,05.
Las pruebas de equilibrio de Hardy-Weinberg y de desequilibrio de ligamiento se realizaron en el programa Vcftools (16). Mientras la primera permite poner a prueba la consistencia entre frecuencias alélicas y genotípicas (por tanto, la ausencia de factores evolutivos), la segunda permite estimar el grado de asociación entre loci. Ambas pruebas se aplicaron a nivel tanto poblacional como macropoblacional y para los dos marcadores considerados en este estudio.
RESULTADOS
Se observó una gran diferencia entre la frecuencia del haplotipo de riesgo GA de los polimorfismos rs1554483 y rs4864548 entre las 5 macropoblaciones de este estudio (Fig. 1). África es la que presenta la mayor divergencia, con la frecuencia más baja para el haplotipo de riesgo, seguida por Europa, Sur Asiático, Latinoamérica y Este Asiático. Esta observación fue ratificada por la prueba exacta de Fisher, en donde la única comparación no significativa (pFisher > 0,05) es entre el Sur Asiático y Latinoamérica (Tabla II).

Figura 1. Frecuencia del haplotipo de riesgo GA de los polimorfismos rs1554483 y rs4864548 en las 5 macropoblaciones de este estudio.
Tabla II. Comparación de las frecuencias del haplotipo de riesgo GA de los polimorfismos rs1554483 y 4864548 para cada par de macropoblaciones usando la prueba exacta de Fisher.
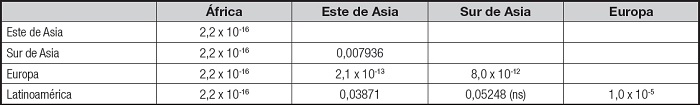
Los valores de las celdas corresponden a los valores p de la prueba de Fisher. ns: no significativo.
En relación con la comparación de la frecuencia del haplotipo de riesgo en las 26 poblaciones revisadas (Fig. 2), se observa una relativa homogeneidad, a excepción de Latinoamérica, en donde la población peruana de Lima y la puertorriqueña presentan altas frecuencias (58,8 % y 59,8 %, respectivamente) en relación con las poblaciones colombiana de Medellín y mexicana de Ciudad de México (38,3 % y 43,8 %, respectivamente) (pFisher < 0,05).

Figura 2. Frecuencia del haplotipo de riesgo GA de los polimorfismos rs1554483 y rs4864548 en las 26 poblaciones de este estudio.
Ambos SNP se encuentran fuera del equilibrio de Hardy-Weinberg cuando la muestra total no se subdivide poblacionalmente (pchi2 = 3,03 x 10-8 y 3,01 x 10-8 para el SNP rs1554483 y rs4864548, respectivamente). Al realizar este análisis en las 5 macropoblaciones, solo América Latina resultó fuera del equilibrio (pchi2 = 0,017 para ambos SNP). Finalmente, al realizar el análisis en las 26 poblaciones se encontró que solo la esan (Nigeria) está fuera del equilibrio de Hardy-Weinberg (pchi2 = 0,015 para ambos SNP).
Finalmente, ambos SNP resultaron altamente ligados a nivel mundial (R2 = 0,995). A nivel de las macropoblaciones, los valores también indican una alta asociación entre los alelos de estos dos marcadores (R2 = 1, 1, 0,983, 0,996 y 0,996 para África, Latinoamérica, Este Asiático, Europa y Sur Asiático, respectivamente). A nivel poblacional, las 26 poblaciones aquí estudiadas mostraron altos valores de desequilibrio de ligamiento (R2 entre 0,96 y 1).
DISCUSIÓN
Entre los aspectos más relevantes que muestran los resultados está el hecho de que África presenta una baja frecuencia del haplotipo de riesgos GA (rs1554483G y rs4864548A), presentando la mayor frecuencia la población del Este Asiático. La heterogeneidad observada entre macropoblaciones, y que fue ratificada por la prueba exacta de Fisher y de equilibrio de Hardy-Weinberg, sugiere la existencia de factores microevolutivos en donde, probablemente, los patrones dietarios, la migración y el mestizaje entre poblaciones ancestrales han afectado a la variación de los alelos de estos loci en el presente.
La alta divergencia encontrada entre las macropoblaciones no se observa al comparar las frecuencias poblacionales dentro de ellas. Dicho de otra forma, la variación entre macropoblaciones es evidentemente mayor que la variación dentro de dichas macropoblaciones. Lo anterior puede explicarse si los factores microevolutivos operaron sobre el haplotipo GA durante la formación de las 5 poblaciones acá estudiadas, pero de manera menos intensa durante tiempos posteriores, durante la formación de las 26 poblaciones aquí abarcadas.
Una excepción al patrón previamente descrito es la alta divergencia entre las poblaciones de Lima y Puerto Rico versus las de Ciudad de México y Medellín. Dada la alta diferenciación entre esos dos pares de poblaciones latinoamericanas, nuestra hipótesis es la existencia de factores microevolutivos, muy probablemente incluyendo la selección natural, y de relativamente alta intensidad, ocurridos durante la formación de las poblaciones de Lima y Puerto Rico. Cabe señalar que la frecuencia de este haplotipo en el trabajo de Sookoian y cols. (12), en una muestra obtenida entre la población de Buenos Aires, fue del 43,3 %. Esta frecuencia es intermedia entre las reportadas aquí para las poblaciones latinoamericanas y muy cercana a la encontrada en la población de Ciudad de México.
La única población en la que se refutó el equilibrio de Hardy-Weinberg fue la esan (Nigeria). Como para esta población se detectó un déficit de heterocigosidad para ambos SNP (pdeficit = 0,015), la causa más probable de la ausencia de equilibrio es la subdivisión poblacional (17) y no un fenómeno evolutivo relacionado con los loci aquí analizados.
En relación al alto desequilibrio de ligamiento encontrado en todas las macropoblaciones y poblaciones, es posible inferir que los haplotipos parentales (GA y CG) se encontrarán en alta frecuencia en relación a los haplotipos recombinantes (GG y AC) en cualquier otra población que se analice, a pesar de que la distancia física entre ellos es de 91986 bp (pares de bases nucleotídicas).
Los resultados obtenidos indican que, a nivel epidemiológico, el haplotipo GA tendría una relevancia heterogénea entre las diferentes macro poblaciones. Además, considerando que los resultados sugieren procesos evolutivos que han resultado en una alta diferenciación macropoblacional para este haplotipo, es plausible que otros loci asociados a la obesidad se distribuyan según el patrón macropoblacional aquí reportado. Posteriores estudios en donde se evalúe la asociación entre i) ancestría genética individual, segregada por las 5 macropoblaciones aquí analizadas, ii) susceptibilidad a la obesidad y iii) acumulación del haplotipo GA y otros alelos y haplotipos de riesgo, permitirían avanzar hacia la aplicación de herramientas genómicas personalizadas para evaluar tanto la susceptibilidad a la obesidad como la probabilidad de respuesta a las dietas bajas en carbohidratos.
Como línea de investigación posterior a este estudio, se propone analizar la asociación del haplotipo GA de los polimorfismos rs1554483 y rs4864548 en conjunto con los rs3749474, rs11932595 y rs6850524, ya que estos se han vinculado con el síndrome metabólico y sus componentes, como la hipertensión arterial, los niveles de triglicéridos y la respuesta a dietas bajas en carbohidratos (11,18).