Mi SciELO
Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO -
 Accesos
Accesos
Links relacionados
-
 Citado por Google
Citado por Google -
 Similares en
SciELO
Similares en
SciELO -
 Similares en Google
Similares en Google
Compartir
Avances en Odontoestomatología
versión On-line ISSN 2340-3152versión impresa ISSN 0213-1285
Av Odontoestomatol vol.29 no.3 Madrid may./jun. 2013
Agenesia dental: Epidemiología, clínica y genética en pacientes antioqueños1
Tooth agenesis: Epidemiological, clinical and genetic analysis in patients from Antioquia
Echeverri Escobar J.*, Restrepo Perdomo L.A.**, Vásquez Palacio G.***, Pineda Trujillo N.****, Isaza Guzmán D.M.*****, Manco Guzmán H.A.******, Marín Botero M.L.*******
* Odontóloga, especialista en Odontología Integral del Niño y Ortopedia Maxilar, Facultad de Odontología.
** Odontóloga, especialista en Odontología Integral del Adolescente y Ortodoncia, Facultad de Odontología.
*** Biólogo, Magíster en Ciencias Básicas Biomédicas, Especialista en Citogenética Humana. Profesor asistente Facultad de Medicina de la Unidad de Genética Médica.
**** Biólogo, Magíster en Ciencias Básicas Biomédicas, PhD en Genética. Profesor asociado. Facultad de Medicina y Coordinador de la Unidad de Mapeo Genético.
***** Bacterióloga, Magíster en Ciencias Básicas Biomédicas. Profesor Titular. Facultad de Odontología.
****** Odontólogo general, egresado Facultad de Odontología, Universidad de Antioquia. Medellín, Colombia. Director de la Empresa Social del Estado (ESE). Hospital San Juan de Dios del Municipio de ANORI (Antioquia, Colombia).
******* Bióloga (Universidad de Antioquia), Odontóloga especialista en Estomatología y Cirugía. Facultad de Odontología. Universidad de Antioquia. Medellín, Colombia.
1 Proyecto de investigación financiado por el CODI (Comité para el Desarrollo de la Investigación) Vicerrectoría de Investigación. Universidad de Antioquia. Medellín. Colombia.
Dirección para correspondencia
RESUMEN
Introducción: La odontogénesis es un proceso molecular complejo, susceptible a errores durante las etapas del desarrollo, que puede generar alteraciones, como agenesias dentales.
Metodología: Se realizó una descripción epidemiológica de la agenesia dental en 814 pacientes de la Facultad de Odontología de la Universidad de Antioquía entre 2006 y 2008, con base en la historia clínica y radiografías panorámicas. Se evaluó clínica y genéticamente una familia con agenesia dental no sindrómica, se determinó el tipo de segregación y patrón de herencia con el fin de identificar la implicación de los genes MSX1 y PAX9 en la agenesia. Se realizó la Reacción de Polimerización en Cadena (PCR), genotipificación y análisis de ligamiento. Se seleccionaron los marcadores D4S2285 y D4S432 (MSX1) y D14S288 y D14S70 (PAX9) por su alto índice de heterocigocidad.
Resultados: El análisis epidemiológico reveló mayor prevalencia de agenesias en la dentición permanente, en el sexo femenino, y los dientes más afectados fueron los tercero molares seguidos de los incisivos laterales superiores. Los resultados genéticos indicaron un posible ligamiento entre el gen MSX1 (LOD 0,97) con la agenesia dental y a su vez se identificó una posible asociación al azar del gen PAX9 (LOD -0,28) en la familia estudiada.
Palabras clave: Agenesia dental, hipodoncia, oligodoncia, PAX9, MSX1, ligamiento genético.
SUMMARY
Introduction: Odontogenesis is a complex molecular process, open to failures during different stages of development, that may lead to alterations like tooth agenesis.
Methods: An epidemiological description was made about tooth agenesis in 814 patients in the Faculty of Dentistry of the Antioquia University between 2006 and 2008, using clinical histories and panoramic X-rays, Furthermore, a family with non-syndromic tooth agenesis, was evaluated clinically and genetically to determine the segregation type and the inheritance pattern, and to identify the possible implication of the MSX1 and PAX9 genes. The analysis of those genes was valuated through Polymerase Chain Reaction (PCR), genotyping and linkage analysis. The markers D4S2285 and D4S432 for MSX1 and D14S288 and D14S70 for PAX9, were selected by their high heterozigocity index.
Results: The epidemiological analysis revealed a high prevalence of agenesis in permanent dentition and in the female gender, with predilection for third molars, followed by upper lateral incisors. The genetic findings showed a possible linkage between MSX1 gene (LOD 0.97) and the anomaly. At the same time a possible random association was found between the PAX9 gene (LOD -0.28) and the anomaly in the studied family.
Key words: Tooth agenesis, hypodontia, oligodontia, PAX9, MSX1, genetic linkage.
Introducción
La agenesia dental es una de las anomalías craneofaciales más comunes en el desarrollo humano (1,2). Se define como un desorden heterogéneo determinado genéticamente que se manifiesta como la ausencia congénita de uno o más dientes (3). Es considerada una condición de origen multifactorial influenciada por factores genéticos, ambientales, patológicos y evolutivos involucrados en los mecanismos normales de la odontogénesis. Es un proceso complejo de interacciones recíprocas y secuenciales entre células epiteliales y mesenquimáticas que dan origen a la formación dental (1,4-6), la agenesia se expresa como un rasgo aislado de forma esporádica o familiar, o como parte de más de 49 síndromes (7), entre ellos displasias ectodérmicas (8), Witkop "de dientes y uñas" (9), Rieger tipo I, Down, entre otros (10,11).
Presenta un patrón de herencia variable; frecuentemente se manifiesta como autosómico dominante (7,8,12-16), y en menor grado autosómico recesivo (17) o ligado al cromosoma X (8), penetrancia incompleta entre el 86% (18,19), y el 97% (20) con expresividad variable. La cantidad, tipo, ubicación, severidad y simetría de los dientes afectados se observa con gran diversidad en un individuo y en los miembros de una misma familia (16,20,21).
Existen asociaciones entre la presencia de agenesia dental y otras características dentales anómalas, incluyendo: alteraciones en la formación y erupción de los dientes permanentes (21), microdoncias (22), incisivos laterales en clavija (23), malposiciones de caninos (24), erupción ectópica de primeros molares permanentes, infraerupción de molares deciduos (25), enanismo radicular, invaginación en incisivos (26), taurodontismo (27) y rotación de incisivos laterales y premolares superiores (28,29).
La embriogénesis dental involucra más de 200 genes (12) que codifican factores de crecimiento, factores de transcripción, moléculas de señalización y proteínas encargadas de regular las actividades celulares y determinar la posición, número y forma de los dientes (29). Entre los genes que participan en el desarrollo dental, se encuentran los de la familia Homeobox (MSX1, MSX2 y PAX9) (11) y otros relacionados con alteraciones de tipo sindrómico como AXIN2 (31-33) y el PITX2 (30). De ellos, los más comprometidos como factor causal de la agenesia dental son MSX1 y PAX9 (33-35).
El gen PAX9 está situado en el cromosoma 14, locus (14q12-q13), pertenece a la familia de genes PAX que codifican factores de transcripción (12). Se expresa ampliamente en el mesénquima derivado de la cresta neural y su principal función es establecer la capacidad inductiva de éste. Desde el punto de vista genético, las mutaciones en este gen detienen la formación dental en el estadio de brote el cual es necesario para la expresión de los genes BMP4, MSX1 y LEF1 (30). Hasta la fecha se han identificado aproximadamente 26 mutaciones en el gen PAX9 asociadas con agenesia dental familiar no sindrómica (30, 36,37), siendo los dientes más afectados los molares y en algunos casos los premolares (35,38,39).
El gen MSX1 se encuentra ubicado en el cromosoma 4 locus (4p16.2), se caracteriza por presentar un homeodominio. Codifica factores de transcripción que participan en las distintas etapas del desarrollo y funciona como represor de la transcripción. Se han identificado aproximadamente 10 mutaciones en el gen MSX1 (30,36,40) que inducen ausencia principalmente de segundos premolares y terceros molares y en raras ocasiones de incisivos laterales superiores (7,11,16,35).
En Colombia, hay pocos estudios que indiquen la prevalencia, tipo de herencia y los genes implicados en la agenesia dental, así como su asociación con otras anomalías y síndromes (5,41).
En este estudio se describe el perfil epidemiológico de agenesia dental de los pacientes activos entre el 2006-2008 de los postgrados de Odontología Integral del Niño y Ortopedia Maxilar y Odontología Integral del adolescente y Ortodoncia en la Facultad de Odontología de la Universidad de Antioquia. También se describen los aspectos clínicos y genéticos de una familia seleccionada por presentar un número significativo de individuos afectados con agenesia dental.
Materiales y métodos
A. PERFIL EPIDEMIOLÓGICO
Se evaluaron los pacientes activos en los postgrados de Odontología Integral del Adolescente y Ortodoncia y Odontología Integral del Niño y Ortopedia Maxilar de la Facultad de Odontología de la Universidad de Antioquia, entre el año 2006 y 2008, con el fin de obtener un perfil epidemiológico de dicha anomalía en esta población.
Se seleccionaron 814 historias clínicas que tuvieran radiografías panorámicas y sin historia de exodoncia o pérdida dental por traumas u otra causa. Se incluyeron las ausencias congénitas en dentición decidua o permanente, incluso la de terceros molares para la cual se tuvo en cuenta la edad promedio de formación entre los 8 y los 12 años (41-42).
Análisis estadístico
Se realizó un análisis descriptivo utilizando distribuciones de prevalencia para las variables cualitativas (edad, sexo, tipo y ubicación de diente afectado y anomalías asociadas). En las variables cuantitativas se calcularon medidas de tendencia central y de dispersión. Mediante un análisis univariado y bivariado, se identificaron los posibles factores asociados de las variables utilizando las pruebas de χ2 (chi cuadrado de independencia) con un valor de significancia p<0,05.
B. ESTUDIO CLÍNICO Y GENÉTICO FAMILIAR
Se seleccionó un paciente probando con agenesia dental y su grupo familiar de 31 individuos, dentro del cual se encontraron 6 individuos afectados (3 mujeres y 3 hombres), distribuidos en 3 generaciones.
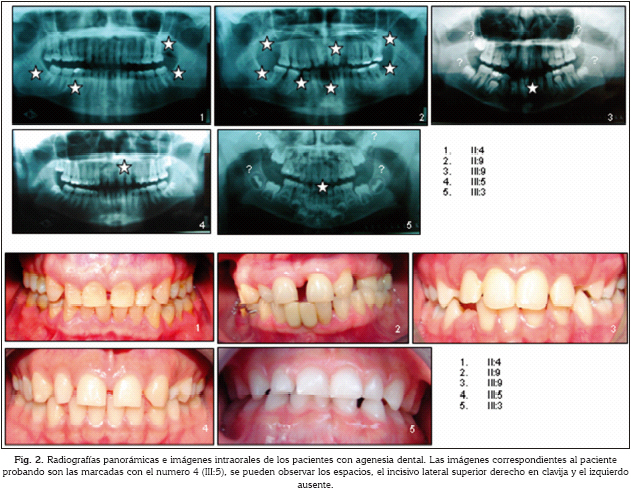
La genealogía fue elaborada empleando el programa Cyrilic para Windows y se determinó el patrón de herencia. Se realizó el estudio clínico correspondiente y el análisis genético. Se elaboraron historias clínicas de todos los pacientes, se obtuvieron radiografías panorámicas y fotografías clínicas intraorales y extraorales. Cada paciente o su acudiente firmaron voluntariamente el consentimiento informado con base en la declaración de Helsinki de la Asociación Médica Mundial de 1964 y las normas científico-técnicas y administrativas para la investigación en salud, resolución 008430 de 1993 del Ministerio de Salud, título II capítulo I.
Para el análisis molecular se tomaron 10 ml de sangre periférica en EDTA o muestras de saliva a cinco de los seis afectados y a 10 individuos sanos.
Extracción de ADN
Para las muestras de sangre periférica como de saliva se obtuvo el ADN genómico mediante el kit Wizard® Genomic DNA purification (PROMEGA - Madison WI, USA) siguiendo las instrucciones del fabricante para volúmenes de 300 μl de sangre o saliva total.
Selección de los marcadores para los genes PAX9 Y MSX1
Para los genes MSX1 y PAX9 se seleccionaron los marcadores por su alto índice de heterocigocidad, los cuales se encontraban flanqueando los genes de interés a partir de las secuencias publicadas en el Genbank® (www.ncbi.nlm.nih.gov). Para el gen PAX9 se seleccionaron los marcadores D14S288 y D14S70 y para MSX1, los marcadores D4S432 y D4S2285. Ver tabla 1.
Reacción de polimerización en cadena (PCR)
La PCR se realizó en un volumen de 50 μl que contenía 1 μM de cada uno de los cebadores, los cuales estaban marcados con HEXTM (Hexaclorofluoresceína) y 6-FAMTM (6-carboxifluoresceína) en los extremos 5´ de los oligonucleótidos forward (sentido) (IDT® - Coralville, IA, USA) 0,2 μM de cada uno de los desoxinucleótidos trifosfatos (dTTP, dATP, dCTP, dGTP - PROMEGA), 2 mM de MgCl2 (PROMEGA), 1,25 U de Taq polimerasa (PROMEGA), 10 μl de buffer de PCR 5X (PROMEGA) y 3 μl de ADN.
La PCR se realizó en un termociclador Mastercycler® Gradient (Eppendorf, Hamburgo, Alemania) el cual se programó para cada uno de los marcadores evaluados. Las condiciones de temperatura fueron las siguientes: un ciclo de desnaturalización inicial a 95o C por 10 minutos seguido por 30-35 ciclos de 95o C por 30 segundos, 56o-60o C por 1 minuto y 72o C por 1 minuto con un ciclo de extensión final a 72o C por 2 minutos. Los productos de PCR se sometieron a electroforesis en gel de agarosa al 1,6% (Invitrogen - Carlsbad, CA, USA) en buffer TAE pH 8,2-8,4 (Tris, ácido acético, EDTA - PROMEGA) adicionado con 5 μl de bromuro de etidio 10 mg/ml (PROMEGA) usando un marcador de peso molecular de ADN de 100 a 1.500 pb (PROMEGA). Los minigeles se corrieron a 100 V durante 40 minutos (Fisher Scientific - Dubuque IA, USA) y se visualizaron en un transiluminador ultravioleta (ChemiDoc XRS System - BIORAD Hércules, CA, USA).
Genotipificación
Para la identificación de los genotipos se hizo una dilución 1:10 de los amplicones marcados con HEXTM (D4S2285 y D14S70) y una dilución 1:15 de los marcados con 6-FAMTM (D4S432 y D14S288) en agua ultrapura. Posteriormente, se preparó una mezcla con 12 μl de formamida desionizada (AMRESCO - Solon, OH, USA) y 0,5 μl de TAMRA® (Applied Biosystems - GeneScanTM Foster City, CA, USA) a la cual se le adicionó 2 μl de la dilución que contenía todos los marcadores de cada individuo. Estas muestras así preparadas, se desnaturalizaron en un bloque de calor a 94o C por 5 minutos e inmediatamente se dejaron en hielo para evitar la renaturalización de las cadenas de ADN. Posteriormente se llevaron al analizador genético ABI PRISM® 310 (Applied Biosystems) y los corridos se analizaron con los programas GENESCAN y GENOTYPER (Applied Biosystems).
Análisis de ligamiento
El análisis de ligamiento se utilizó para determinar si los genes PAX9 y MSX1 segregaron independientemente o no de la agenesia.
El método estadístico de LOD fue empleado para evaluar la existencia de ligamiento entre el fenotipo de la enfermedad y los marcadores seleccionados (D4S2285, D4S432, D14S70 y D14S288) para los genes MSX1 y PAX9 respectivamente. Los valores LOD en las diferentes fracciones de recombinación se calcularon con el programa MLINK del paquete LINKAGE versión 5.1 (43) y asumiendo un patrón de herencia autosómico dominante con penetrancia del 67% calculada a partir de la genealogía en estudio.
Análisis de haplotipos
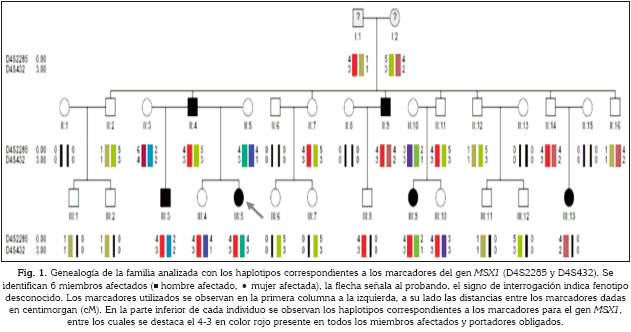
Para la identificación de los haplotipos (o cromosomas) segregando con la anomalía se utilizó el método de Cadenas de Montecarlo implementado en el Programa Simwalk 2 (44). Los haplotipos fueron dibujados con ayuda del Programa HaploPainter (45) (Figura 1).
Resultados
Estudio epidemiológico
Se estudiaron 814 pacientes (378 hombres y 436 mujeres) de los cuales 221 fueron positivos para agenesia dental excluyendo los terceros molares (12,3%), la edad promedio fue de 15,8 años (rango entre 2 y 61 años). En cuanto al género las mujeres afectadas correspondieron al 14,0% mientras que los hombres al 13,14%. Estos datos sugieren que hay una mayor proporción de mujeres afectadas en la población estudiada. Sin embargo, estos resultados no fueron estadísticamente significativos (p= 0,4895) para relacionar el género con la manifestación de la anomalía. Solo hubo un caso de dentición decidua afectada (0,9%) y los dientes ausentes en orden de frecuencia hallada, fueron: terceros molares (51,5%), incisivos laterales superiores (10,6%), segundos premolares inferiores (8,3%), segundos premolares superiores (6,6%), incisivos centrales inferiores (5,5%), e incisivos laterales inferiores (4,4%). Predomino la presentación bilateral y en los casos unilaterales de incisivos laterales superiores y segundos premolares inferiores, el lado más afectado fue el derecho y a nivel de terceros molares, el izquierdo.
La asociación entre la agenesia de terceros molares y otros tipos de dientes correspondió al 11,3% de los pacientes con el fenotipo. Excluyendo los terceros molares, el 49% de los individuos afectados presentaron agenesia de un diente, el 40% entre 2 y 5 dientes, el 10% presentó oligodoncia y solamente en el 1% anodoncia.
Análisis clínico familiar
El análisis genealógico determinó un patrón de herencia autosómico dominante, con penetrancia incompleta del 67% y expresividad variable, y una distribución similar entre ambos sexos
En cuanto a los abuelos o miembros de la primera generación, no fue posible diagnosticar ni radiográfica ni clínicamente el compromiso dental, ya que estos eran edéntulos totales. Una mujer de la segunda generación y su núcleo familiar correspondiente (su hija afectada), no pudieron ser incluidos en el estudio ya que decidieron no participar en él; sin embargo, fueron considerados en el análisis de la genealogía (ver figura 1. Individuos: II:11 y III:6).
A nivel familiar fue posible evidenciar agenesia de dientes tanto deciduos como permanentes, disminuciones en el tamaño dental (dientes en clavija), ausencia de terceros molares, malposiciones dentarias, espaciamientos, retraso en la erupción dental y rotaciones de los premolares superiores. Estos hallazgos son consistentes con la mayoría de estudios de hipodoncia familiar no sindrómica (5,15,20,29). Ver figura 2.
Análisis genético y molecular
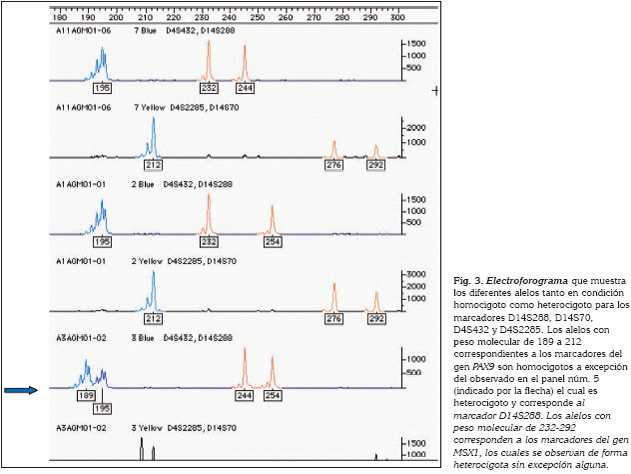
La tasa de genotipificación fue del 100%. Así, de todos aquellos individuos de quienes se pudo tomar una muestra, se obtuvieron sus cuatro genotipos. En la figura 3 se muestran el electroforograma con los diferentes alelos para los marcadores D14S288, D14S70, D4S432 y D4S2285 y, en la figura 1, los genotipos y haplotipos para dos de los microsatélites analizados. En esta figura contrastan, en cuanto a los genotipos, los individuos II:11 y III:6 quienes no consintieron la toma de la muestra. De este modo la tasa de genotipificación fue 90%.
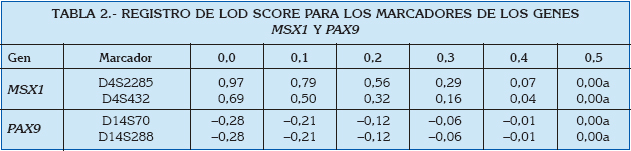
Los valores de LOD máximos obtenidos en las fracciones de recombinación en (θ)= 0,00 fueron 0,97 para el locus D4S2285 y 0,69 para el D4S432, sugiriendo ligamiento del gen MSX1 y la anomalía. Los valores de LOD encontrados para los marcadores seleccionados para evaluar el gen PAX9 (D14S70 y D14S288) fueron negativos (-0,28), por lo que se observa una posible asociación al azar de este gen con la agenesia. Adicionalmente, estos valores se correlacionaron con la forma de segregación del haplotipo en todos los individuos afectados y portadores obligados por lo que es posible suponer que el gen MSX1 es portador de la mutación que causa la agenesia en la familia estudiada (tabla 2).
En el análisis de haplotipos, fue posible identificar un caso en condición heterocigota conformado por los alelos 4-3 para los marcadores D4S2285-D4S432; este haplotipo estuvo presente en todos los individuos afectados y en los portadores obligados (figura 1). Ambos marcadores están flanqueando el gen MSX1, lo que permite suponer la implicación de este gen en la etiología de la agenesia. La segregación de los mismos, permitió identificar al abuelo (primera generación I:1) como portador del haplotipo asociado con el riesgo para la condición. El abuelo fundador (I:1), se consideró como portador de la variante de susceptibilidad en la familia.
Discusión
En la familia estudiada se encontraron variaciones en el número, tamaño, forma ubicación y simetría de los dientes afectados. El hecho de encontrar alteraciones en la forma y desarrollo de otros dientes, sugiere variaciones comunes en la expresión de los genes involucrados en la señalización dentaria que pueden influenciar la respuesta del fenotipo (11,16).
La frecuencia de agenesia excluyendo los terceros molares informada en la literatura varia (rango 1.6% a 9.6%). En este trabajo se encontró un 12,2% de agenesias, similares a las encontradas en Irlanda 11,3% (1,20,38,46).
En la población general y teniendo en cuenta el tipo de diente, los más comúnmente ausentes son los terceros molares, seguidos de los incisivos laterales maxilares o segundos premolares mandibulares e incluso los incisivos centrales mandibulares, con diferencias estadísticas entre uno y otro estudio (1,10, 20,47). Según la teoría de los campos, se correlaciona la alta vulnerabilidad y variabilidad de estos dientes con su posición anatómica en el extremo distal de la lámina dental o en las áreas donde se fusionan los procesos faciales (48).
En este trabajo encontramos que la agenesia de terceros molares se presentó con mayor frecuencia en comparación con los otros grupos de dientes, con una prevalencia del 51,5%, la cual supera los reportes previos en Colombia (49) del 21% y México (50), del 33%, respectivamente.
Lundstrom (51) y Vastardis (1) reportaron que es más frecuente la manifestación unilateral mientras que Bailit (52), Quintero (5) y Pinho (53) indicaron que se presenta de forma simétrica y bilateral, al igual que lo encontrado en el presente estudio. Igualmente, un alto porcentaje de individuos presentó agenesia de un diente, seguido de dos a cinco dientes y finalmente el porcentaje más bajo correspondió a la anodoncia; datos similares fueron encontrados en Venezuela (54) y en la mayoría de estudios en caucásicos (15).
A nivel molecular, está ampliamente documentado el estricto control genético que subyace la odontogénesis, determinando las posiciones, el número y la forma de los diferentes dientes (8). Los diversos reportes de casos y las líneas de evidencia molecular, sugieren que entre los genes asociados con la formación dental, los genes PAX9 Y MSX1 codifican factores de trascripción esenciales para activar el potencial odontogénico entre el epitelio y el mesénquima (11).
La presencia de posibles mutaciones o polimorfismos en dichos genes, genera un interés especial para la comprensión de la patogénesis de la agenesia dental. Las mutaciones en el gen MSX1 se asocian con hipodoncia de premolares y en menor frecuencia de terceros molares e incisivos y las del gen PAX9, se asocian principalmente con la ausencia de molares y rara vez premolares (11,16).
Nieminen (29) excluyó el ligamiento de los genes MSX1 y MSX2 con hipodoncia en 20 familias finlandesas. Así mismo, Scarel (55), descartó el ligamiento y la presencia de polimorfismos y mutaciones en el gen MSX1, concluyendo que otros genes pueden estar involucrados en la etiología de la hipodoncia en humanos.
Lidral (15) usó el análisis de ligamiento paramétrico, con el fin de determinar la relación existente entre la presencia de la condición a nivel familiar y la posible mutación del gen mencionado (MSX1). En dicho estudio se encontró un valor de LOD de 1.68 y tras una secuenciación del gen, se identificó una mutación por sustitución (Met61Lys), que afecta la función normal del MSX1 por diversos mecanismos. Chishti (17) obtuvo un valor máximo de LOD de 2,85 en el marcador D4S2285 del gen MSX1, al estudiar una familia con oligodoncia con patrón hereditario autosómico recesivo y finalmente encontró una mutación sin sentido en el homeodominio de este gen.
Stockton (7) encontró un valor LOD de 6,83 para el marcador D14S288 del gen PAX9 indicando ligamiento de éste con la anomalía. Posteriormente identificó una mutación de marco de lectura en el dominio par del PAX9 en una familia con hipodoncia autosómica dominante en la que los principales dientes afectados fueron molares permanentes. De la misma manera, Frazier (56) y Das (57) obtuvieron valores LOD significativos indicando ligamiento entre la oligodoncia y el gen PAX9.
En Colombia, Berrocal (58) realizó estudios en 7 grupos familiares (35 individuos) con el fin de determinar ligamiento para el gen PAX9 con los marcadores D14S288 y D14S70, sin encontrar asociación entre éste y la presencia de la anomalía.
En conclusión, las bases genéticas y moleculares de la agenesia dental no sindrómica permanecen inciertas (59) debido a que son muchos los mecanismos moleculares que predisponen a las errores en el proceso del desarrollo normal de la estructura dental. Por otra parte, se debe considerar la influencia de la heterogeneidad genética en las expresiones fenotípicas de las poblaciones estudiadas.
Se recomienda realizar la secuenciación del gen MSX1 con el fin de identificar las posibles mutaciones e incluir más familias con agenesia que permitan comparar y fortalecer los resultados clínicos y genéticos encontrados.
Agradecimientos
A Mariano Ospina Pérez, biólogo, por su valiosa colaboración en la organización de los datos y en el enfoque de la unidad de análisis.
Al CODI (Comité para el Desarrollo de la Investigación) de la Universidad de Antioquia por la financiación que prestó a este proyecto de investigación.
Bibliografía
1. Vastardis H. The genetics of human tooth agenesis: new discoveries for understanding dental anomalies. Am J Orthod Dentofacial Orthop 2000 Jun;117(6):650-6. [ Links ]
2. Feng HL, Zhang XX, Wu H. Research advances in tooth agenesis. Beijing Da Xue Xue Bao 2007 Feb 18;39(1):13-7. [ Links ]
3. Peres RC, Scarel-Caminaga RM, do Espirito Santo AR, Line SR. Association between PAX-9 promoter polymorphisms and hypodontia in humans. Arch Oral Biol 2005 Oct;50(10):861-71. [ Links ]
4. Nasman M, Forsberg CM, Dahllof G. Long-term dental development in children after treatment for malignant disease. Eur J Orthod 1997 Apr;19(2):151-9. [ Links ]
5. Quintero ME, Restrepo MA, Ordoñez A, Riaño C, Berrocal MA. Tooth agenesis and its heredity pattern in seven Colombian familiar groups. Univ Odontol 2002;22:27-36. [ Links ]
6. Schalk-van der WY, Steen WH, Bosman F. Distribution of missing teeth and tooth morphology in patients with oligodontia. ASDC J Dent Child 1992 Mar;59(2):133-40. [ Links ]
7. Stockton DW, Das P, Goldenberg M, D´Souza RN, Patel PI. Mutation of PAX9 is associated with oligodontia. Nat Genet 2000 Jan;24(1):18-9. [ Links ]
8. Arte S, Nieminen P, Pirinen S, Thesleff I, Peltonen L. Gene defect in hypodontia: exclusion of EGF, EGFR, and FGF-3 as candidate genes. J Dent Res 1996 Jun;75(6):1346-52. [ Links ]
9. Jumlongras D, Bei M, Stimson JM, Wang WF, DePalma SR, Seidman CE, et al. A nonsense mutation in MSX1 causes Witkop syndrome. Am J Hum Genet 2001 Jul;69(1):67-74. [ Links ]
10. Frazier-Bowers SA, Pham KY, Le EV, Cavender AC, Kapadia H, King TM, et al. A unique form of hypodontia seen in Vietnamese patients: clinical and molecular analysis. J Med Genet 2003 Jun;40 (6):e79. [ Links ]
11. Ogawa T, Kapadia H, Feng JQ, Raghow R, Peters H, D´Souza RN. Functional consequences of interactions between Pax9 and Msx1 genes in normal and abnormal tooth development. J Biol Chem 2006 Jul 7;281(27):18363-9. [ Links ]
12. Klein ML, Nieminen P, Lammi L, Niebuhr E, Kreiborg S. Novel mutation of the initiation codon of PAX9 causes oligodontia. J Dent Res 2005 Jan;84(1):43-7. [ Links ]
13. Burzynski NJ, Escobar VH. Classification and genetics of numeric anomalies of dentition. Birth Defects Orig Artic Ser 1983;19(1):95-106. [ Links ]
14. Graber LW. Congenital absence of teeth: a review with emphasis on inheritance patterns. J Am Dent Assoc 1978 Feb;96(2):266-75. [ Links ]
15. Lidral AC, Reising BC. The role of MSX1 in human tooth agenesis. J Dent Res 2002 Apr;81(4):274-8. [ Links ]
16. Vastardis H, Karimbux N, Guthua SW, Seidman JG, Seidman CE. A human MSX1 homeodomain missense mutation causes selective tooth agenesis. Nat Genet 1996 Aug;13(4):417-21. [ Links ]
17. Chishti MS, Muhammad D, Haider M, Ahmad W. A novel missense mutation in MSX1 underlies autosomal recessive oligodontia with associated dental anomalies in Pakistani families. J Hum Genet 2006;51(10):872-8. [ Links ]
18. Burzynski NJ, Escobar VH. Classification and genetics of numeric anomalies of dentition. Birth Defects Orig Artic Ser 1983;19(1):95-106. [ Links ]
19. Grahnen H. Hypodontia in the permanent dentition. A clinical and gentical investigation. Odont Revy 1956;7(Suppl 3):1-100. [ Links ]
20. Arte S, Nieminen P, Apajalahti S, Haavikko K, Thesleff I, Pirinen S. Characteristics of incisor-premolar hypodontia in families. J Dent Res 2001 May;80(5):1445-50. [ Links ]
21. Uslenghi S, Liversidge HM, Wong FS. A radiographic study of tooth development in hypodontia. Arch Oral Biol 2006 Feb;51(2):129-33. [ Links ]
22. Garn SM, Lewis AB. The gradient and the pattern of crown-size reduction in simple hypodontia. Angle Orthod 1970 Jan;40(1):51-8. [ Links ]
23. Alvesalo L, Portin P. The inheritance pattern of missing, peg-shaped, and strongly mesio-distally reduced upper lateral incisors. Acta Odontol Scand 1969 Dec;27(6):563-75. [ Links ]
24. Camilleri S. Maxillary canine anomalies and tooth agenesis. Eur J Orthod 2005 Oct;27(5):450-6. [ Links ]
25. Bjerklin K, Kurol J, Valentin J. Ectopic eruption of maxillary first permanent molars and association with other tooth and developmental disturbances. Eur J Orthod 1992 Oct;14(5):369-75. [ Links ]
26. Apajalahti S, Arte S, Pirinen S. Short root anomaly in families and its association with other dental anomalies. Eur J Oral Sci 1999 Apr;107(2):97-101. [ Links ]
27. Seow WK, Lai PY. Association of taurodontism with hypodontia: a controlled study. Pediatr Dent 1989 Sep;11(3):214-9. [ Links ]
28. Baccetti T. Tooth rotation associated with aplasia of nonadjacent teeth. Angle Orthod 1998 Oct;68 (5):471-4. [ Links ]
29. Nieminen P, Arte S, Tanner D, Paulin L, Alaluusua S, Thesleff I, et al. Identification of a nonsense mutation in the PAX9 gene in molar oligodontia. Eur J Hum Genet 2001 Oct;9(10):743-6. [ Links ]
30. Kolenc-Fuse FJ. Tooth agenesis: in search of mutations behind failed dental development. Med Oral Patol Oral Cir Bucal 2004 Nov;9(5):385-395 [ Links ]
31. Lammi L, Arte S, Somer M, Jarvinen H, Lahermo P, Thesleff I, et al. Mutations in AXIN2 cause familial tooth agenesis and predispose to colorectal cancer. Am J Hum Genet 2004 May;74 (5):1043-50. [ Links ]
32. Lammi L, Halonen K, Pirinen S, Thesleff I, Arte S, Nieminen P. A missense mutation in PAX9 in a family with distinct phenotype of oligodontia. Eur J Hum Genet 2003 Nov;11(11):866-71. [ Links ]
33. Mostowska A, Biedziak B, Jagodzinski PP. Axis inhibition protein 2 (AXIN2) polymorphisms may be a risk factor for selective tooth agenesis. J Hum Genet 2006;51(3):262-6. [ Links ]
34. Peters H, Balling R. Teeth. Where and how to make them. Trends Genet 1999 Feb;15(2):59-65. [ Links ]
35. Vieira AR, Meira R, Modesto A, Murray JC. MSX1, PAX9, and TGFA contribute to tooth agenesis in humans. J Dent Res 2004 Sep;83(9):723-7. [ Links ]
36. Suda N, Ogawa T, Kojima T, Saito C, Moriyama K. Non-syndromic oligodontia with a novel mutation of PAX9. J Dent Res. 2011 Mar;90(3): 382-6. [ Links ]
37. Paixão-Côrtes VR, Braga T, Salzano FM, Mundstock CA, Bartolini MC. PAX9 and MSX1 transcription factor genes in non-syndromic dental agenesis Archives of Oral Biology 2011;56 (4):337-44. [ Links ]
38. Gerits A, Nieminen P, De MS, Carels C. Exclusion of coding region mutations in MSX1, PAX9 and AXIN2 in eight patients with severe oligodontia phenotype. Orthod Craniofac Res 2006 Aug;9(3): 129-36. [ Links ]
39. Ogawa T, Kapadia H, Wang B, D´Souza RN. Studies on Pax9-Msx1 protein interactions. Arch Oral Biol 2005 Feb;50(2):141-5. [ Links ]
40. Wang Y, Kong H, Mues G, D´Souza RN. Msx1 Mutations: How Do They Cause Tooth Agenesis? J Dent Res 2011;90(3):311-316. [ Links ]
41. Moorrees CF, Fanning EA, Hunt EE Jr. Age variation of formation stages for ten permanent teeth. J Dent Res 1963 Nov;42:1490-502 [ Links ]
42. Nolla CM. The development of the permanent teeth. J Dent Child 1960;4(4):254-66. [ Links ]
43. Lathrop GM, Lalouel JM, Julier C, Ott J. Strategies for multilocus linkage analysis in humans. Proc Natl Acad Sci USA 1984 Jun;81(11):3443-6. [ Links ]
44. Sobel E, Lange K. Descent graphs in pedigree analysis: applications to haplotyping, location scores, and marker-sharing statistics. Am J Hum Genet 1996 Jun;58(6):1323-37. [ Links ]
45. Thiele H, Nurnberg P. HaploPainter: a tool for drawing pedigrees with complex haplotypes. Bioinformatics 2005 Apr 15;21(8):1730-2. [ Links ]
46. O´Dowling IB, McNamara TG. Congenital absence of permanent teeth among Irish school-children. J Ir Dent Assoc 1990;36(4):136-8. [ Links ]
47. Xuan K, Jin F, Liu YL, Yuan LT, Wen LY, Yang FS et al. Identification of a novel missense mutation of MSX1 gene in Chinese family, with autosomal-dominant oligodontia. Arch Oral Biol 2008; 53:773-9. [ Links ]
48. Nieminen P. Molecular genetics of tooth agenesis. Finland: University of Helsinki; 2007. [ Links ]
49. Sarmiento P, Herrera A. Agenesia de terceros molares en estudiantes de Odontologia de la Universidad del Valle entre 16 y 25 años. Colomb Méd 2004;35(supl1):5-9. [ Links ]
50. Nuno González MM, Llarena del Rosario ME. Radiographic study of formation and calcification of the third molar. Pract Odontol 1990 Nov;11 (11):27-1. [ Links ]
51. Lundstrom A. Asymmetries in the number and size of the teeth and their aetiological significance. Trans Eur Orthod Soc 1960; 36:167-85. [ Links ]
52. Bailit HL. Dental variation among populations. An anthropologic view. Dent Clin North Am 1975 Jan;19(1):125-39. [ Links ]
53. Pinho T, Tavares P, Maciel P, Pollmann C. Developmental absence of maxillary lateral incisors in the Portuguese population. Eur J Orthod 2005 Oct;27(5):443-9. [ Links ]
54. Pier-Domenico B, Jiménez H. Prevalencia e interpretación radiográfica de la agenesia dentaria en el área de influencia del servicio de Ortopedia Dentofacial de la Facultad de Odontología de la Universidad de Carabobo. Odous Cientifica 2006 Enero-Junio Vol. VII (1):37-45. [ Links ]
55. Scarel RM, Trevilatto PC, Di HO, Jr., Camargo LE, Line SR. Absence of mutations in the homeodomain of the MSX1 gene in patients with hypodontia. Am J Med Genet 2000 Jun 19;92(5): 346-9. [ Links ]
56. Frazier-Bowers SA, Guo DC, Cavender A, Xue L, Evans B, King T, et al. A novel mutation in human PAX9 causes molar oligodontia. J Dent Res 2002; 81:129-33. [ Links ]
57. Das P, Hai M, Elcock C, Leal SM, Brown DT, Brook AH, et al. Novel missense mutations and a 288-bp exonic insertion in PAX9 in families with autosomal dominant hypodontia. Am J Med Genet A 2003;118A(1):35-42. [ Links ]
58. Berrocal M, Ordóñez A, Gutiérrez S, Durán C, Gómez M, et al. Agenesia dental: Exclusión de los genes MSX1 y PAX9. Rev Latin de Orthod 2003; 2:5-10. [ Links ]
59. Ichikawa E, Watanabe A, Nakano Y, Akita S, Hirano A. Kinoshita A, et al. PAX9 and TGFB3 are linked to susceptibility to nonsyndromic cleft lip with or without cleft palate in the Japanese: population-based and family-based candidate gene analyses. J Hum Genet 2006;51 (1):38-46. [ Links ]
![]() Dirección para correspondencia:
Dirección para correspondencia:
Martha Lucía Marín Botero
Bióloga. Profesora titular de medio tiempo
Facultad de Odontología.
Universidad de Antioquia.
Calle 64, No 52-59
Medellín, Colombia
E-mail: marthaluciamarin@gmail.com
Fecha de recepción: 31 de agosto de 2011.
Aceptado para publicación: 18 de septiembre de 2011.