Mi SciELO
Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO -
 Accesos
Accesos
Links relacionados
 Citado por Google
Citado por Google -
 Similares en
SciELO
Similares en
SciELO  Similares en Google
Similares en Google
Compartir
Oncología (Barcelona)
versión impresa ISSN 0378-4835
Oncología (Barc.) vol.27 no.2 feb. 2004
ORIGINALES
Técnicas empleadas para el estudio de la interacción entre agentes antitumorales y el DNA
M. A. García; B. de Pascual-Teresa
Departamento de Ciencias Químicas. Facultad de Ciencias Experimentales y de la Salud. Universidad San Pablo CEU. Madrid
Dirección para correspondencia
RESUMEN
Los procesos oncológicos se caracterizan por un aumento incontrolado de la proliferación celular y en ellos está implicada la molécula de DNA. El DNA es la diana biológica con la que interaccionan muchos de los fármacos antitumorales y diversos compuestos potencialmente antineoplásicos. Tras muchos años de trabajo, los investigadores han sido capaces de diseñar y sintetizar numerosos compuestos que se unen al DNA por diferentes mecanismos y entre ellos se encuentran muchos de los agentes antitumorales conocidos. Diversas técnicas nos permiten hoy en día establecer los tipos de interacciones que tienen lugar entre los ligandos y el DNA, algunas de ellas son ya clásicas y otras, sin embargo, son de reciente desarrollo. Una combinación racional de métodos puede ayudar a determinar el mecanismo molecular de la unión, a establecer relaciones estructura-actividad y, lo que es más importante, a diseñar de forma racional nuevos agentes.
Palabras clave: DNA. Ligandos. Antitumorales. Interacciones ligando-DNA.
SUMMARY
Oncological processes are characterized by an uncontrolled cell replication always associated to DNA implication. DNA molecule is the biological target for the action of many antitumor drugs and potential antineoplastic compounds. As a result of many years of work, researchers have been able to design and synthesize a great number of compounds binding to DNA through different mechanisms, among which many known antitumor agents are found. Several techniques allow us nowadays to establish the type of interactions between DNA and its ligands, some classical and some recently developed. A reasoned combination of methods can help to differentiate the molecular mechanisms of the binding processes, to establish the structure-activity relationships, and, what is more important, to develop reasoned designs for the discovery of new agents.
Key words: DNA. Ligands. Antitumor agents. Ligand-DNA interactions.
Introducción
Existen moléculas pequeñas capaces de interaccionar con la molécula de DNA y lo hacen a través de diversos mecanismos. El conocimiento de cómo lo hacen y la especificidad que presentan, constituye uno de los objetivos principales para el posterior diseño de agentes más afines y más selectivos. Los esfuerzos dedicados a este fin, desde las representaciones de Scatchard en la década de los setenta hasta las modernas herramientas informáticas aplicadas en la actualidad, han permitido de alguna manera medir estas interacciones entre los diferentes compuestos, a los que en este trabajo nos referimos como ligandos, independientemente de su modo de unión y la molécula de DNA.
Una clasificación muy básica consiste en distinguir interacciones reversibles e interacciones irreversibles. Se entiende como interacción reversible aquella en la que el ligando se une al DNA en unas condiciones determinadas, que si cambian, pueden hacer que el ligando deje de unirse. Estas interacciones son conocidas también como interacciones no enlazantes y son propias de los fármacos que poseen como diana la molécula de DNA.
Dentro de las interacciones reversibles se pueden encontrar varios subtipos como son:
· Agentes intercalantes: ligandos que se disponen apilados entre los pares de bases, sus interacciones con las bases nitrogenadas son de tipo electrostático o de van der Waals y en ocasiones mediante la formación de enlaces de hidrógeno1.
· Agentes que se unen a un surco: la disposición en doble hélice de las cadenas de DNA hace que se formen unas hendiduras a lo largo del exterior de la hélice donde residen los grupos fosfato de los nucleótidos que son conocidas según su amplitud. Así, se habla de surco mayor y de surco menor. Estos surcos, debido a su diferente estructura, muestran preferencia por determinados ligandos2.
· Rompedores de hebra: Este tipo de agentes interaccionan con el DNA de forma que, una parte de su estructura es capaz de unirse reversiblemente al DNA, mientras que otra forma radicales libres que rompen la doble hélice imposibilitando su replicación3, 4.
Las interacciones irreversibles, son también conocidas como interacciones enlazantes. En ellas se forma un enlace que une ambas moléculas el cual no se puede deshacer. Este tipo de interacciones son propias de los gases de guerra y algunos antitumorales como el Cisplatino5.
Los distintos tipos de interacción ligando-DNA se pueden caracterizar mediante diversas técnicas experimentales y teóricas que se describen a continuación.
Técnicas experimentales
Los ensayos de unión (binding) permiten monitorizar la interacción del ligando con la molécula de DNA para así determinar su constante de asociación. Estos ensayos aprovechan generalmente cambios en las propiedades espectroscópicas o físicas del ligando o del DNA.
Centrifugación en gradiente de densidad:6 Al unirse un ligando a la molécula de DNA, ésta aumenta su masa y su densidad, por lo que se puede diferenciar su velocidad de sedimentación respecto a la misma molécula de DNA sin ligando unido, mediante una centrifugación en gradiente de densidad en disolución acuosa. Mediante el empleo de la ecuación de Svedberg, se mide la velocidad de sedimentación o coeficiente de sedimentación de los complejos DNA-ligando frente a DNA aislado como control:

donde s es el coeficiente de sedimentación, D es el coeficiente de difusión, M es la masa molecular y el término k incluye el volumen empleado, la densidad del agua y la constante de los gases.
La centrifugación en gradiente de densidad, constituye un método rápido y eficaz para determinar la existencia de interacción entre un ligando y la molécula de DNA. Sin embargo, no permite determinar el tipo de unión.
Viscosimetría:7 Si a la molécula de DNA se le une un ligando, el conjunto aumenta su viscosidad con respecto al DNA libre. Esta diferencia de viscosidad es la que hace que esta técnica permita determinar si un compuesto se une o no al DNA, ya que detecta cambios estructurales y variaciones en el tamaño de la molécula. Dado que, en la unión por intercalación el principal efecto es un alargamiento del DNA por desenrollamiento de la doble hélice, permite también diferenciar entre la unión por intercalación y la unión a surco, en la que no se produce un alargamiento de la cadena tan pronunciado.
Los cambios que se producen en la viscosidad de una disolución que contiene DNA al añadir un ligando, se detectan mediante la utilización de un viscosímetro y midiendo el cambio en el tiempo de elución de dicha disolución. Así, manteniendo la concentración de DNA constante, podemos obtener la viscosidad relativa h de la siguiente ecuación:

donde t el tiempo de elución de la disolución ligando-DNA y t0 es el tiempo de elución de DNA libre.
Para el cálculo de la longitud aparente del fragmento de DNA (L/L0) se necesita conocer la fracción molar ligando/par de bases de DNA (r) y los coeficientes de fricción (f(p), f(p)o) según va variando r8, 9.
De la representación de L/Lo frente a r se obtiene la pendiente m que indica si el ligando intercala o no, y en caso positivo, si se trata de un monointercalante o bisintercalante10-13.
La viscosimetría es por tanto una técnica sencilla y rápida para detectar interacciones ligando-DNA, que además proporciona información relativa al modo de unión.
Espectrofotometría UV/Visible:14 Un compuesto, al unirse al DNA, modifica su espectro de absorción, ya que sufre modificaciones en su estructura electrónica. En el espectro de absorción se producen desplazamientos en los máximos con respecto al ligando libre. Éste puede ser por desplazamiento a longitudes de onda mayores, efecto batocrómico, junto con una disminución de los coeficientes de extinción molar, efecto hipocrómico. Estos cambios espectrales permiten conocer aspectos como la estequiometría del complejo ligando-DNA y el tamaño del sitio de unión.
En un equilibrio químico en el que sólo una especie absorbe, aplicando la ley de Lambert-Beer, se puede determinar la constante de equilibrio midiendo la absorbancia como una función de la concentración, eso sí, la relación entre la absorbancia o fluorescencia y la concentración debe ser lineal en los rangos de concentración utilizados en el ensayo.
Se pueden conocer el número de sitios de unión realizando estudios de equilibrio de unión y utilizando métodos de tritiación. Esto consiste en que una disolución del ligando de concentración conocida es transferida a una cubeta con temperatura controlada y el cambio progresivo de absorbancia y fluorescencia se recoge después de la adición de una serie de alícuotas de DNA. La adición de la disolución de DNA a la disolución del ligando da lugar a un cambio en la absorbancia y en la fluorescencia que depende del comportamiento óptico del compuesto analizado.
La medida de la absorbancia (A) a cualquier longitud de onda, es un sumatorio entre la absorbancia debida a la forma libre y a la forma unida, como se puede ver en la siguiente ecuación:

donde C es la concentración de ligando conocida (Clibre+ Cunido), y e es el coeficiente de extinción molar de cada especie. El análisis de la unión requiere la determinación de Cl y por tanto, la cantidad r de ligando unido por unidad de DNA. La determinación de Cu requiere una extrapolación a altas concentraciones de DNA añadido, para asegurarnos que toda el ligando esté unido, pero con la precaución de que el exceso de DNA no cause un incremento de absorbancia más allá del punto de saturación.
Se han desarrollado muchos métodos para determinar los valores de r y Cl y el tratamiento empleado depende del modelo de unión usado. Por ejemplo, para un modelo de unión sencillo (dos estados), los datos pueden ser ajustados a una ecuación de Scatchard15 donde se representa r/Cl frente a r:

donde Ki es la constante de equilibrio intrínseca y n representa el número de sitios de unión en el DNA (normalmente nucleótidos o par de bases) ocupados por el ligando unido. Una relación lineal es difícil de obtener para los ácidos nucleicos por lo que es más frecuente utilizar el modelo de McGhee, Von Hippel16 y Crothers17 para tener en cuenta la ocupación en múltiples sitios y el principio de exclusión18, 19.
Una forma más compleja de la ecuación de McGhee-Von Hippel introduce el factor de cooperatividad w , que mide la interacción de las moléculas ya unidas.
La elección de un modelo u otro depende del sistema ligando-DNA. Lo que se suele hacer es un primer intento para ver si se ajusta con el modelo de Scatchard que es el más sencillo, si los resultados no son los óptimos, se utiliza el modelo de McGhee-Von Hippel o su extensión teniendo en cuenta la cooperatividad (w).
Uno de los métodos empleados para la dilucidación de la estequiometría de la unión es el método de variación continua sugerido por Job20 en 1980 y desarrollado posteriormente por Likussar y Boltz21, así como por Cantor y Schimmel22. Este método consiste en realizar medidas ópticas de disoluciones donde las concentraciones del DNA y del ligando, se van variando, pero la suma de las concentraciones de ambos permanece constante. El cambio de absorbancia o fluorescencia se representa frente a la fracción molar del ligando y se observa una inflexión que nos va a proporcionar información relativa a la estequiometría del proceso. En la mayoría de los casos, los resultados se ajustan al modelo de McGhee-von Hippel11, 12.
Por tanto, la utilización de espectrometría UV/Visible permite conocer aspectos como la estequiometría del complejo ligando-DNA y el tamaño del sitio de unión. La extensión de estas técnicas permite además definir la especificidad por una determinada secuencia y la constante de asociación. Sin embargo, no dan información sobre el tipo de unión.
El método de tritiación reversa de sales permite hacer una estimación de la carga del ligando. Este método se basa en el principio de que las interacciones entre ácidos nucleicos y ligandos cargados son sensibles a los efectos electrostáticos producidos por los cationes, así, el valor de Ki generalmente desciende a medida que aumenta la concentración de sal (NaCl) como resultado de la liberación estequiométrica de contraiones por la unión del ligando cargado, de forma que:

donde Z es la carga positiva formal en el ligando y y es la proporción de contraiones asociados con cada fosfato del DNA, normalmente 0,88 para un B-DNA. Así, el valor de la medida de Ki a diferentes concentraciones de NaCl nos proporciona información directa sobre la carga Z del ligando, o el estado de protonación al pH utilizado para los experimentos. El análisis por mínimos cuadrados de los datos resultantes del análisis nos da el valor de -Zy y una estimación de la carga de la molécula.
Dicroísmo circular:23 De los métodos disponibles para el análisis de interacciones ligando-DNA en disolución, el dicroísmo circular es uno de los más sencillos, no requiere mucho material y proporciona información que no puede ser obtenida por otros métodos. El principio en el que se basa el dicroísmo circular es el siguiente: se utilizan pulsos de campo eléctrico para alinear las moléculas de DNA en la dirección del vector del campo eléctrico, éste alineamiento molecular se analiza mediante la medida de la absorbancia de la luz polarizada. Cuando se unen moléculas al DNA, si esta unión provoca cambios en la conformación del DNA, su espectro sufrirá variaciones en mayor o menor medida.
La dirección y la magnitud del cambio de absorbancia inducido por los pulsos de campo indican la orientación del cromóforo con respecto al eje principal del DNA; las constantes de tiempo del proceso de rotación molecular proporcionan información sobre la dimensión hidrodinámica del complejo. Esta técnica permite también conocer los parámetros eléctricos del complejo.
Si se realiza un experimento a una longitud de onda para la detección en la cual sólo absorbe el ligando, no el DNA y tras incubar ambos juntos, no se produce absorbancia, se puede concluir que el ligando se une al DNA y si se repite el experimento a distintas concentraciones se puede utilizar para determinar la constante de unión. La ausencia de absorbancia se debe a que el ligando unido al DNA se debe orientar preferentemente en dirección perpendicular al eje del DNA.
El dicroismo circular se utiliza preferentemente en la caracterización de agentes intercalantes aunque requiere bien un cromóforo asimétrico o un cromóforo simétrico en un ambiente asimétrico como es la molécula de DNA.
Electroforesis capilar:24 Cuando un ligando se une al DNA se altera su movilidad, pudiendo ser detectada la interacción. Utilizando un conjunto de fragmentos de DNA de tamaño homogéneo, se pueden ver diferencias en la migración entre el DNA libre y el DNA con ligando unido. Por este motivo, se puede utilizar esta técnica para el análisis de la interacción DNA-compuesto intercalante ya que además es una técnica de alto poder de resolución en la que se utilizan unas cantidades muy pequeñas de muestra. Aunque esta técnica es capaz de detectar interacciones de ligandos con el DNA, no permite identificar el tipo de interacción que se produce.
Los métodos calorimétricos permiten analizar el perfil termodinámico de la interacción y su dependencia respecto a la secuencia de unión.
En principio, se puede calcular mediante este tipo de técnicas tanto la DG (afinidad) como el cambio de entalpía DH del proceso. No obstante, hay muchos sitios en el DNA para la interacción y asignar valores de DH para cada uno depende del modelo matemático utilizado en el análisis.
Lo que se puede obtener en la mayoría de los casos es la DH de transferencia del ligando en un entorno acuoso hacia un sitio de unión de alta afinidad en un DNA desocupado. Mediante el empleo de calorímetros más sofisticados se pueden hacer análisis a concentraciones utilizadas en estudios de UV-Visible. Las medidas calorimétricas en combinación con el análisis de la correspondiente isoterma de unión proporcionan tanto los cambios de entalpía como los de entropía (DG = DH - TDS) de la unión del ligando al DNA.
La principal limitación que presentan estas técnicas para la medida de la afinidad resulta cuando el ligando se agrega a la concentración empleada en la medida, el calor observado es la suma de al menos estos dos términos:

En muchos casos DHunión es exotérmica (negativa) y DHdeagg, es decir, el cambio de entalpía para la producción de agregado a partir de monómeros, normalmente es endotérmica. Los problemas asociados a éste fenómeno se pueden evitar realizando las medidas a concentraciones a las cuales la fracción del ligando en el estado de monómero sea cercana a uno. Un análisis completo del calor observado cuando ocurre la agregación, requiere una constante de equilibrio para el proceso de agregación.
La técnica calorimétrica más utilizada es la técnica de desnaturalización térmica:25 el DNA de doble hélice se desnaturaliza a elevadas temperaturas, esto significa que sus dos hebras se separan por rotura de los enlaces de hidrógeno que las unen. A la temperatura que ocurre el fenómeno de desnaturalización se le llama temperatura de fusión (Tm). El proceso de determinación de la Tm se realiza subiendo la temperatura gradualmente desde 20° C hasta 80° C, midiendo la absorbancia de forma permanente a 260 nm. El valor de la absorbancia registrado va aumentando con la temperatura hasta alcanzar un máximo, que corresponde con el estado en que las dos hebras se han separado completamente. Cuando se une un compuesto al DNA, esta unión puede estabilizar las dos hebras frente al aumento de la temperatura, elevando así la Tm de dicho segmento, o por el contrario favorecer la ruptura de los enlaces de hidrógeno, disminuyendo su Tm (Fig. 1).
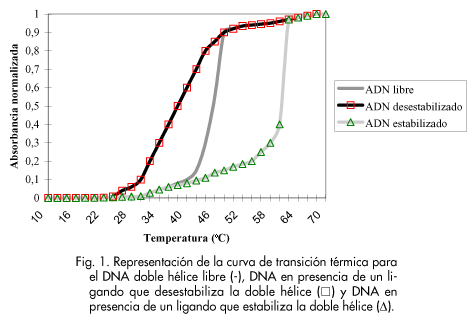
Para determinar si un compuesto se une al DNA se incuba con diferentes concentraciones del mismo y se hacen las medidas del DNA sólo y en presencia del compuesto. Si se ha formado un complejo entre el DNA y el compuesto, se producirá un desplazamiento del máximo de la curva de desnaturalización del DNA.
La adición de ligandos a las disoluciones de ácidos nucleicos también puede cambiar la forma de las curvas de transición. Para polímeros de ácidos nucleicos homogéneos, la curva de transición es afilada en presencia o ausencia de ligando debido a la homogeneidad de su composición. En sistemas más heterogéneos, la adición de los ligandos causa un incremento en el rango de la transición y una transición bifásica en algunos casos. Este cambio en las transiciones se explica por la mayor fuerza de enlace de un ligando a determinadas secuencias o estructuras que por lo tanto quedan más estabilizadas que otras regiones.
Los agentes que se unen al DNA mediante un proceso de intercalación, se unen con mayor preferencia a los ácidos nucleicos de doble hélice que a los de hélice sencilla, porque los primeros tienen características estructurales uniformes con energías libres favorables para la formación de los complejos. Las bases apiladas proporcionan un ambiente adecuado para la intercalación de un ligando, lo cual estabiliza la doble hélice e incrementa la Tm. Los intercalantes clásicos como el bromuro de etidio, aumentan la Tm tanto del DNA como del ARN de doble hélice. Generalmente los intercalantes no tienen una especificidad de secuencia marcada por lo que los valores de DTm para secuencias ricas en GC y secuencias ricas en AT, son prácticamente los mismos.
Los agentes que se unen al surco mayor o menor reconocen la geometría específica de estas zonas cargadas negativamente debido a los grupos fosfato a lo largo de toda la hélice. Los ligandos clásicos que se unen al surco menor como distamicina, pentamidina, netropsina y compuestos relacionados, están cargados positivamente y se unen en el surco a zonas ricas en adenina y timina, por lo tanto, estabilizan esas secuencias con el consecuente aumento de la Tm.
Sin embargo, los DNA de hélice sencilla, no tienen surco específico. La distamicina por ejemplo, que se une a surco de DNA doble hélice, no puede unirse con una alta afinidad al DNA de hélice sencilla.
Existen ligandos que se unen de forma dual al DNA, es decir, se unen fuertemente al surco menor en secuencias poliA-T, proporcionando valores muy altos de DTm, a la vez que se unen por intercalación a secuencias G-C, registrándose menores valores de DTm que en secuencias ricas en A-T. Como ejemplos de este tipo de agentes se encuentran el berenilo, furamidina y DAPI.
La constante de asociación de interacción entre un ligando y un ácido nucleico de doble hélice se puede calcular a partir de los resultados de Tm, utilizando la siguiente ecuación:

donde Tm° y Tm son las temperaturas de fusión o melting en ausencia y presencia de ligando, respectivamente. DH° es el incremento de entalpía asociado a la fusión de la doble hélice cuando está sin ligando, Kh y Kss son las constantes de unión del ligando a la doble hélice y a la cadena sencilla; Nh y Nss son el número de sitios de unión; R es la constante de los gases. La actividad a está relacionada con la cantidad de ligando libre a la temperatura Tm. Hay que destacar que las constantes de equilibrio de unión están calculadas para la temperatura Tm y la extrapolación de esos valores a otras temperaturas como pueden ser 25° ó 37° C requiere el conocimiento de la entalpía de formación del complejo y el uso de la ecuación de van't Hoff.
La ecuación descrita tiene cinco incógnitas: Nh, Kh, Nss, Kss y a. En muchos casos, el producto Kss·a es muy inferior a 1 y el segundo término de la ecuación desaparece y se reduce a:

Nh se suele determinar con un experimento de tritiación en condiciones que favorezcan la unión del ligando a la doble hélice, como por ejemplo una concentración pequeña de sal. En estas condiciones, Tm se representa frente al porcentaje de ácido nucleico unido al ligando. Cuando los sitios de unión del ligando empiezan a saturarse, el efecto de seguir aumentando la relación ligando unido a ácido nucleico hace que disminuya la Tm. El cambio en los valores de Tm hace que se llegue a una meseta y Nh es la relación ligando-ácido nucleico que corresponde al punto de cambio. Para el cálculo de Kh, si la temperatura de fusión se determina en condiciones de saturación pero sin exceso de ligando, entonces, para los ligandos con altos valores de Kh (Kh > 106), la actividad a puede ser aproximada a la mitad del total de la concentración de ligando. Con Nh y DHo determinados independientemente, la ecuación anterior puede usarse para calcular Kh. Debe recordarse que Kh es la constante de asociación a la temperatura Tm y no se han considerado uniones a las especies de cadena sencilla. Aunque es posible determinar Kh y Nh en una representación de Tm frente a concentración de ligando mediante el empleo de una rutina de ajuste a una curva, la determinación independiente de Nh generalmente nos proporciona unos resultados más ajustados.
Las técnicas de desnaturalización térmica proporcionan información detallada sobre la entalpía del proceso, la constante de asociación y el número de sitios de unión. El procedimiento experimental resulta relativamente sencillo, siendo más complejo el tratamiento de los datos.
La utilización de las técnicas de footprinting26-28 supuso un salto cuantitativo en el estudio de las interacciones ligando-DNA.
Mediante estas técnicas se comprueba si el ligando se une específicamente a una secuencia de bases determinada. Esta técnica es simple, rápida y razonablemente económica. Se basa en la capacidad de algunas enzimas como la desoxirribonucleasa I (DNAsa I) o reactivos sintéticos, ej. metidio-propil-EDTA (MDE) de hidrolizar el DNA con baja especificidad de secuencia. El ligando protege sus sitios de unión de la hidrólisis por DNAsa I o MDE, permitiendo así la identificación de las secuencias a las que se une. Se utiliza para ello un fragmento de DNA de 100-200 pb de secuencia conocida marcado en uno de los extremos de la cadena, se produce la digestión del DNA donde queda protegida la zona a la que está unido el ligando. Tras este proceso, se separa el producto de la reacción en un gel de poliacrilamida y se revela; la posición del ligando puede verse como un hueco vació en la secuencia de nucleótidos (Fig. 2).
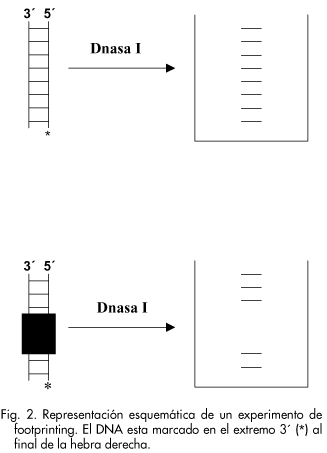
La técnica de footprinting utilizando DNAsa I ha sido utilizada satisfactoriamente para identificar o confirmar los sitios de unión de diversos ligandos como pueden ser la actinomicina28-30, mitramicina31, antibióticos quinoxalínicos26,32, daunomicina33,34, nogalamicina35 y agentes que se unen al surco menor28, 29, 36.
La puesta a punto de las técnicas de RMN y de espectroscopía de masas para adecuarlos al estudio de macromoléculas les ha valido a los doctores John Fenn, Koichi Tanaka y al profesor Kurt Wütrich el premio Nobel de química del año 2002. Se puede obtener información más detallada del proceso de formación de complejos DNA-ligando por técnicas como la difracción de rayos X y métodos espectroscópicos como la RMN 1H y RMN 13C.
Espectrometría de masas: La espectrometría de masas es una técnica que data de los comienzos del siglo XX. Esta técnica se desarrolló con vistas a determinar el peso molecular de los compuestos químicos. En la actualidad es una técnica de gran exactitud, sensibilidad y rapidez que permite llevar a cabo determinaciones en mezclas químicas complejas, sin embargo la espectroscopía de masas no resultó aplicable por muchos años a las macromoléculas biológicas por no ser de fácil aplicación a estas los procesos tradicionales de vaporización. El Prof. Fenn contribuyó al desarrollo de la espectrometría de masas para macromoléculas modificando adecuadamente la técnica de electrospray para volatilizar sin inducir alteraciones en las macromoléculas. La espectrometría de masas de ionización por electrospray (ESI-MS) se está aplicando en la actualidad para la caracterización de biomoléculas, en particular para proteínas37, 38. Posteriormente se ha revelado como una técnica de considerable interés para la detección de asociaciones no covalentes entre biomoléculas39. ESI-MS es una técnica a tener en cuenta en la caracterización estructural de los complejos ligando-DNA, que ofrece ventajas potenciales como sensibilidad y rapidez en el análisis en comparación con otros métodos estructurales como RMN o difracción de rayos X.
Los métodos de RMN nos proporcionan información sobre el grado y el tipo de interacción entre el ligando y el DNA, mediante la comparación de los espectros de resonancia de los oligonucleótidos libres, los ligandos libres y los complejos formados por cada oligonucleótido y el ligando.
Aunque los métodos cristalográficos son los que aportan una mayor cantidad de estructuras resueltas,40 la resonancia magnética nuclear permite una determinación estructural en disolución. (En la actualidad, entre el 15 y el 20% de las estructuras tridimensionales conocidas han sido determinadas por resonancia magnética nuclear). La RMN se basa en el hecho de que ciertos núcleos atómicos cuando se orientan mediante un campo magnético absorben radiación a frecuencias características. Este hecho es aprovechado en el caso del 1H que proporciona diferentes líneas en el espectro dependiendo de su entorno, como resultado es posible observar señales para átomos individuales en macromoléculas de 30 kDa de masa (unos 20 pb para ácidos nucleicos). Así podemos conocer la estructura molecular, la conformación y la posición dinámica de los ácidos nucleicos. El primer paso para cualquier estudio de RMN es la asignación de las señales a cada átomo y este suele ser el paso limitante de toda la operación. La sustitución selectiva de protones por deuterio puede acelerar este proceso, ya que evitaríamos solapamiento de señales. Es muy importante el estudio de las conectividades espaciales mediante estudios NOESY (Nuclear Overhauser Spectroscopy) y ROESY (Rotating-frame Overhauser Spectroscopy) ya que la mayor parte de la información sobre la estructura en disolución viene del análisis de las distancias internucleares NOESY.
La RMN se puede aplicar al estudio de dobles y triples hélices tanto de DNA como de RNA, complejos entre ligandos y DNA y entre proteínas y DNA.
Los métodos de difracción de rayos X permiten determinar la estructura tridimensional de macromoléculas biológicas con resolución atómica. La cristalografía de DNA sigue un tratamiento similar a la cristalografía de proteínas41, donde se distinguen los siguientes pasos:
· Formación del cristal: Para ello se llega a una disolución sobresaturada42 donde se empiezan a formar agregados que actúan como núcleos de cristalización. El DNA se condensa en presencia de poliaminas, cationes inorgánicos trivalentes, alcoholes y cationes divalentes.43 Para encontrar las condiciones favorables para la formación del cristal se han de tener en cuenta una multitud de parámetros44: tipos de iones metálicos y poliamidas empleadas, tipo y concentración de los ligandos de DNA, alcohol y tampón utilizado, pH, temperatura y agentes precipitantes.
· Obtención de datos por difracción de rayos X: El objetivo final de un experimento de difracción de rayos X es determinar la densidad electrónica,r (x, y, z) para cada átomo de la macromolécula de acuerdo con la siguiente ecuación:

· donde V es el volumen de la celda unidad, |F(hkl)| es el factor estructural de amplitud, hkl es el índice de Miller, xyz las coordenadas espaciales reales y ahkl la fase relativa de la reflexión hkl. En un experimento de difracción de rayos X se mide la intensidad [I (hkl)] de muchos miles de reflexiones, utilizando una cámara CCD o una pantalla detectora. Las intensidades se convierten en factores de amplitud estructural [|F(hkl)|] mediante I(hkl) = |F(hkl)|2.
· Uno de los objetivos de todos los experimentos de difracción es obtener los datos a la mayor resolución posible, para ello se debe utilizar la menor intensidad posible, lo cual requiere un tiempo de recogida de datos mayor, unos detectores sensibles, etc. En la actualidad es imposible obtener pocos datos a alta resolución simultáneamente con reflexiones debidas al apilamiento. Una de las posibles estrategias es obtener los datos de intensidad para el apilamiento independientemente de los datos obtenidos a gran resolución del mismo cristal. Las intensidades de apilamiento se obtienen con exposiciones cortas y bajo amperaje sobre un ánodo rotatorio.
· Determinación de la fase mediante reemplazamiento molecular, reemplazamiento de múltiples isomorfos (MIR) o difracción anómala de múltiples longitudes de onda (MAD). La utilización de la técnica de desplazamiento molecular implica la determinación de la orientación del eje de la hélice mediante un análisis de Patterson combinado con información sobre la simetría, para simplificar la resolución de la estructura. La determinación mediante MIR y MAD requiere la sustitución con átomos pesados. Los derivados pueden ser generados mediante la formación de un nuevo cristal de DNA modificado. Las bases Citosina y Uracilo pueden ser sustituidas con bromo o yodo en la posición C545-47.
· Refinamiento.
La difracción de rayos X es la técnica empleada en aproximadamente el 85% de las estructuras resueltas y depositadas en el Protein Data Bank48. Es una técnica que en algunos casos es capaz de elucidar estructuras con una resolución menor a 2 Å siendo la mayoría de ellas inferior a 3 Å. Sin embargo, la formación de cristales constituye el paso limitante para la utilización de esta técnica.
Métodos teóricos
La aplicación de los métodos teóricos al estudio de la interacción de estos compuestos con el DNA, se conoce como modelado molecular, que si bien ha alcanzado en la actualidad su máxima expresión y utilidad con la aplicación de sistemas informáticos49-51, comenzaron en los años cincuenta con James Watson y Francis Crick que postularon un modelo de estructura para el DNA52. Los resultados que podemos obtener con estos métodos van desde energías de interacción ligando-DNA, modo de interacción, cambios conformacionales sufridos por el ligando o el DNA, formación o destrucción de enlaces de hidrógeno, etc.
El conjunto de estas técnicas permite conocer diversas variables de la interacción entre los ligandos y el DNA, como puede ser el modo de interacción (intercalación, unión a surco...), la especificidad de secuencia, la estequiometría del complejo, la afinidad de unión, etc.53-57
Entre los métodos teóricos utilizados hoy en día para el estudio de interacciones fármaco-receptor se incluyen las minimizaciones de energía (ME), las simulaciones de dinámica molecular (DM), las simulaciones de Monte Carlo (MC), y las perturbaciones de la energía libre (PEL)58. Todas estas técnicas ofrecen en última instancia la posibilidad de comprender la interacción entre los ácidos nucleicos y moléculas orgánicas en términos de fuerzas moleculares. Para ello dependen de la fiabilidad de la representación analítica de la hipersuperficie energética de las moléculas, que viene dada por la suma de una serie de funciones simples que expresan tanto la energía requerida para distorsionar las coordenadas internas más allá de unos valores de equilibrio, como las interacciones entre átomos no enlazados (Fig. 3).

El modelado molecular continúa su evolución aplicando una gran variedad de métodos computacionales al problema de identificar las complejas relaciones existentes entre estructuras moleculares y actividades biológicas en términos de interacciones entre átomos. Aunque el modelado molecular se utiliza en muchas ocasiones como método predictivo, el fin último de estas técnicas es utilizar los conocimientos adquiridos tanto de forma experimental como teórica para diseñar compuestos con un determinado perfil de actividad.
Técnicas de implantación futura
La microscopía de escaneado atómico promete revolucionar el estudio de las interacciones ligando-receptor en general y de ligandos-DNA en particular, en un corto espacio de tiempo. A pesar de que todavía esta en fase de desarrollo, ha producido imágenes excelentes de DNA sencillo, tanto libre como unido a un ligando. Esta técnica presenta la ventaja de poder trabajar con muestras en vacío, en disolución, en el mismo aire ambiental, a temperaturas altas o criogénicas59.
Conclusiones
La determinación de la estructura en doble hélice del DNA se considera uno de los hitos científicos del siglo XX. Cincuenta años después, la doble hélice ha cobrado un protagonismo inesperado, entre otras muchas razones por ser la diana farmacológica de muchos fármacos antitumorales y por lo tanto constituye un candidato ideal para el diseño racional de fármacos.
Resulta por lo tanto imprescindible disponer de técnicas que permitan evaluar la interacción entre los ligandos y la doble hélice de DNA. En el presente artículo de revisión, se resumen las técnicas más habituales utilizadas para determinar tanto el tipo de interacción como la selectividad y las constates de afinidad.
Bibliografía
1. Braña MF, Cacho M, Gradillas A, de Pascual-Teresa B, Ramos A. Intercalators as Anticancer Drugs. Curr. Pharm. Des. 2001; 7:1745-1780. [ Links ]
2. Wemmer DE, Dervan PB. Targeting the minor groove of DNA. Curr. Opin. Struct. Biol. 1997; 7:355-61. [ Links ]
3. Hecht SM. The chemistry of activated bleomycin. Acc. Chem. Res. 1986; 383-391. [ Links ]
4. Hecht SM. DNA strand scission by activated bleomycin group antibiotics. Fed. Proc. 1986; 45:2784-91. [ Links ]
5. Cohen SM, Lippard SJ. Cisplatin: from DNA damage to cancer chemotherapy. Prog. Nucleic. Acid. Res. Mol. Biol. 2001; 67:93-130. [ Links ]
6. Darnell J, Lodish H, Beck A, Zipursky L, Matsudaria P, Baltimore D. Biología celular y molecular. Barcelona: Omega, 1993. [ Links ]
7. Suh D, Chaires JB. Criteria for the mode of binding of DNA binding agents. Bioorg. Med. Chem. 1995; 3:723-728. [ Links ]
8. Cory M, Mckee DD, Kagan J, Henry DW, Miller JA. Design, Synthesis, and DNA Binding Properties of Bifunctional Intercalators. Comparison of Polymethylene and Diphenyl Ether Chains Connecting Phenanthridine. J. Am. Chem. Soc. 1985; 107:2528-2536. [ Links ]
9. Graves DE, Watkins CL, Yielding LW. Ethidium bromide and its photoreactive analogues: spectroscopic analysis of deoxyribonucleic acid binding properties. Biochemistry 1981; 31:1887-92. [ Links ]
10. Cohen G, Eisenberg H. Viscosity and Sedimentation Study of Sonicated DNA-Proflavine Complexes. Biopolymers 1969; 8:45-55. [ Links ]
11. Molina A, Vaquero JJ, Garcia Navio JL, Alvarez-Builla J, de Pascual-Teresa B, Gago F, Rodrigo MM. Novel DNA Intercalators Based on the Pyridazino[1',6':1,2] pyrido[4,3-b]indol-5-inium System. J. Org. Chem. 1999; 64:3907-3915. [ Links ]
12. Molina A, Vaquero JJ, García-Navio JL, Alvarez-Builla J, Pascual-Teresa B, Gago F, Rodrigo MM, Ballesteros M. Synthesis and DNA binding properties of g-carbolinium derivates and benzologues. J. Org. Chem. 1996; 61:5587-5599. [ Links ]
13. Pastor J, Siro JG, Garcia Navio JL, Vaquero JJ, Alvarez-Builla J, Gago F, de Pascual-Teresa B, Pastor M, Rodrigo M. Azino Fused Benzimidazolium Salts as DNA Intercalating Agents 2. J. Org. Chem. 1997; 62:5476-5483. [ Links ]
14. Skoog DA, Leary JJ. Análisis instrumental. 4ª ed. Madrid: McGraw-Hill, 1994. [ Links ]
15. Scatchard G. The attractions of proteins for small molecules and ions. Ann. N. Y. Acad. Sci 1949; 51:660. [ Links ]
16. McGhee JD, von Hippel PH. Theoretical aspects of DNA-protein interactions: co-operative and non co-operative binding of large ligands to a one-dimensional homogeneous lattice. J. Mol. Biol. 1974; 86:469-489. [ Links ]
17. Crothers DM. Calculation of binding isotherms for heterogeneous polymers. Biopolymers 1968; 6:575-584. [ Links ]
18. Blackburn GM, Gait MJ. Nucleic Acids in Chemistry and Biology. New York: Oxford University Press, 1996. [ Links ]
19. Saenger W. Principles of Nucleic Acid Structure. New York: Springer-Verlag, 1984. [ Links ]
20. Job P. Formation and stability of inorganic complexes in solution. Ann. Chim. 1928; 9:113-203. [ Links ]
21. Likussar W, Boltz DF. Theory of continuous variation plots and a new method for spectrophotometric determination of extraction and formation constants. Ann. Chim. 1971; 43:1265-1272. [ Links ]
22. Cantor CR, Schimmel PR. The Behavior of Biological Macromolecules. Biophysical Chemistry, Part III:. San Francisco: W. H. Freeman, 1980; 1135-1139. [ Links ]
23. Porschke D. Electric Dichroism. In: Fox KR, ed. Methods in Molecular Biology: Drug-DNA Interaction Protocols. Totowa, New Jersey, 1997; 241-258. [ Links ]
24. Hamdan II, Skellern GG, Waigh RD. Use of capillary electrophoresis in the study of ligand-DNA interactions. Nucleic Acids Res. 1998; 26:3053-3058. [ Links ]
25. Wilson WD, Tanious FA, Fernandez-Saiz M, Rigl CT. Evaluation of Drug-Nucleic Acid Interactions by Thermal Melting Curves. In: Fox KR, ed. Methods in Molecular Biology: Drug-DNA Interaction Protocols. Totowa, New Jersey, 1997; 219-240. [ Links ]
26. Low CM, Drew HR, Waring MJ. Sequence-specific binding of echinomycin to DNA: evidence for conformational changes affecting flanking sequences. Nucleic Acids Res. 1984; 12:4865-4879. [ Links ]
27. Van Dyke MW, Hertzberg RP, Dervan PB. Map of distamycin, netropsin, and actinomycin binding sites on heterogeneous DNA:DNA cleavage-inhibition patterns with methidiumpropyl-EDTA.Fe(II). Proc. Natl. Acad. Sci. USA 1982; 79:5470-4. [ Links ]
28. Lane MJ, Dabrowiak JC, Vournakis JN. Sequence specifity of actinomycin D and netropsin binding to pBR322 analysed by protection from DNAase I. Proc. Natl. Acad. Sci. U.S.A. 1983; 80:3260-3264. [ Links ]
29. Scamrov AV, Beabealashvilli RS. Binding of actinomycin D to DNA revealed by DNAase I footprinting. FEBS Lett. 1983; 164:97-101. [ Links ]
30. Fox KR, Waring MJ. DNA structural variations produced by actinomycin and distamycin as revealed by DNAase I footprinting. Nucleic Acids Res. 1984; 12:9271-9285. [ Links ]
31. Fox KR, Howarth NR. Investigations into sequence-selective binding of mithramycin and releated ligands to DNA. Nucleic Acids Res. 1985; 13:8695-8714. [ Links ]
32. Low CM, Olsen RK, Waring MJ. Sequence preferences in the binding to DNA of triostin A and TANDEM as reported by DNAase I footprinting. FEBS Lett. 1984; 176:414-419. [ Links ]
33. Chaires JB, Fox KR, Herrera JE, Britt M, Waring MJ. Site and sequence specifity of the daunomycin-DNA interaction. Biochemistry 1987; 26:8227-8236. [ Links ]
34. Chaires JB, Herrera JE, Waring MJ. Preferential binding of daunomycin to 5'(A/T)CG and 5'(A/T)GC sequences revealed by footprinting titration experiments. Biochemistry 1990; 29:6145-6153. [ Links ]
35. Fox KR, Waring MJ. Nucleotide sequence binding preferences of nogalamycin investigated by DNAase I footprinting. Biochemistry 1986; 25:4249-4356. [ Links ]
36. Abu-Daya A, Brown PM, Fox KR. DNA sequence preferences of several AT-selective minor groove binding ligands. Nucleic Acids Res. 1995; 23:3385-3392. [ Links ]
37. Fenn JB, Mann M, Meng CK, Wong SF, Whitehouse CM. The ESI paper. Mass.Spectrom. Rev. 1990; 9:37-70. [ Links ]
38. Smith RD, Loo JA, Orgorzalek-Loo RR, Busman M, Udseth HR. Principles and Practice of Electrospray Ionization-Mass Spectrometry for Large Polypeptides and Proteins. Mass. Spectrom. Rev. 1991; 10:359-451. [ Links ]
39. Gale DC, Goodlett DR, Light-Wahl KJ, Smith RD. Observation of Duplex DNA-Drug Noncovalent Complexes by Electrospray Ionization Mass Spectrometry. J. Am. Chem. Soc. 1994; 116:6027-6028. [ Links ]
40. Berman HM, Westbrook J, Feng Z, Gilliland G, Bhat TN, Weissig H, Shindyalov IN, Bourne PE. The Protein Data Bank. Nucleic Acids Res. 2000; 28:235-242. [ Links ]
41. Peek ME, Williams LD. X-Ray Crystallography of DNA-Drug Complexes. Methods Enzimol. 2001; 340:282-290. [ Links ]
42. McPherson A. Preparation and Analysis of Protein Crystals. Malabar: Robert E. Krieger Publishing Co, 1989. [ Links ]
43. Bloomfield VA. DNA condensation by multivalent cations. Biopolymers 1997; 44:269-282. [ Links ]
44. Wang AH-J, Gao YG. Crystallization of oligonucleotides and their complexes with antitumor drugs. Methods, A Companion to Methods in Enzymology. New York: Academic Press, Inc, 1990; 91-99. [ Links ]
45. Shui X, Peek ME, Lipscomb LA, Gao Q, Ogata C, Roques BP, Garbay-Jaureguiberry C, Williams LD. Effects of Cationic Charge on Three-Dimensional Structure of Intercalative Complexes: Structure of a bis-Intercalated DNA Complex Solved by MAD Phasing. Curr. Med. Chem. 2000; 7:59-71. [ Links ]
46. Kielkopf CL, Erkkila KE, Hudson BP, Barton JK, Rees DC. Structure of photoactive rhodium complex intercalated into DNA. Nature Str. Biol. 2000; 7:117-121. [ Links ]
47. Lipscomb LA, Zhou FX, Presnell SR, Woo RJ, Peek ME, Plaskon RR, Williams LD. Structure of a DNA-Porphyrin Complex. Biochemistry 1996; 35:2818-2823. [ Links ]
48. Berman HM, Westbrook J, Feng Z, Gilliland G, Bhat TN, Weissig H, Shindyalov IN, Bourne PE. The Protein Data Bank. Nucleic Acids Res. 2000; 28:235-242. [ Links ]
49. Bailly C, Echepare S, Gago F, Waring MJ. Recognition elements that determine affinity and sequence-specific binding to DNA of 2QN, a biosynthetic bis-quinoline analogue of echinomycin. Anticancer Drug Des. 1999; 14:291-303. [ Links ]
50. Gago F. Stacking Interactions and Intercalative DNA Binding. Methods 1998; 14:277-292. [ Links ]
51. Gallego J, Luque FJ, Orozco M, Burgos C, Alvarez-Builla J, Rodrigo MM, Gago F. DNA sequence-specific reading by echinomycin: role of hydrogen bonding and stacking interactions. J. Med. Chem. 1994; 37:1602-1609. [ Links ]
52. Watson JD, Crick FH. A Structure for Deoxyribose Nucleic Acid. Nature 1953; 171:737-38. [ Links ]
53. Braña MF, Cacho M, García MA, de Pascual-Teresa B, Ramos A, Domínguez MT, Pozuelo JM, Abradelo C, Rey-Stolle MF, Yuste M, Báñez-Coronel M, Lacal JC. New analogues of amonafide and elinafide containing aromatic heterocycles: Synthesis, antitumor activity, molecular modelling and DNA binding properties. J. Med. Chem.; Aceptado. [ Links ]
54. Braña MF, Cacho M, García MA, de Pascual-Teresa B, Ramos A, Acero N, Llinares F, Muñoz-Mingarro D, Abradelo C, Rey-Stolle MF, Yuste M. Synthesis, Antitumor Activity, Molecular Modeling and DNA-binding Properties of a New Series of Imidazonaphthalimides. J. Med. Chem. 2002; 45:5813-5816. [ Links ]
55. de Pascual-Teresa B, Gallego J, Ortiz AR, Gago F. Molecular Dynamics Simulations of the Bis-Intercalated Complexes of Ditercalinium and Flexi-Di with the Hexanucleotide d(GCGCGC)2: Theoretical Analysis of the Interaction and Rationale for the Sequence Binding Specificity. J. Med. Chem. 1996; 39:4810-4824. [ Links ]
56. Gallego J, de Pascual-Teresa B, Ortiz AR, Pisabarro MT, Gago F. Molecular Electrostatic Potencials of DNA Base Pairs and Drug Chromophores in Relation to DNA Conformation and Bisintercalation by Quinoxaline Antibiotics and Ditercalinium. In: Sanz F, Giraldo J, Manaut F, eds. QSAR and Molecular Modelling: Concepts: Prous Science Publisher, 1995; 274-281. [ Links ]
57. Gallego J, Ortiz AR, de Pascual-Teresa B, Gago F. Structure-Affinity relationships for the binding of actinomycin D to DNA. J. Comput.-Aided Mol. Des. 1997; 11:114-128. [ Links ]
58. Leontis NB, SantaLucia JJ. Molecular Modeling of Nucleic Acids. Washington DC: American Chemical Society, 1998. [ Links ]
59. Fox KR. Methods in Molecular Biology: Drug-DNA Interaction Protocols. New Jersey: Totowa, 1997. [ Links ]
 Dirección para correspondencia
Dirección para correspondencia
Dra. B. de Pascual-Teresa
Departamento de Ciencias Químicas
Facultad de Ciencias Experimentales y de la Salud
Universidad San Pablo CEU
Urbanización Montepríncipe
Ctra. Boadilla del Monte, Km. 5,3
E-28668 Madrid
Recibido: 16.06.03
Aceptado: 10.10.03














