INTRODUCCIÓN
En la última década, los avances en la genómica y la tecnología de secuenciación de alto rendimiento han transformado nuestra comprensión de la base genética de las enfermedades complejas. Las puntuaciones de riesgo poligénico (PRS), conocidas como PRS por sus siglas en inglés (polygenic risk scores), han emergido como una herramienta innovadora que promete revolucionar la predicción del riesgo de enfermedades y la medicina personalizada. Estas puntuaciones se basan en la premisa fundamental de que muchas enfermedades complejas, incluyendo enfermedades cardiacas (1), diabetes (2), cáncer (3) o trastornos del tejido óseo (4), son el resultado de la interacción de múltiples variantes genéticas, cada una con un efecto pequeño aisladamente. En este contexto, las PRS permiten calcular el riesgo genético individual al incorporar información de cientos, e incluso miles, de variantes genéticas dispersas a lo largo del genoma (5).
La aplicación de las PRS en investigación médica y en la atención clínica ha generado un creciente interés debido a su potencial para identificar a las personas con mayor riesgo de desarrollar enfermedades, lo que, a su vez, puede orientar la prevención temprana y las estrategias de manejo personalizado. Sin embargo, su implementación en la práctica clínica tiene importantes implicaciones éticas, legales y sociales (ELSI) y plantea cuestiones de privacidad y desafíos en la interpretación de resultados, así como la necesidad de considerar el contexto clínico y otros factores de riesgo (6).
Esta revisión tiene como objetivo proporcionar una visión general no exhaustiva de las PRS, desde su fundamentación teórica hasta su aplicación clínica, y examinar las perspectivas futuras y desafíos en el campo. La comprensión de esta herramienta revolucionaria es fundamental en el contexto de la genómica de enfermedades y tiene el potencial de transformar la práctica médica, brindando nuevas oportunidades para el diagnóstico, la prevención y el tratamiento individualizado de enfermedades complejas.
GENERACIÓN DE PUNTUACIONES DE RIESGO POLIGÉNICO (PRS)
Una puntuación de riesgo poligénico (PRS) representa una estimación de la carga genética de un individuo relacionada con un rasgo o enfermedad específica. Su cálculo se basa en la suma de los alelos de riesgo del sujeto, los cuales son ajustados en función del tamaño del efecto de dichos alelos, tal como se deriva de los resultados de estudios de asociación de genoma completo (GWAS). La generación de las PRS involucra una serie de pasos metodológicos fundamentales. Un artículo publicado en Nature Protocols en 2020, describe detalladamente estos pasos, proporcionando un punto de partida y una guía de referencia para los investigadores interesados en realizar análisis de puntuación poligénica (7).
En primer lugar, se seleccionan las variantes genéticas relevantes a partir de los resultados de GWAS que han demostrado una asociación significativa con la enfermedad de interés. Luego, se ponderan estas variantes según su fuerza de asociación, asignando pesos que reflejan su contribución al riesgo genético. Posteriormente, el proceso de “clumping”, agrupa las variantes, teniendo en cuenta el desequilibrio de ligamiento (LD) entre ellos, de modo que los SNP retenidos sean en gran medida independientes entre sí. Adicionalmente, el proceso de “thresholding” implica la aplicación de un umbral para decidir qué variantes se incluyen en la PRS. Esto se hace considerando la fuerza de asociación de cada variante y, si su p-valor o estadístico de asociación supera el umbral especificado, se incluye en la PRS. La PRS se calcula sumando los productos de las variantes ponderadas por sus alelos en el genoma del individuo, y finalmente, se evalúa su capacidad de predicción utilizando datos de otros grupos independientes (7,8).
En los últimos años han surgido otros métodos para calcular las PRS, como son, el método LASSO (Least Absolute Shrinkage and Selection Operator, por sus siglas en inglés) (9), que utiliza regresión penalizada para seleccionar SNP informativos incorporando información de desequilibrio de ligamiento (LD) y los métodos de regresión bayesianos. Ambos han demostrado que pueden logran un mejor rendimiento que el método “clumping + threshold” (C+T) (10).
No obstante, existe una notoria variabilidad en los procedimientos de generación y validación de las PRS, lo que dificulta la comparación entre ellas y traslación de estas a la atención clínica. El grupo de trabajo de enfermedades complejas de ClinGen, en colaboración con el Catálogo de Puntuaciones Poligénicas (PGS), ha actualizado la “declaración de información de predicción de riesgo genético” (GRIPS, por sus siglas en inglés) para reflejar el estado actual del campo. En ese documento se define la información mínima necesaria para interpretar y evaluar las PRS, especialmente en relación con las aplicaciones clínicas posteriores. Además, se enfatiza la importancia de garantizar la disponibilidad y transparencia de datos, alentando a los investigadores a depositar y compartir las PRS a través del PGS, para facilitar la replicación en otros estudios (11).
APLICACIONES CLÍNICAS
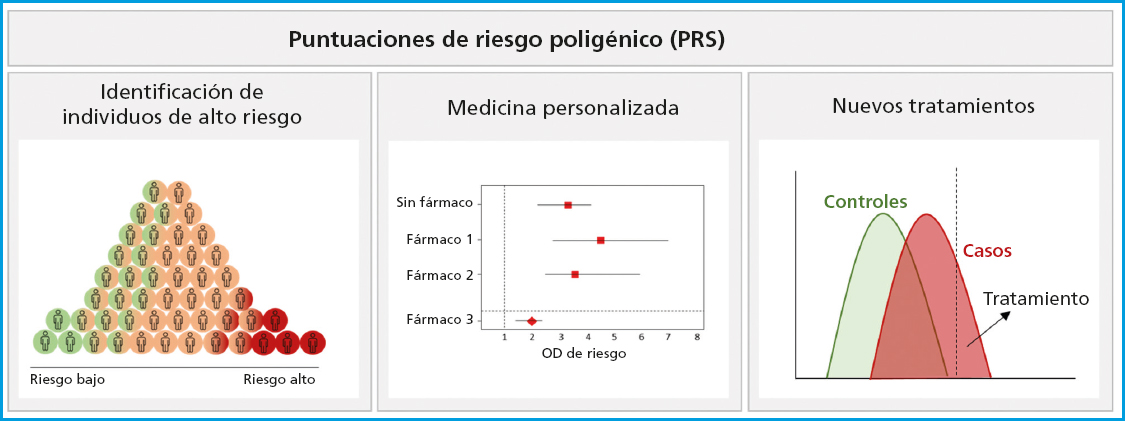
Las PRS han demostrado ser herramientas versátiles en una amplia gama de contextos y aplicaciones clínicas (Fig. 1). Una de las destacadas es la identificación de individuos de alto riesgo (11). Al calcular la PRS de un paciente, se pueden identificar aquellos que tienen un riesgo genético significativamente mayor de desarrollar una enfermedad en comparación con la población general. Esto permite una estratificación más precisa del riesgo y la posibilidad de intervenciones tempranas, como cambios en el estilo de vida, pruebas de detección o medidas de prevención farmacológica, que pueden reducir la incidencia y la gravedad de la enfermedad.
Los modelos habituales de riesgo no genético generalmente incorporan factores clínicos y de laboratorio, y pueden identificar individuos con alto riesgo que son adecuados para estrategias de prevención selectiva, o para la prescripción de medicamentos para reducir el riesgo de enfermedad. Sin embargo, estos factores no consiguen detectar una buena parte de las personas que finalmente desarrollarán la enfermedad. Por ejemplo, con las calculadoras de riesgo cardiovascular, casi el 40 % de las personas que finalmente padecerán enfermedades cardiacas no son detectadas, sobre todo si se trata de individuos jóvenes (12). Respecto al cáncer de mama, los múltiples factores que se utilizan ofrecen predicciones relativamente débiles, identificando solo una pequeña proporción de las personas con un alto riesgo a largo plazo (13).
En el campo de las enfermedades metabólicas óseas, el “Fracture Risk Assessment Tool” (FRAX) es una herramienta ampliamente utilizada para evaluar el riesgo de fracturas. Tras los primeros análisis y comprobaciones en mujeres españolas, sin análisis de densidad mineral ósea (DMO), demostró una capacidad de discriminación deficiente para predecir fracturas mayores, pero una buena capacidad de discriminación para fracturas de cadera según el área bajo la curva ROC. Sin embargo, su capacidad predictiva no es mucho mejor que la de modelos simples basados en la edad y la DMO (14). Más recientemente, usando las medias de corte de EE. UU., pudieron comprobar que FRAX tuvo un mejor desempeño en la identificación de pacientes que no sufrirán una fractura mayor osteoporótica o fractura de cadera en los próximos 10 años, que en aquellos que sí lo harán. Un número considerable de pacientes que desarrollaron fracturas mayores no fueron identificados en la evaluación inicial de FRAX (15). Es decir, según esos resultados, sería mayor su especificidad que su sensibilidad.
Algunos estudios han evaluado las PRS en paralelo con FRAX, como es el caso del PRS conocido como gSOS, que se relaciona con riesgo de fractura (16). Los autores demostraron que gSOS predecía la aparición de una fractura mayor osteoporótica o de una fractura de cadera mejor que la mayoría de los factores de riesgo clínicos tradicionales, incluyendo fracturas previas, uso de corticosteroides, artritis reumatoide y tabaquismo, aunque siempre por debajo del nivel de predicción de la DMO. También demostraron que agregar gSOS a FRAX mejoraba la predicción de riesgo de FRAX, aunque quedando también por debajo del nivel de predicción de FRAX combinado con DMO (16). En un estudio reciente, hemos analizado la capacidad de cuatro PRS diferentes para predecir la osteoporosis en la población española. Pudimos comprobar que el grupo de osteoporosis mostró un riesgo genético significativamente más alto en comparación con el grupo de control en 3 de estos PRS evaluados. Ello sugiere su potencial utilidad en estrategias de identificación de sujetos en riesgo basadas en la combinación de criterios clínico y genéticos (17).
Otra aplicación importante es la medicina personalizada. Las PRS pueden guiar la toma de decisiones médicas adaptadas a las características genéticas individuales de un paciente. Las PRS pueden ayudar a determinar el enfoque óptimo para el tratamiento, seleccionando terapias específicas que se ajusten al perfil genético del paciente y pronosticando la respuesta al tratamiento. En un estudio de terapias en cáncer de mama avanzado, los autores generaron un modelo final con 13 SNP (polimorfismos puntuales de un solo nucleótido), que, unidos a las covariables clínicas, mostró una capacidad predictiva con un área bajo la curva (AUC) integrada en el tiempo de 0,81, en comparación con un AUC de 0,64 para el modelo con solo las covariables clínicas (18). Este tipo de trabajo muestra la capacidad que tiene una PRS en el campo de la farmacogenómica, con el fin de orientar la elección de medicamentos y las dosis ideales para maximizar la eficacia y minimizar los efectos secundarios. Una revisión sistemática con el propósito de obtener información sobre el rendimiento de las PRS en la predicción de resultados terapéuticos identificó 89 artículos que incorporaron variantes farmacogenéticas en el desarrollo de modelos poligénicos. Los autores pudieron comprobar que casi todos los estudios encontraron una asociación significativa entre su modelo poligénico y el resultado del medicamento investigado (93 %). Sin embargo, menos de la mitad (47 %) compararon el rendimiento del modelo poligénico con predictores clínicos, y solo el 40 % validaron las predicciones del modelo en una cohorte independiente (19). Manousaki y colaboradores han explorado la aplicación del PRS-gSOS anteriormente descrito (16), como un factor de riesgo independiente para la aparición de fracturas en usuarios de medicamentos antiosteoporóticos. Mostraron que los pacientes con gSOS por debajo de la media tuvieron un aumento del 54 % en las probabilidades ajustadas de fractura mayor osteoporótica y un aumento del doble en las probabilidades ajustadas de fractura de cadera, en comparación con aquellos con gSOS por encima de la media. Demostraron así que el PRS-gSOS está asociado de manera independiente con fracturas incidentes entre los pacientes que reciben tratamiento para la osteoporosis (20).
Con todo esto, aunque hay muchos análisis de asociación entre riesgo poligénico y respuesta a fármacos en diversos campos como la anticoagulación, los trastornos neuropsiquiátricos, el cáncer o diversos trastornos metabólicos (21), aún hay muchas consideraciones clave para mejorar y facilitar la traslación a la clínica de estudios de este tipo.
Las PRS también se utilizan en la investigación de nuevos tratamientos y terapias. Al identificar a individuos con alto riesgo genético de una enfermedad, las PRS permiten seleccionar participantes para ensayos clínicos de prevención y tratamiento. Esto facilita la investigación de nuevas intervenciones terapéuticas, farmacológicas o no farmacológicas, con un enfoque específico en poblaciones de alto riesgo, acelerando el desarrollo de tratamientos más efectivos (22). Además, las PRS contribuyen a la comprensión de la base genética de estas enfermedades, aportando información sobre las vías biológicas subyacentes, lo que puede llevar a una mejor comprensión de su patogenia y, en última instancia, al desarrollo de estrategias de prevención y tratamiento más efectivas (21,23).
LIMITACIONES
A pesar de su potencial y utilidad en la genética de enfermedades y la medicina personalizada, las PRS presentan diversas limitaciones que deben ser consideradas en su aplicación y análisis. Una de las principales limitaciones es la precisión limitada en la predicción individual. Aunque las PRS pueden proporcionar una estimación del riesgo genético de una persona para una enfermedad, esta estimación es relativa y no garantiza la ocurrencia de la enfermedad. El riesgo genético es solo uno de los diferentes factores que influyen en la aparición de enfermedades complejas, y no tiene en cuenta factores ambientales, de estilo de vida u otros factores de riesgo que también desempeñan un papel crucial. Por tanto, no se pueden utilizar en exclusividad para predecir la ocurrencia de la enfermedad (24). Las herramientas de análisis de datos disponibles en la actualidad y el tamaño de las cohortes disponibles no permiten aún establecer un análisis que considere en profundidad las posibles interacciones entre los múltiples factores genéticos y ambientales.
Además, la precisión de las PRS puede variar según la población y la etnia. La mayoría de los estudios de asociación de genoma completo se han realizado en poblaciones de ascendencia europea, lo que puede limitar la aplicabilidad de las PRS en poblaciones más diversas. De hecho, varios estudios apuntan que las diferencias en la estructura genética de las diversas poblaciones pueden influir en la precisión de las PRS y requerir adaptaciones específicas para cada grupo poblacional (25,26).
Otra limitación importante se relaciona con la interpretación clínica. La información proporcionada por las PRS puede ser difícil de interpretar tanto para médicos como para pacientes. La toma de decisiones clínicas basadas en PRS requiere una comprensión sólida de la genética y la epidemiología, lo que puede no estar disponible en todos los entornos clínicos. Además, la comunicación de los resultados de PRS a los pacientes plantea desafíos éticos y de consejo genético (6,11).
CONSIDERACIONES ÉTICAS Y DE PRIVACIDAD
Las PRS plantean importantes cuestiones éticas y de privacidad. La información genética personal contenida en una PRS es sensible y debe manejarse con precaución. La protección de la privacidad del individuo y la seguridad de los datos genéticos son cuestiones críticas en la implementación de PRS en la atención médica (27).
Por otro lado, la aplicación de las PRS plantea la importancia de una correcta comunicación de los resultados a los pacientes. La información sobre el riesgo genético de una persona puede ser útil, pero también puede generar ansiedad y estrés, especialmente si la interpretación de estos resultados no es clara o si no se acompañan de recomendaciones efectivas de manejo. El consejo genético adecuado y la comunicación de los resultados de manera juiciosa, comprensible y sensible es esencial para garantizar que los pacientes comprendan el significado de sus PRS y puedan tomar decisiones informadas sobre su salud (11).
La privacidad de los datos genéticos es una preocupación crítica en el uso de PRS. La información genética es altamente sensible y puede revelar datos personales, como la ascendencia y las características genéticas individuales. La protección de la privacidad de los datos genéticos es esencial para evitar el uso indebido de esta información, incluyendo la discriminación genética en ámbitos como el empleo o los seguros de enfermedad (28). La seguridad de los datos genéticos es otra preocupación. La información genética debe ser guardada de manera segura para prevenir la exposición no autorizada o el robo de datos. La implementación de prácticas de seguridad sólidas y la encriptación de datos son esenciales para proteger la privacidad de los individuos y evitar vulnerabilidades en la gestión de datos genéticos (29).
Por último, la equidad en el acceso a la información genética es otra cuestión ética relevante. La disponibilidad de PRS puede estar sesgada hacia aquellos que tienen acceso a servicios de secuenciación genética, dejando a algunos grupos de la población en. Garantizar un acceso equitativo y justo a las PRS es un desafío ético importante (30).
PERSPECTIVAS FUTURAS
El campo de las PRS continúa evolucionando y promete desempeñar un papel cada vez más relevante en la genética clínica y la medicina de precisión. Las perspectivas futuras en el campo de las PRS se centran en una mejora de los riesgos y limitaciones que se han comentado anteriormente. Se requiere una mayor precisión predictiva, que mejore a medida que se acumule más evidencia y se desarrollen modelos más sofisticados. La incorporación de datos de secuenciación genómica completa y la identificación de variantes más raras y de bajo efecto podrían aumentar significativamente la capacidad de predicción de las PRS. La adaptación a poblaciones diversas con la inclusión de datos de diferentes grupos étnicos y poblaciones permitirá una aplicación más amplia de las PRS en un contexto global y mejorará su precisión en poblaciones no europeas.
A corto plazo y como ya se puede comprobar en diversas publicaciones de distintos campos de la medicina, incluyendo las enfermedades esqueléticas, existen PRS prometedoras y es verosímil que aparezcan nuevas PRS con mayor sensibilidad y especificidad. Estas PRS, en combinación con otros factores de riesgo (clínicos, analíticos o de imagen), podrían mejorar la estratificación de pacientes en riesgo de fractura ósea y diseñar estrategias preventivas personalizadas. Algo así se aplica ya en algunos cánceres, donde se utilizan estrategias que combinan paneles de genes y factores clínicos para determinar el riesgo individual (31,32).
A medida que las PRS se generalicen, habrá discusiones éticas y legales más intensas sobre temas como la privacidad, la discriminación genética y la equidad en el acceso. Será fundamental abordar estas cuestiones de manera efectiva y promover políticas que protejan los derechos de los individuos.











 texto en
texto en