ESTUDIOS DE ASOCIACIÓN DE GENOMA COMPLETO (GWAS)
En los últimos años los estudios de asociación de genoma completo (GWAS) han sido una herramienta esencial para la identificación de genes involucrados en enfermedades complejas (1). Estos estudios consisten en establecer una asociación entre la frecuencia génica o alélica de millones de marcadores tipo SNP (polimorfismos de un único nucleótido; del inglés single nucleotide polymorphism) distribuidos a lo largo del genoma y un fenotipo o enfermedad de estudio (2). Esta aproximación representa la herramienta más completa e imparcial para el estudio de enfermedades complejas. A diferencia de los estudios de asociación de genes candidatos, los estudios GWAS son una aproximación sin hipótesis previa, permitiendo el descubrimiento de nuevos genes o vías de señalización implicadas en el fenotipo de estudio que hasta el momento eran totalmente desconocidas (3). El establecimiento de los GWAS ha sido posible gracias a los nuevos avances en tecnología genómica de alto rendimiento, diseño de estudio, análisis estadísticos mejorados y la posibilidad de disponer de grandes biobancos (4,5). Debido al gran número de pruebas estadísticas simultáneas realizadas y, por lo tanto, a las correcciones estadísticas (que requieren un umbral del valor p de 5x10-8 para considerarse significativo a nivel de genoma completo y al pequeño efecto que presenta cada variante en las enfermedades complejas, son necesarias cohortes muy grandes. Esto se ha conseguido mediante los metaanálisis de GWAS, donde diferentes estudios se han unido con el objetivo de incrementar el tamaño muestral (6,7).
Aun con el evidente éxito, los estudios GWAS presentan 3 limitaciones cruciales. Primero, las variantes genéticas utilizadas para validar la asociación con el fenotipo concreto son SNP marcadores (en inglés, tagSNPs) que se distribuyen homogéneamente por todo el genoma con una frecuencia del alelo minoritario (MAF) mayor o igual al 5 % en la población. Por lo tanto, las variantes raras con posibles efectos fuertes en el fenotipo no están incluidas en estos estudios. Esta limitación se ha intentado solucionar incluyendo variantes de menor frecuencia en los chips de genotipado, secuenciando el exoma o genoma completo (whole exome/genome sequencing, WES/WGS) y/o utilizando los extremos fenotípicos de las cohortes. Segundo, el éxito de los estudios GWAS depende en gran manera del tamaño de la muestra. Por esta razón, como se ha comentado, la estrategia más común en la actualidad es establecer grandes consorcios que incluyen diferentes cohortes de todo el mundo. Así, se obtienen supercohortes con mayor poder estadístico, pero genéticamente heterogéneas, de manera que es muy difícil el descubrimiento de variantes específicas de población. Tercero, los GWAS reportan el SNP más relevante estadísticamente, llamado SNP centinela. Aunque este SNP puede ser el SNP causal de la asociación, también lo pueden ser otras variantes que se encuentren en desequilibrio de ligamiento con la variante centinela. Si el SNP asociado se encuentra en una región codificante e implica un cambio de aminoácido, hay una alta probabilidad de que el SNP sea causal, pero la realidad es que la mayoría de los SNP centinela se encuentran en regiones no codificantes (96 %), tanto intrónicas (41 %), como intergénicas (54 %), dificultando por tanto la demostración de su papel causal. Debido a su naturaleza no-codificante, realizar estudios funcionales de estos SNP centinelas es un verdadero reto (8-10). Es por esta razón que estos estudios funcionales siguen siendo pocos hasta el momento, y establecer las bases funcionales de las asociaciones encontradas en dichos análisis es la asignatura pendiente de esta era post-GWAS.
Para realizar los estudios de funcionalidad, es necesaria una aproximación interdisciplinar, que va desde los análisis in silico (consistentes en aproximaciones computacionales) (11,12), como pueden ser las herramientas predictoras de patogenicidad; pasando por los estudios in vitro, que incluyen entre otros, los ensayos de gen reportero (p. ej. luciferasa) (13); hasta los estudios in vivo usando modelos animales como el pez cebra o el ratón (14,15).
En esta revisión se presenta de forma sumaria los principales trabajos GWAS que se han reportado utilizando fenotipos óseos y a continuación algunos ejemplos de estudios in vitro e in vivo que se han generado a raíz del primer gran metaanálisis de GWAS (16) que se realizó sobre densidad mineral ósea (DMO) y riesgo de fractura.
GWAS Y HUESO
Para realizar GWAS de enfermedades óseas y principalmente de la osteoporosis, se pueden tener en cuenta parámetros como la DMO, la geometría y la microarquitectura del hueso. De estas propiedades, la más utilizada y la que mejor predice la fractura osteoporótica es la DMO, que es un rasgo cuantitativo que se mide en una escala continua por métodos como la absorciometría dual de rayos X (DXA por sus siglas en inglés). Se estima que la DMO es un rasgo con una heredabilidad aproximada de 50-80 %. Asimismo, la geometría del hueso muestra una heredabilidad de entre 30-70 %, y la microarquitectura ósea determinada por tomografía computarizada de alta resolución (HR-pQCT) una heredabilidad entre 20-80 % (17).
Hasta el momento, se han realizado más de 50 grandes estudios GWAS utilizando parámetros óseos, junto con una plétora de estudios de asociación en cohortes más pequeñas y homogéneas. Con esto, se han identificado más de 500 loci asociados. Aunque el porcentaje de varianza explicado a través de los GWAS ha incrementado sustancialmente en los últimos años al usar cohortes de mayor tamaño, todos estos loci solamente explican una pequeña proporción (20 %) de la contribución genética a la DMO (18,19). Esto ha llevado a que exista una diferencia entre la variabilidad explicada por factores genéticos y la heredabilidad de la DMO, debido, posiblemente, a una sobreestimación de la heredabilidad o a que no se están considerando otros factores genéticos como son las variantes de número de copias (en inglés copy number variants, CNV) o la epigenética (20).
Con todo, estos estudios GWAS han dado lugar a hallazgos importantes, como la confirmación de las asociaciones de genes como SOST y LRP5 (que ya habían estado implicados en enfermedades monogénicas esqueléticas) con algunos fenotipos óseos, o la identificación de nuevos genes de los que no se conocía su implicación en hueso (21). En la tabla I se presentan algunos de los estudios GWAS más relevantes relacionados con la DMO, la mayoría de los cuales están reportados en el GWAS catalog (https://www.ebi.ac.uk/gwas/). Para acotar, solo se han considerado estudios de cohortes de más de 10 000 individuos.
Tabla I (cont.). Estudios GWAS sobre hueso y genes encontrados con variantes asociadas a fenotipos óseos.

Tabla I (cont.) Estudios GWAS sobre hueso y genes encontrados con variantes asociadas a fenotipos óseos.

El estudio está representado por el primer autor y el año. Los genes son aquellos más relevantes en el estudio por su asociación con fenotipos óseos y por la novedad de su hallazgo. A: antebrazo. AE: ángulo del eje. AEC: ancho del estrecho del cuello. ARX-MC: absorciometría de rayos X-modos derivados de la forma de la cadera. AUBA: atenuación de ultrasonido de banda ancha. C: cadera. CF: cuello femoral. CL: columna lumbar. CL-AH: columna lumbar-área del hueso. CT: cadera total. CuT: cuerpo total. DMO: densidad mineral ósea. DMOe: densidad mineral ósea estimada. FO: fractura osteoporótica. FVC: fractura vertebral clínica. FVR: fractura vertebral radiográfica. LCF: longitud de cuello femoral. MBSM: medida bilateral semicuantitativa de mano. MMCBP: masa magra corporal de brazos y piernas. MMT: masa magra del tronco. OA: osteoartritis. SMCF: sección modular del cuello femoral. T: talón. TO: tamaño óseo. UT: ultrasonido de talón. VS: velocidad de sonido.
Muchos de los estudios GWAS presentados en la tabla I corresponden a trabajos en los que se han realizado metaanálisis grandes que han dejado como resultado cientos de variantes en diferentes loci asociadas con fenotipos óseos. Sin embargo, la mayoría de estos estudios carecen de aproximaciones funcionales.
ESTUDIOS FUNCIONALES DE LA ERA POST-GWAS
A pesar de la gran cantidad de estudios de asociación realizados hasta ahora, los estudios funcionales no han tenido el mismo desarrollo. De los 1051 artículos que han citado al primer gran metaanálisis de GWAS sobre hueso (16), solo una pequeña fracción incluye estudios funcionales (164, es decir, 15 %), ya sean in vitro o in vivo.
Un ejemplo de éxito en los estudios funcionales es la caracterización de la regulación de SOST. Este gen codifica la proteína esclerostina, un inhibidor de la vía canónica de WNT (49-51), que se ha encontrado asociado a multitud de parámetros óseos en diferentes estudios de asociación en distintas poblaciones (17,28,33,38,40,43,52,53) (Fig. 1A). Su función inhibidora sobre la formación ósea ha sido ampliamente estudiada mediante modelos in vivo e in vitro, y en la actualidad se utilizan anticuerpos antiesclerostina como terapia en enfermedades óseas, como son la osteoporosis o la osteogénesis imperfecta (54-59). Es por esto, que los factores reguladores de la expresión del gen se incluyen entre los nuevos candidatos como diana para el desarrollo de nuevas terapias. En humanos, variantes en el gen SOST han sido asociadas a condiciones caracterizadas por un exceso de formación ósea: esclerosteosis, displasia craneodiafisaria, y el fenotipo de alta masa ósea (60) (Fig. 1B). Junto con estas, también se encuentra la enfermedad de van Buchem causada por la deleción de la región reguladora denominada ECR5 situada a 52 kb cascada abajo del gen, que es necesaria para la correcta expresión de SOST (61) (Fig. 1A). En realidad, la transcripción de SOST está finamente regulada por un gran número de señales, tanto a través de la regulación directa sobre el promotor de SOST como a través de la región reguladora distal ECR5 (62,63), cuya interacción física se ha demostrado en un estudio reciente de nuestro grupo realizado en células óseas (64) (Fig. 1A). El factor de transcripción MEF2C es el regulador de SOST mejor descrito en relación con su expresión en osteocitos (63,65). La importancia de MEF2C en el efecto potenciador de ECR5 ha sido confirmada con el modelo de ratón KO (knockout) de Mef2c en osteoblastos/osteocitos, que presenta alta masa ósea y bajos niveles de esclerostina (66). Precisamente, MEF2C es otra de las señales más repetidas en los estudios GWAS con parámetros óseos (16,23,36,37,67-70). Junto con MEF2C, también se ha descrito HDAC5, un regulador negativo de la expresión de SOST que ejerce su función bloqueando la unión de MEF2C a ECR5 durante la diferenciación de los osteocitos inmaduros (Fig. 1C). En consonancia con esto, el modelo de ratón KO de HDAC4/5 muestra una baja DMO y una alta expresión de SOST (71-73). De nuevo HDAC4/5 se encuentra dentro de los loci más repetidos en los estudios de asociación con parámetros óseos (18,23,34,39,74) (Fig. 1B).
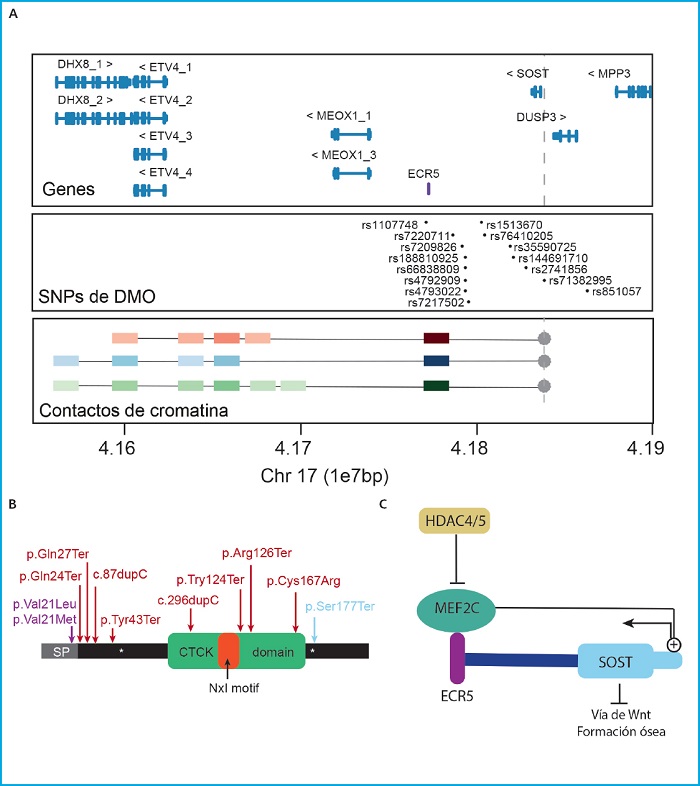
Figura 1. SOST. A. Panel superior: Locus que contiene SOST y sus genes vecinos (GRC37/hg19). En lila, la región reguladora ECR5. Panel central: SNP asociados con diferentes parámetros óseos en diferentes estudios GWAS, extraídos de GWAS catalog (https://www.ebi.ac.uk/gwas7). Panel inferior: Resultados principales del ensayo 4C de Martínez-Gil et al. 2021, donde se representan las principales interacciones entre el promotor de SOST (usado como anzuelo e indicado con un punto y una línea discontinua gris). Los cuadrados de colores representan las interacciones con la intensidad de color proporcional a la intensidad de la interacción. Los cuadrados rojos representan las interacciones en células madre mesenquimales, en azul las interacciones de las células hFOB y en verde las interacciones de las células Saos2. Las unidades de la escala genómica utilizada (1e7pb) corresponden a 10 Megabases (1x107 pares de bases). B. Representación esquemática de la proteína esclerostina señalando sus dominios funcionales y las variantes descritas causantes de enfermedades esqueléticas humanas. En lila las variantes asociadas a displasia craneodiafisaria, en rojo asociadas a esclerosteosis y en azul la variante asociada al fenotipo de alta masa ósea.CTCK:C/terminal cysteine knot-like. C. Esquema de algunos de los reguladores positivos y negativos de la expresión de SOST.
Otro ejemplo de la importancia de realizar estudios funcionales de las regiones asociadas es el locus de DKK1. DKK1 es otro inhibidor de la vía canónica de WNT, con un papel muy importante en la morfogénesis de la cabeza (75,76), y en el desarrollo del hueso (77,78). En la actualidad no hay ninguna variante en DKK1 descrita causante de ninguna patología ósea en la base de datos HGMD. A pesar de esto, nuestro grupo ha identificado dos variantes de cambio de aminoácido (missense) en pacientes con el fenotipo de DMO alta, que muestran una pérdida de función de su capacidad inhibitoria (13,79). De manera contraria, una de estas variantes también se ha encontrado en pacientes con fenotipos totalmente opuestos como la osteoporosis o malformaciones anales (80,81). Además, es interesante resaltar que no se han encontrado SNP en DKK1 asociados con la DMO o con diferentes parámetros óseos entre los diferentes GWAS. En cambio, sí se ha demostrado asociación con la DMO de un conjunto de SNP agrupados en una región a 350 kb cascada abajo de DKK1 y a 92 kb cascada arriba de MBL2 (16,18,19,29,33,34,36,37,39,74) (Fig. 2). Para tratar de distinguir cuál de estos dos genes era el responsable de esta asociación, en un trabajo de nuestro grupo (13), se realizó un ensayo de conformación de la cromatina 4C utilizando la región rica en señales GWAS como anzuelo en tres tipos celulares óseos. Con esto se pudo confirmar la interacción física entre esta región y el promotor de DKK1 y descartar la interacción con el gen MBL2 (Fig. 2, panel inferior). Precisamente, en esta región se encuentra el gen LNCAROD que especifica un ARN largo no codificante (en inglés, long noncoding RNA, lncRNA) activador de DKK1 que, por lo tanto, es el posible responsable de la asociación de los estudios GWAS (82).
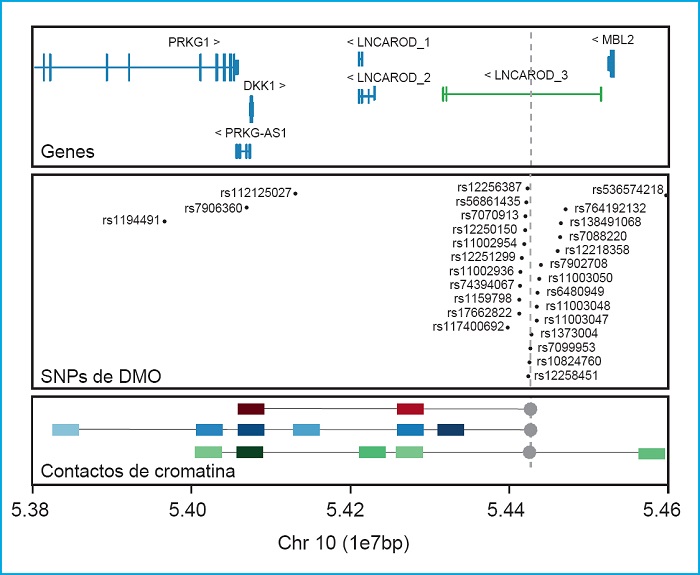
Figura 2. DKK1. Panel superior: locus que contiene DKK1 y sus genes vecinos (GRC37/hg19). En verde, el lncRNA LNCAROD de GENCODE v32.2 (GRC38/hg18). Panel central: SNP asociados con diferentes parámetros óseos en diferentes estudios GWAS, extraídos del GWAS catalog (https://www.ebi.ac.uk/gwas7). Panel inferior: Resultados principales del ensayo 4C de Martínez-Gil et al. 2020, donde se representan las principales interacciones con la región rica en SNP asociados a DMO (usado como anzuelo e indicado con un punto y una línea discontinua gris). Los cuadrados de colores representan las interacciones con la intensidad de color proporcional a la intensidad de la interacción. Los cuadrados rojos representan las interacciones en células madre mesenquimales, en azul las interacciones de las células hFOB y en verde las interacciones de las células Saos2. Las unidades de la escala genómica utilizada (1e7pb) corresponden a 10 megabases (1x107 pares de bases).
Uno de los loci más consistentes entre distintos GWAS sobre la DMO es la región genómica situada en 7q31.31 que incluye el gen WNT16. Se trata de un locus muy complejo que incluye, además de WNT16, los genes vecinos ING3, FAM3C y CPED1. El papel de WNT16 sobre la determinación de la DMO ha sido claramente establecido en estudios funcionales de modelos de ratón KO o KO condicional específico de osteoblasto (6,83,84), que a grandes rasgos coinciden en mostrar fracturas espontáneas debidas a una baja DMO, junto con un grosor cortical y la resistencia ósea reducidos.
Sin embargo, también se han encontrado pruebas sobre la importancia de los otros tres genes vecinos en el metabolismo óseo. En el caso de ING3 (Inhibitor Of Growth Family Member 3), que forma parte del complejo NuA4 histona acetiltransferasa (HAT) implicado en funciones de regulación de la cromatina, se ha determinado que se expresa abundantemente en el tejido óseo (85) y los estudios funcionales en un modelo celular in vitro de células mesenquimales KO para ING3 muestran afectación de la osteoblastogénesis y una estimulación de la diferenciación adipogénica (86).
Para CPED1 (Cadherin Like And PC-Esterase Domain Containing 1), no se conoce la función concreta ni en humanos ni en ratón. Sin embargo, en ratón, los estudios funcionales muestran que Cped1 se encuentra uniformemente expresado en una variedad de tejidos, incluido el óseo y se han descrito diferentes isoformas debidas a empalmes (splicing) alternativos, así como tres regiones promotoras activas durante la diferenciación osteogénica (87). Para acabar de definir su posible rol en la homeóstasis ósea, faltarían estudios funcionales adicionales, en modelos celulares in vitro o en modelos animales. FAM3C (Family of sequence similarity 3c) es un factor de crecimiento tipo citocina expresado en multitud de tejidos (88), que juega un papel muy importante en la transición epitelio-mesénquima y en la metástasis del cáncer (89). Su relación con el metabolismo óseo se ha confirmado con el modelo de ratón KO que presenta alteraciones en la estructura ósea (88).
Se han realizado algunos estudios funcionales que abordan la regulación de la expresión de los distintos genes de esta región. Por ejemplo, nuestro grupo ha llevado a cabo estudios de eQTL (del inglés expresssion Quantitative Trait Locus) en osteoblastos primarios que demuestran que SNP situados dentro del gen WNT16 son reguladores de los niveles de expresión de FAM3C en esas células (90). También hemos visto, en células del linaje osteoblástico, que se da una interacción física entre varios potenciadores génicos (enhancers) situados dentro del gen CPED1 y el promotor de WNT16 (91). Todo ello ilustra una compleja relación entre estos cuatro genes y sugiere la posibilidad de que se dé una sinergia entre ellos. Con todo, queda clara la necesidad de estudios funcionales adicionales que acaben de definir el papel de cada uno de ellos y sus interacciones.
Los trabajos aquí citados evidencian la importancia que tienen los estudios funcionales a partir de los descubrimientos que traen consigo los análisis de GWAS y ponen en el horizonte el reto, en la era post-GWAS, de continuar encontrando correlaciones entre las variantes que se derivan de los estudios de GWAS y los aspectos funcionales de esas variantes, bien sea in silico, in vitro o in vivo, buscando con ello nuevas aproximaciones que puedan dar luces y opciones terapéuticas para las patologías asociadas.











 text in
text in 



